Идентификация генетических маркеров генов APOM и CYP7A1, влияющих на показатели молочной продуктивности у китайских голштинов
Наше предыдущее исследование определило гены аполипопротеина М (APOM) и полипептида 1 подсемейства А цитохрома P450 семейства 7 (CYP7A1) как кандидатов на молочные признаки у молочного скота, которые были значительно повышены в ткани печени коров голштинской породы между периодами сухостоя и лактации. Два гена играют критическую роль в пути рецептора, активируемого пролифераторами пероксисом (PPAR). В этом исследовании мы дополнительно подтвердили, имели ли гены APOM и CYP7A1 значительное генетическое влияние на признаки молочной продуктивности в популяции китайских голштинов.
Аннотация
С помощью двухнаправленного секвенирования продуктов полимеразной цепной реакции (ПЦР) полных кодирующих последовательностей и 2000 п.н. 5′ и 3′ фланкирующих областей в объединенном образце ДНК были идентифицированы семь и три однонуклеотидных полиморфизма (SNP) вAPOMиCYP7A1, соответственно. В SAS 9.2 анализ ассоциаций фенотипа и генотипа показал, что такие однонуклеотидные полиморфизмы (SNP) достоверно ассоциированы как минимум с одним признаком молочной продуктивности, включая 305-дневный удой, выход молочного жира, процентное содержание молочного жира, выход молочного белка и процентное содержание молочного белка за первую и вторую лактации (p= <0,01–0,04). В Haploview 4.2 мы также обнаружили, что шесть однонуклеотидных полиморфизмов (SNP) в генеAPOMи три однонуклеотидных полиморфизма (SNP) в генеCYP7A1образуют один гаплотип соответственно. Гаплотипы также достоверно ассоциированы как минимум с одним признаком молочной продуктивности (p= <0,01–0,02). Следует отметить, что мы обнаружили, что однонуклеотидные полиморфизмы (SNP) в 5′ регуляторной области, rs209293266 и rs110721287 вAPOMи rs42765359 вCYP7A1, значительно повлияли на транскрипционную активность гена после мутации (p< 0,01) посредством изменения участков связывания факторов транскрипции с помощью экспериментов по анализу люциферазы. Кроме того, с помощью веб-сервера RNAfold, rs110098953 и rs378530166 изменили вторичную структуру мРНК геновAPOMиCYP7A1, соответственно. Подводя итог, можно сказать, что наше исследование впервые продемонстрировало, что геныAPOMиCYP7A1оказывают значительное генетическое влияние на показатели надоев и состава молока, а идентифицированные SNP могут служить доступными генетическими маркерами для программы геномной селекции у молочного скота.
1. Введение
Поскольку уровень потребления молока постепенно увеличивается, крайне важно улучшать состав молока и его пищевую ценность. С 2009 года исследователи используют технологию геномной селекции (GS) для достижения ранней селекционной селекции и улучшения производственных показателей молочных коров [ 1 ]. Исследования показали, что точность геномных оценочных племенных ценностей (GEBV) может быть улучшена, если данные микрочиповых маркеров, используемые GS, содержат информацию о полиморфизмах одиночных нуклеотидов (SNP), которые существенно влияют на целевые признаки [ 2 ]. Наши начальные исследования РНК-секвенирования (RNA-Seq) показали, что уровни экспрессии генов аполипопротеина М ( APOM ) и цитохрома P450 семейства 7 подсемейства А полипептида 1 ( CYP7A1 ) в печени в ранний период лактации были значительно выше, чем в сухостойный период у коров голштинской породы [ 3 ]. Ген APOM находится вблизи известных локусов количественных признаков (QTL) для выхода молочного жира (1,10 Мб), выхода молочного белка (0,10 Мб) и процентного содержания молочного белка (0,10 Мб) у молочного скота; ген CYP7A1 находится на расстоянии 0,50 Мб и 1,90 Мб от областей QTL для процента молочного жира и выхода жира соответственно ( https://www.animalgenome.org/cgi-bin/QTLdb/index , дата обращения: 12 мая 2024 г.). Таким образом, гены APOM и CYP7A1 были выведены в качестве критически важных кандидатов для определения признаков, связанных с молоком у молочного скота.
Ген APOM у крупного рогатого скота расположен на 23-й хромосоме, имеет 5 экзонов длиной 2106 п.н. и участвует в различных аспектах липидного обмена, включая связывание фосфолипидов, ассоциацию с частицами липопротеинов высокой плотности (ЛПВП) и облегчение оттока холестерина [ 4 , 5 ]. APOM преимущественно находится в ЛПВП, липопротеиновых частицах, богатых холестерином и фосфолипидами, которые играют ключевую роль в транспортировке холестерина и других биоактивных молекул из периферических тканей в печень [ 6 , 7 ]. Ген CYP7A1 расположен на BTA14 и имеет 6 экзонов общей длиной 10 337 п.н. Он принадлежит к семейству ферментов системы цитохрома P450 и участвует в регуляции активности холестерин-7-альфа-монооксигеназы, биосинтезе желчных кислот, рецепторов, активируемых пролифераторами пероксисом (PPAR), и сигнальных путях метаболизма холестерина [ 8 , 9 ]. CYP7A1 играет ключевую роль в качестве фермента, ограничивающего скорость расщепления холестерина и синтеза желчных кислот (ЖК) как класса метаболитов холестерина [ 10 , 11, 12 ].
До сих пор неясно, оказывают ли эти два гена заметное генетическое влияние на показатели состава молока у молочного скота. Целью данного исследования было выявление всех потенциальных однонуклеотидных полиморфизмов (SNP) в генах APOM и CYP7A1 и оценка их генетического влияния на продуктивность и показатели состава молока у популяции китайской голштинской породы, что позволило бы получить ценные генетические маркеры для программ геномной селекции молочного скота.
2. Материалы и методы
2.1. Животные и фенотипические данные
В данном исследовании были получены образцы крови от 1123 китайских коров голштинской породы от 80 семей быков-производителей с молочных ферм Циншаньцзе и Шуанфэн (Баодин, провинция Хэбэй, Китай). Рабочая станция животноводства и мелкопородного разведения провинции Хэбэй предоставила полную информацию о родословной и стандартные данные по улучшению молочного стада (DHI), включая пять показателей молочной продуктивности: удой за 305 дней, выход молочного жира, процентное содержание молочного жира, выход молочного белка и процентное содержание молочного белка. Мы суммировали фенотипические значения показателей молочной продуктивности за первую и вторую лактации в таблице S1 .
2.2. Идентификация SNP и генотипирование
Геномная ДНК была выделена из образцов цельной крови 1123 коров с помощью набора TIANamp Blood DNA Kit (Tiangen, Пекин, Китай). Были определены концентрация и качество экстракции образцов ДНК, а также длина образцов ДНК была протестирована методом электрофореза в 1,5% агарозном геле. Затем мы случайным образом выбрали 111 образцов ДНК и разбавили концентрацию ДНК в каждом образце до 50 нг/мкл. Затем мы извлекли по 5 мкл из каждого образца и распределили их по пяти пулам ДНК.
На основе геномных последовательностей генов APOM и CYP7A1 (идентификаторы GenBank: NC_037350.1 и NC_037341.1) мы последовательно сконструировали 10 и 16 пар праймеров для расширения полных кодирующих последовательностей и 2000 п.н. 5′- и 3′-фланкирующих областей, используя Primer3 версии 4.1.0 ( https://bioinfo.ut.ee/primer3/ , дата обращения: 20 мая 2024 г.; Таблица S2 ). Система и условия реакции ПЦР описаны в инструкции к ДНК-полимеразе PrimeSTAR Max (Takara, Далянь, Китай). Продукты ПЦР были подвергнуты двунаправленному секвенированию. С помощью программы BLAST ( https://blast.ncbi.nlm.nih.gov/Blast.cgi , дата обращения: 10 июня 2024 г.) мы сопоставили последовательности продуктов ПЦР с референтными последовательностями крупного рогатого скота (ARS-UCD1.2) для выявления потенциальных однонуклеотидных полиморфизмов (SNP). Затем 1123 коровы голштинской породы были генотипированы по выявленным SNP с помощью масс-спектрометрии с времяпролетной лазерной десорбцией и ионизацией на матричном носителе (MALDI-TOF MS).
2.3. Оценка неравновесия по сцеплению (LD)
Мы использовали программу Haploview 4.2 (Институт Брода, Кембридж, Массачусетс, США) для оценки степени неравновесного сцепления (LD) между семью однонуклеотидными полиморфизмами (SNP) в гене APOM и тремя однонуклеотидными полиморфизмами в гене CYP7A1, обнаруженными в данном исследовании, соответственно. Степень неравновесного сцепления определялась по значению D', которое измерялось как индикатор силы LD. Гаплотипы с частотой встречаемости более 0,05 рассматривались для дальнейшего анализа.
2.4. Анализ ассоциаций по признакам молочной продуктивности
Мы проанализировали ассоциации генотипов/гаплотипов с пятью признаками молочной продуктивности за первую и вторую лактации, используя модуль MIXED в программном обеспечении SAS 9.2 (SAS Institute Inc., Кэри, Северная Каролина, США). Для анализа ассоциаций использовалась следующая модель животных:
где y — фенотипическое значение признака; µ — среднее значение популяции; hys — комбинация фермы (2 уровня), года отела (7 уровней) и сезона отела (4 уровня); M (ковариата) — месяц при отеле, где b — его коэффициент регрессии; G — эффект генотипа или гаплотипа; a – индивидуальный случайный аддитивный генетический эффект; e – случайный остаток. Многочисленные тесты для разных генотипов одного и того же SNP подвергались коррекции Бонферрони, которая корректирует уровень значимости путем деления исходного p -значения на количество генотипов или комбинаций гаплотипов. Мы также рассчитали аддитивный (a), доминантный (d) и субституционный (α) эффекты с помощью программы SAS 9.2.
2.5 Прогнозирование биологических функций
Для оценки влияния (относительная оценка ≥ 0,90) однонуклеотидных полиморфизмов (SNP) в 5'-фланкирующих областях генов APOM и CYP7A1 на сайты связывания факторов транскрипции (TFBS) использовалось программное обеспечение Jaspar 2024 ( http://jaspar.genereg.net/ , дата обращения 25 июня 2024 г.) . Изменения во вторичной структуре мРНК, затрагивающие SNP в нетранслируемых областях (UTR), и экзоны были спроецированы с помощью веб-сервера RNAfold (http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi , дата обращения 25 июня 2024 г.), в котором стабильность структуры мРНК определяется минимальной свободной энергией (MFE) оптимальной вторичной структуры и обратно пропорциональна стабильности структуры мРНК.
2.6. Создание рекомбинантной плазмиды, культивирование клеток и анализ люциферазы
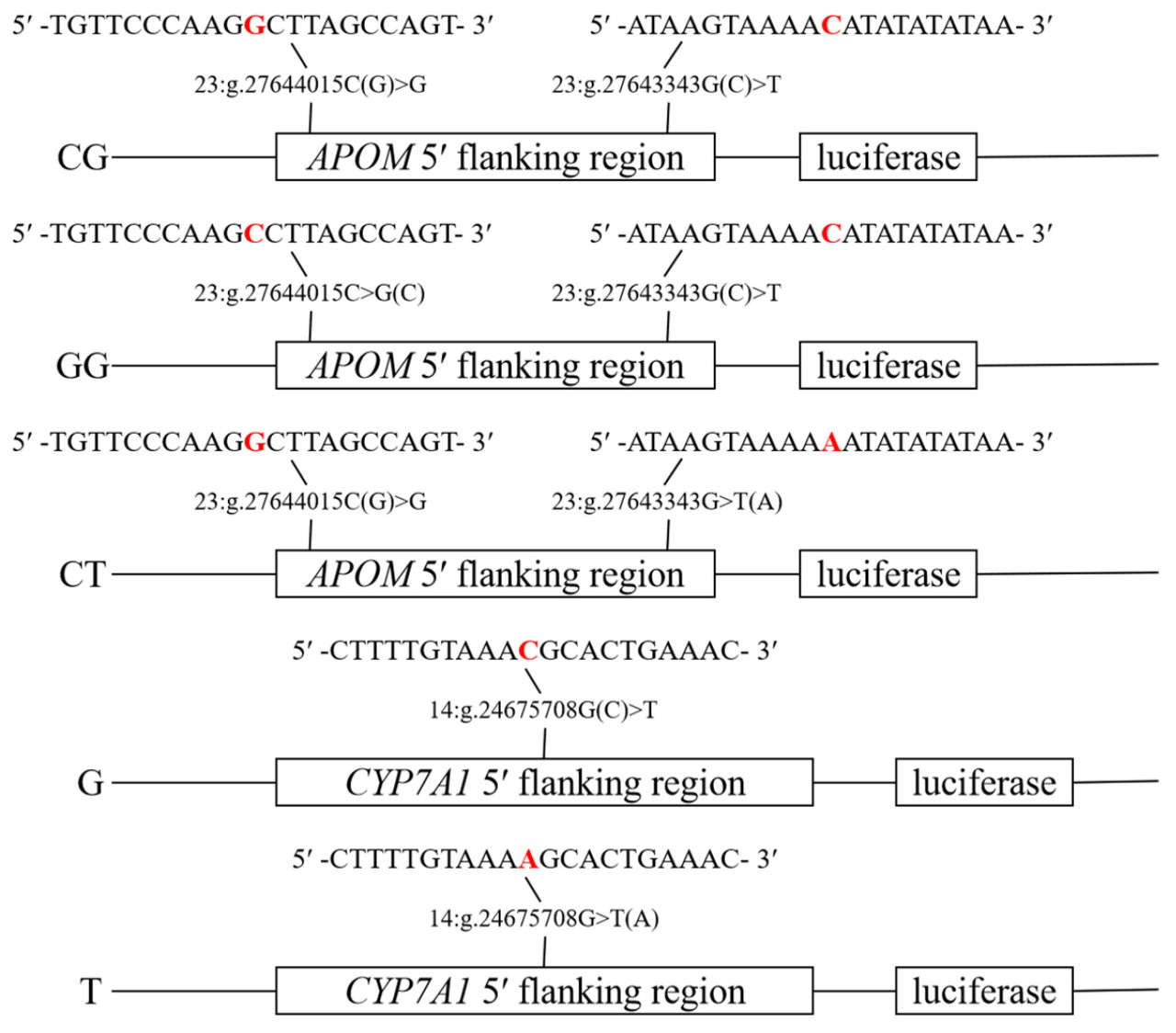
Чтобы исследовать влияние однонуклеотидных полиморфизмов (SNP), которые, как предполагалось, модифицируют сайты связывания факторов транскрипции в 5'-фланкирующей области генов APOM и CYP7A1 , мы сконструировали три и два фрагмента репортерного гена люциферазы соответственно ( Рисунок 1 ). Мы синтезировали фрагменты, содержащие 2000 п.н. 5'-фланкирующих последовательностей генов APOM и CYP7A1 , с помощью Hitrobio (Пекин, Китай). Затем мы клонировали эти фрагменты в вектор pGL4.14 Luciferase Assay Vector (Promega, Мэдисон, Висконсин, США). Мы секвенировали очищенные плазмиды для подтверждения правильного количества каждого фрагмента репортерного гена люциферазы.
Рисунок 1. Эскизы рекомбинантных плазмид. Красный текст обозначает различные аллели. CG, GG и CT: Эскизы гена APOM рекомбинантных плазмид. C и A: Эскизы гена CYP7A1 рекомбинантных плазмид.
Мы инокулировали 1 × 10 5 Т-клеток эмбриональной почки человека (HEK)-293 на лунку 48-луночных планшетов. Мы трансфицировали 250 нг сконструированной плазмидной ДНК вместе с 5 нг репортерного вектора pRL-TK renilla luciferase (Promega) в каждую лунку с помощью Lipofectamine 3000 (Invitrogen, SD, CA, США). Этот процесс повторяли трижды для каждой конструкции. Через 36 часов после трансфекции мы измерили активность люциферазы светлячка и renilla с помощью системы DualLuciferase Reporter Assay System (Promega) с помощью многофункциональной системы детекции для микропланшетов (BioTek, Winooski, VT, США) и рассчитали нормализованные данные по люциферазе (светлячок/renilla).
3. Результаты
3.1. Идентификация однонуклеотидных полиморфизмов (SNP)
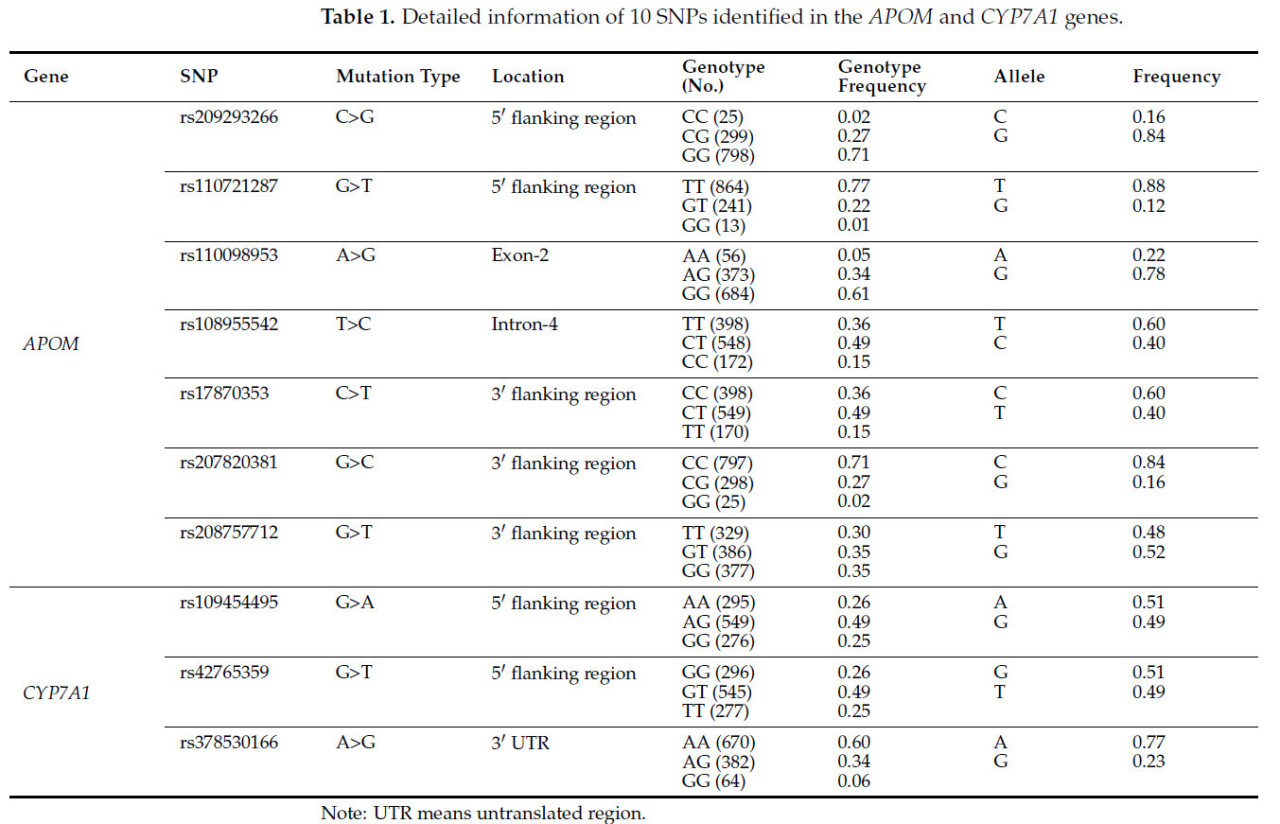
В результате повторного секвенирования всех кодирующих и фланкирующих участков двух генов мы идентифицировали семь и три однонуклеотидных полиморфизма (SNP) в генах APOM и CYP7A1 соответственно. В гене APOM два SNP располагались в 5′-регуляторной области, один — в экзоне 2 (синонимичная мутация), один — в интроне 4 и три — в 3′-регуляторной области. В гене CYP7A1 два SNP располагались в 5′-фланкирующей области и один — в 3′-нетранслируемой области (UTR). Подробная информация о 10 SNP представлена в таблице 1 .
Таблица 1. Подробная информация о 10 однонуклеотидных полиморфизмах (SNP), выявленных в генах APOM и CYP7A1 .
3.2. Генетические ассоциации между выявленными однонуклеотидными полиморфизмами и пятью признаками молока
С помощью программного обеспечения SAS 9.2 мы оценили генетические ассоциации между 10 идентифицированными однонуклеотидными полиморфизмами и 5 признаками молока в популяции из 1123 коров китайской голштинской породы.
Для гена APOM однонуклеотидный полиморфизм rs209293266 был достоверно связан с 305-дневным надоем молока, процентным содержанием жира и белка ( p = <0,01~0,04) в обеих лактациях. Rs110721287 оказал значительное влияние на 305-дневный надой молока, жир и белок, а также процентное содержание белка ( p < 0,01) в первую лактацию и 305-дневный надой молока и процентное содержание жира ( p = <0,01~0,03) во вторую лактацию. Rs110098953 показал значительные генетические корреляции с выходом и процентным содержанием жира, выходом и процентным содержанием белка ( p = <0,01~0,04) в первую лактацию, а также надоем молока, жиром и белком, а также процентным содержанием жира ( p = <0,01~0,02) во вторую лактацию. Rs108955542 оказал сильное влияние на надои молока, процентное содержание жира и белка ( p = <0,01~0,02) в течение первой лактации, а также на надои молока, жира, белка и процентное содержание жира ( p = <0,01~0,02) во время второй лактации. Rs17870353 был сильно генетически связан с надоями молока, процентным содержанием жира и белка ( p < 0,01) в течение первой лактации, а также надоями молока, белка и процентным содержанием жира ( p < 0,01) во время второй лактации. Rs207820381 имел выраженные генетические корреляции с надоями молока, процентным содержанием жира и белка ( p < 0,01) в течение первой лактации, а также с выходом жира и процентным содержанием жира ( p < 0,01) во время второй лактации. Однонуклеотидный полиморфизм rs208757712 имел значимые ассоциации с четырьмя характеристиками молока, за исключением надоя ( p = <0,01~0,02) в течение первой лактации и выхода белка ( p < 0,01) в течение второй лактации ( таблица S3 ).
В CYP7A1 , rs109454495 имел выраженные ассоциации с надоем молока, выходом белка и процентом в течение первой лактации, а также процентом жира, выходом белка и процентом ( p = <0,01~0,02) во время второй лактации. Однонуклеотидный полиморфизм rs42765359 отчетливо коррелировал с надоем молока и белка и процентом жира и белка в течение первой лактации, а также процентом жира, выходом белка и процентом ( p = <0,01~0,02) во время второй лактации. Rs378530166 был значимо связан с четырьмя признаками молока, за исключением процента белка в течение первой лактации и надоя молока и белка и процента жира и белка ( p < 0,01) во время второй лактации ( Таблица S4 ).
Соответственно, мы рассчитали аддитивный (a), доминантный (d) и аллельный (α) эффекты выявленных однонуклеотидных полиморфизмов ( таблица S5 ). Было выявлено, что десять однонуклеотидных полиморфизмов показали значимый аддитивный эффект на процентное содержание белка ( p < 0,05), восемь – на процентное содержание жира ( p < 0,05), семь – на надои молока ( p < 0,05) и пять – на выход жира и выход белка ( p < 0,05). Аналогичным образом, аллельный эффект этих 10 однонуклеотидных полиморфизмов был значимым как минимум для одного признака молочной продуктивности ( p < 0,05).
3.3 Генетические ассоциации между блоками гаплотипов с пятью признаками молока
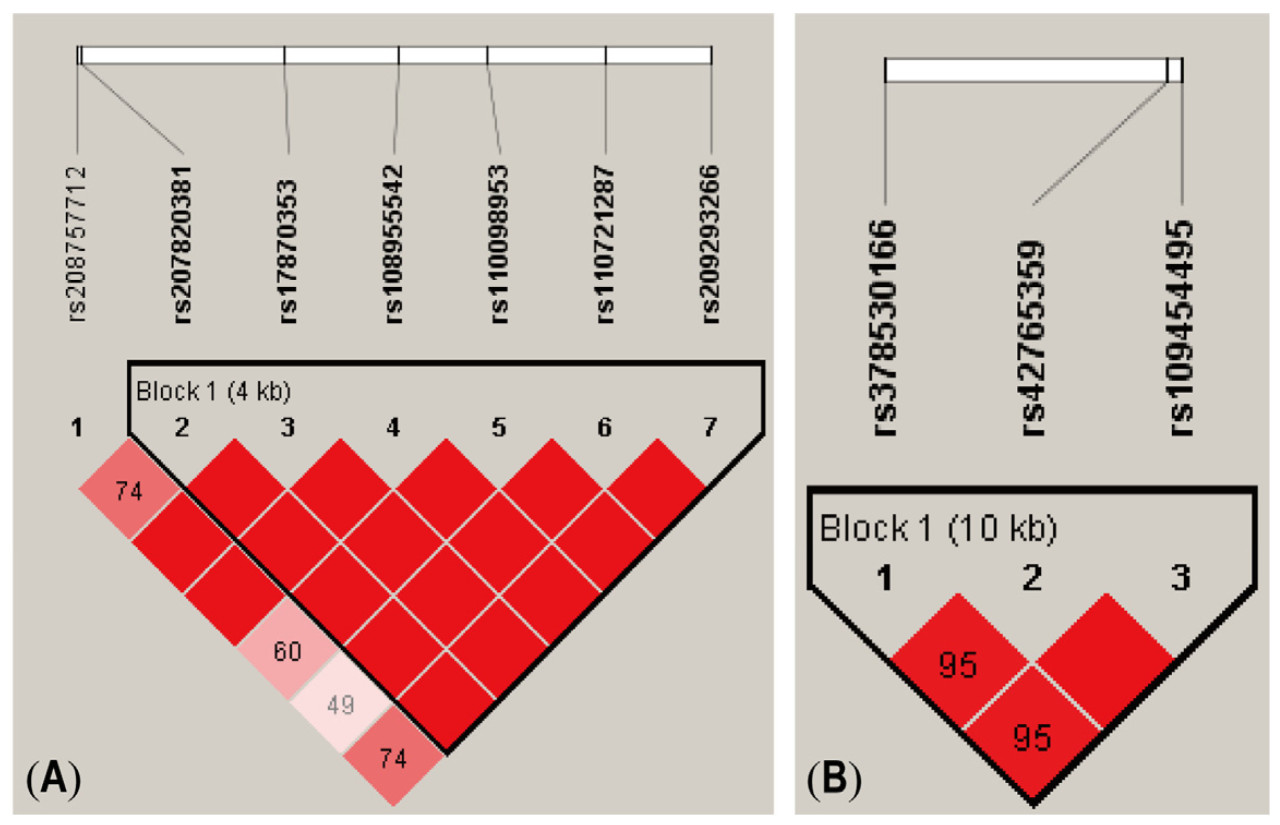
Используя Haploview4.2, мы оценили степень LD среди 7 SNP в APOM и 3 SNP в CYP7A1 . В гене APOM 6 SNP, rs209293266, rs110721287, rs110098953, rs108955542, rs17870353 и rs207820381, составили блок гаплотипов ( Рисунок 2 А), в котором частота пяти гаплотипов, H1 (CTGCTG), H2 (CGACTG), H3 (GGACTC), H4 (CGATCG) и H5 (CGATTG), составила 0,40, 0,23, 0,15, 0,12 и 0,10 соответственно. В гене CYP7A1 было обнаружено 3 тесно связанных однонуклеотидных полиморфизма (ОНП), образующих один блок гаплотипа ( рисунок 2 B), а частота H1 (ATG), H2 (AGA) и H3 (GGA) составила 0,49, 0,28 и 0,22 соответственно.
Рисунок 2. Распространение LD среди выявленных SNP в APOM и CYP7A1 . Текст над блоками гаплотипов содержит названия SNP. Числа в блоках гаплотипов представляют D′: чем краснее блоки гаплотипа, тем сильнее LD. ( A ) LD среди 7 SNP в APOM . ( B ) LD среди 3 SNP в CYP7A1 .
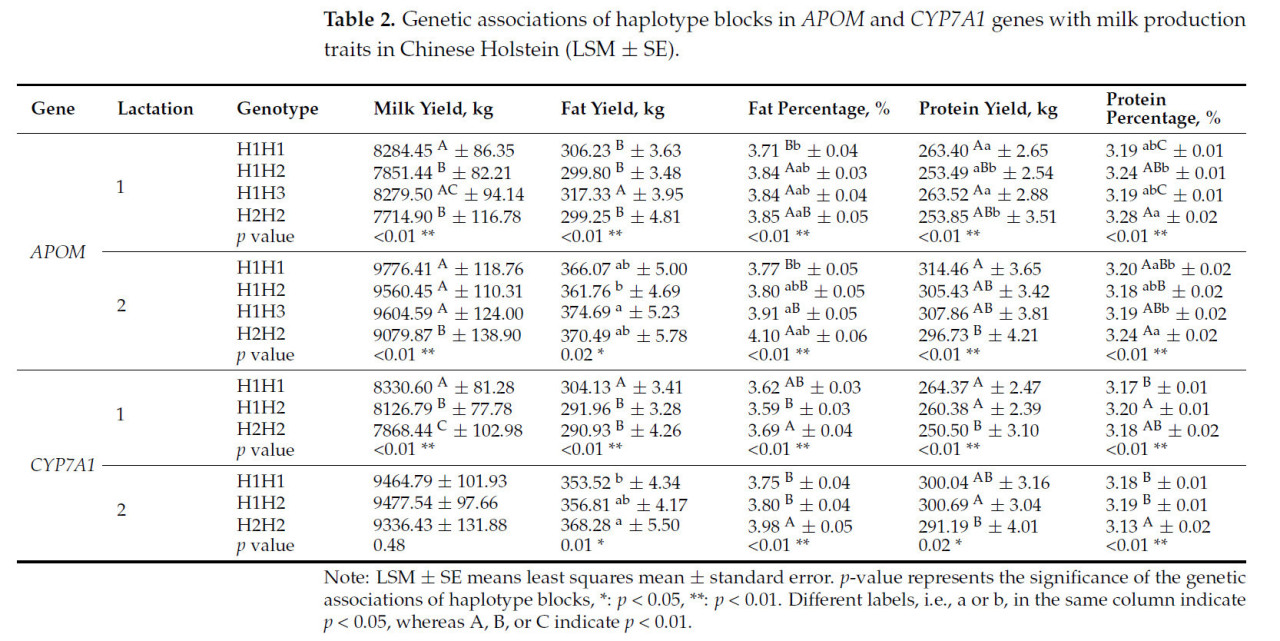
Ассоциативный анализ на основе гаплотипа показал, что блок гаплотипа APOM имел высокую генетическую корреляцию со всеми пятью признаками молока в течение обеих лактаций ( p = <0,01~0,02). Кроме того, блок CYP7A1 продемонстрировал высокую значимую корреляцию с признаками удоя и состава молока в течение первой лактации ( p < 0,01) и признаками содержания жира и белка в молоке во время второй лактации ( p = <0,01~0,02) ( таблица 2 ).
Таблица 2. Генетические ассоциации блоков гаплотипов в генах APOM и CYP7A1 с признаками молочной продуктивности у китайских голштинов (LSM ± SE).
3.4 Изменения транскрипционной активности, вызванные четырьмя однонуклеотидными полиморфизмами в промоторных областях
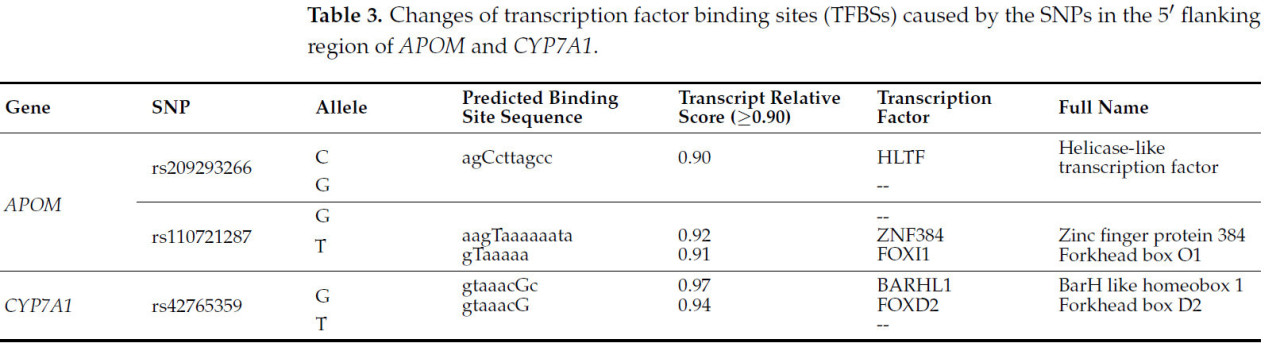
Во-первых, мы использовали программное обеспечение Jaspar 2024 для оценки замен TFBS трех SNP (rs209293266, rs110721287 и rs42765359) в 5′-промоторной области генов APOM и CYP7A1 (таблица 3).
Таблица 3. Изменения участков связывания факторов транскрипции (TFBS), вызванные однонуклеотидными полиморфизмами (SNP) в 5′-фланкирующей области APOM и CYP7A1 .
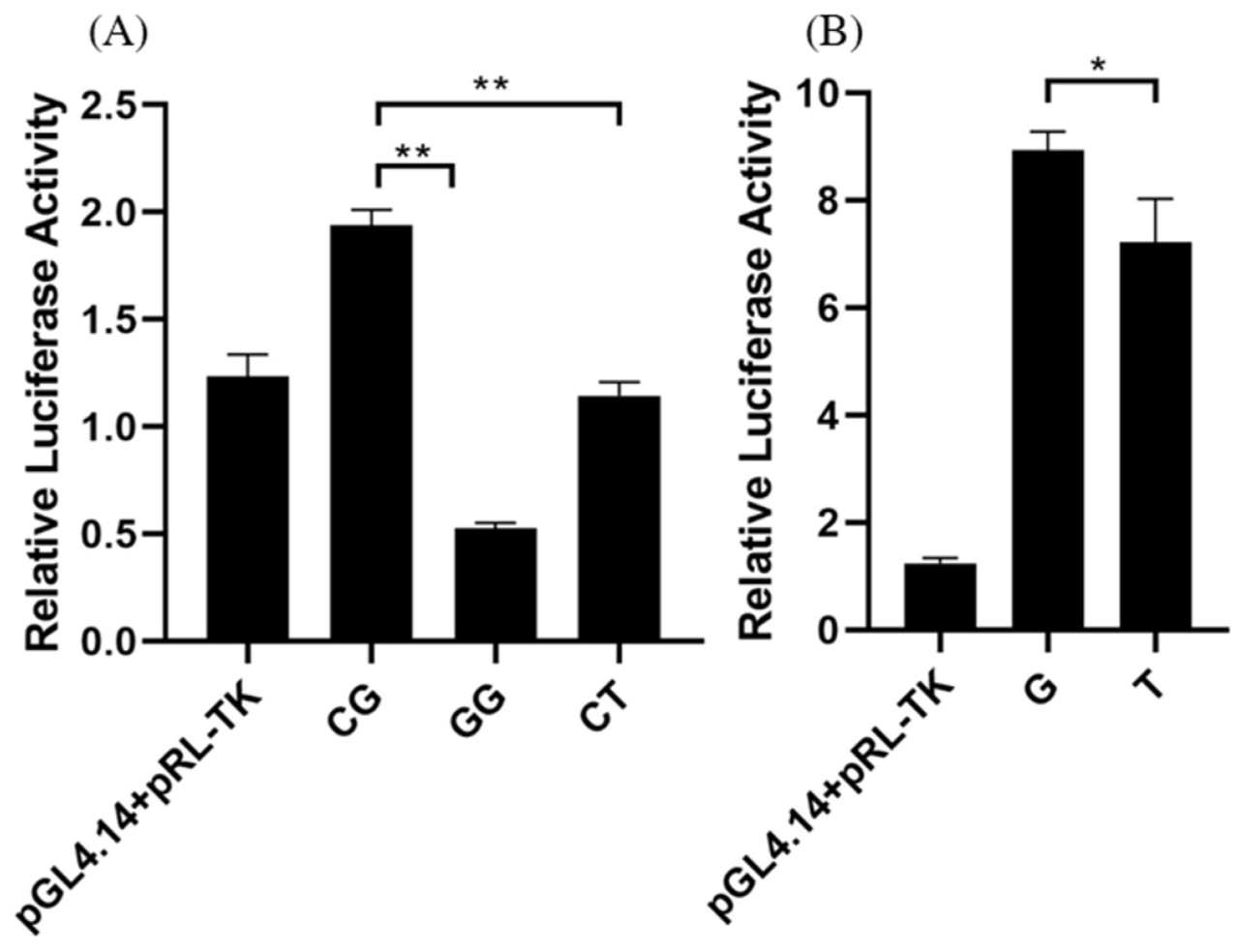
Впоследствии, проведя анализ люциферазы, мы обнаружили, что активность люциферазы гаплотипа CG была значительно выше, чем у pGL4.14 и pRL-TK ( p < 0,01), а также у конструкции GG ( p < 0,01) и CT ( p < 0,01, рисунок 3 А), что указывает на то, что присутствие аллеля G в rs209293266 и аллеля T в rs110721287 приводило к снижению транскрипционной активности гена APOM по сравнению с аллелями C и G соответственно. Кроме того, активность люциферазы конструкции, содержащей аллель G в rs42765359 гена CYP7A1 , была значительно выше, чем у pGL4.14 и pRL-TK ( p < 0,01), а также у конструкции, содержащей аллель T ( p = 0,02, рисунок 3 B), что указывает на то, что аллель T снижает транскрипционную активность CYP7A1 по сравнению с аллелем G.
Рисунок 3. Результаты анализа люциферазы в клетках HEK293. pGL4.14 + pRL-TK: пустой вектор. ( A ) Анализ активности люциферазы rs209293266 и rs110721287 в гене APOM . ( B ) Анализ активности люциферазы rs42765359 в гене CYP7A1 . **: p < 0,01. *: p < 0,05.
3.5 Изменения вторичной структуры мРНК, вызванные двумя однонуклеотидными полиморфизмами (ОНП) в экзоне и нетранслируемой области (UTR)
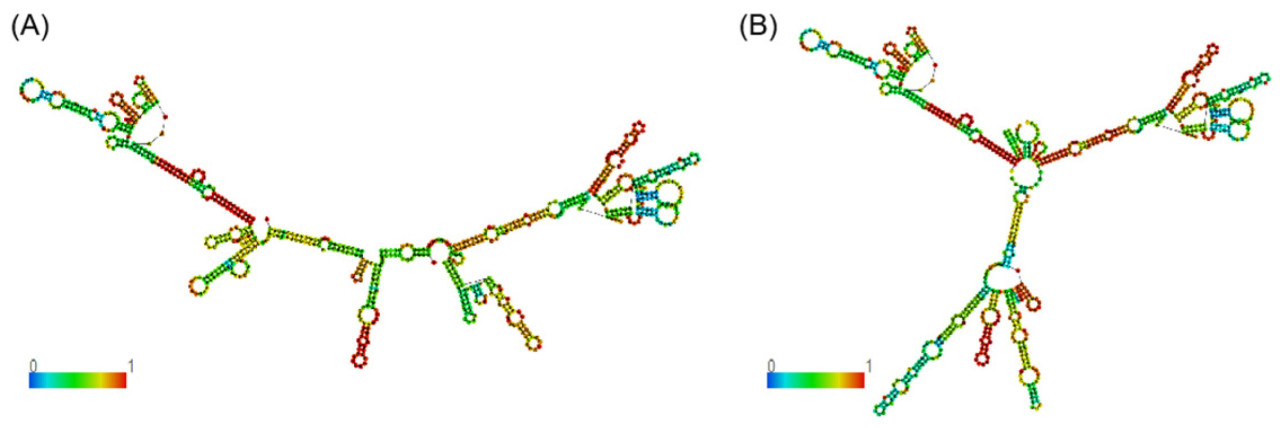
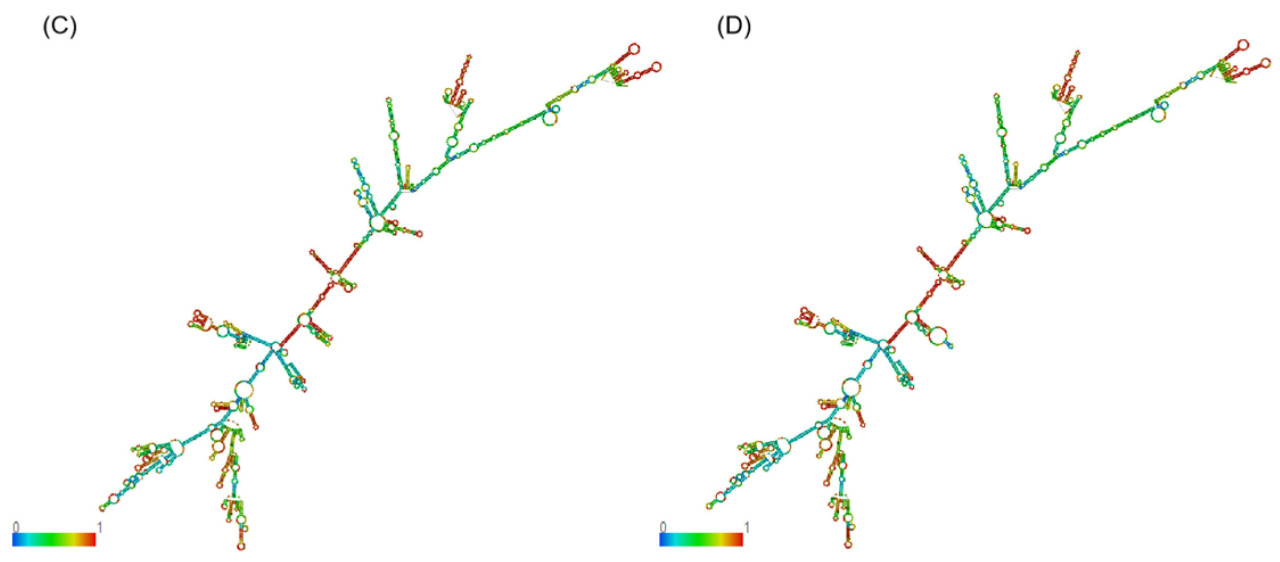
С помощью веб-сервера RNAfold было предсказано, что rs110098953 в APOM изменяет вторичную структуру РНК до и после мутации ( рисунок 4 A, B), тогда как rs378530166 в CYP7A1 не изменяет вторичную структуру РНК ( рисунок 4 C, D). Когда G заменяет A в rs110098953, а G заменяет A в rs378530166, было предсказано, что MFE вторичных структур мРНК увеличивается ( таблица 4 ), тем самым вызывая нестабильность вторичных структур мРНК APOM и CYP7A1 , что может повлиять на экспрессию двух генов.
Рисунок 4. Прогнозирование вторичной структуры мРНК для однонуклеотидных полиморфизмов (SNP) в экзонах и нетранслируемых областях APOM и CYP7A1 . ( A ) Вторичная структура мРНК мутации A rs110098953 в APOM . ( B ) Вторичная структура мРНК мутации G rs110098953 в APOM . ( C ) Вторичная структура мРНК мутации A rs378530166 в CYP7A1. (D) Вторичная структура мРНК мутации G rs378530166 в CYP7A1 . Различные цвета представляют вероятность комплементарного спаривания оснований в последовательностях РНК, при этом вероятность варьируется от 0 до 1 с использованием синего к красному.
Таблица 4. Значения минимальной свободной энергии (МСЭ) оптимальной вторичной структуры мРНК APOM и CYP7A1 .
4. Обсуждение
В нашем предыдущем исследовании был выявлен повышенный уровень экспрессии генов APOM и CYP7A1 в печени коров голштинской породы в период сухостоя и лактации. Примечательно, что эти гены играют ключевую роль в путях PPAR и расположены вблизи выявленных QTL, связанных с признаками молочной продуктивности. В настоящем исследовании мы представили дополнительные генетические доказательства, подтверждающие значительное влияние APOM и CYP7A1 на показатели надоя и состава молока у молочных коров.
APOM является членом семейства аполипопротеинов (APO), которое играет решающую роль в метаболизме и превращении холестерина и триацилглицеридов. Эти белки связывают и транспортируют липиды в различные ткани организма для метаболизма и использования [ 13 , 14 ]. Исследования показали, что нокаутирование гена APOM может усилить экспрессию белка 1, связывающего регуляторный элемент транскрипционного фактора стерол-фактора (SREBP1), который связан с белками, участвующими в синтезе жирных кислот в печени мышей [ 15 , 16 ]. APOM участвует в метаболизме липидов и инсулинорезистентности, в частности в синтезе пре-β-ЛПВП и обратном транспорте холестерина [ 17 , 18 , 19 ], и этот процесс позволяет транспортировать холестерин к частицам ЛПВП, которые затем возвращаются в печень для преобразования в жирные кислоты через лимитирующий фермент CYP7A1 [ 20 ]. Предыдущие исследования показали, что генетические полиморфизмы гена CYP7A1 связаны с метаболизмом глюкозы и липопротеинов, гомеостазом, концентрацией дезоксихолевой кислоты, диабетом 2 типа, а также с более высокими концентрациями холестерина ЛПНП и триацилглицеридов у человека [ 21 , 22 ]. Эти результаты согласуются с результатами нашего исследования, согласно которым гены APOM и CYP7A1 оказывают значительное влияние на показатели жирности молока. Значимая связь этих двух генов с показателями надоя и содержания молочного белка обусловлена главным образом высокой генетической связью и корреляцией между показателями молочной продуктивности.
В целом, 5′ фланкирующая область регулирует экспрессию генов путем связывания факторов транскрипции, которые являются ДНК-связывающими белками и специфически связываются с определенными последовательностями ДНК. Следовательно, определенные SNP в этих областях могут изменять связывание TF, что приводит к модификациям активности транскрипции генов, тем самым влияя на ассоциированные признаки [ 23 , 24 , 25 ]. В этом исследовании аллель C для rs209293266 в APOM мог связываться с фактором транскрипции HLTF, а аллель G для rs42765359 в CYP7A1 мог связывать BARHL1 и FOXD2, тем самым повышая уровни экспрессии APOM и CYP7A1 , соответственно. Аналогичным образом, сообщалось, что эти три фактора транскрипции усиливают экспрессию целевых генов у людей и мышей [ 26 , 27 , 28 ]. Напротив, аллель T rs110721287 в APOM может создавать два TFBS для ZNF384 и FOXI1, тем самым подавляя экспрессию их целевых генов [ 29 , 30 ]. Таким образом, мы предполагаем, что три регуляторных SNP изменили TFBS, тем самым приведя к связыванию факторов транскрипции HLTF с геном APOM и BARHL1 и FOXD2 с геном CYP7A1 для активации экспрессии APOM и CYP7A1 , что, в свою очередь, способствует формированию признаков молочного жира и белка.
Кроме того, вторичная структура мРНК не только напрямую влияет на стабильность своей собственной структуры, но и регулирует биологические процессы, такие как инициация трансляции РНК, расширение и скорость процесса трансляции и сворачивание посттранслированных белков, влияя на функцию высших структур РНК [ 31 , 32 ]. В этом исследовании мы наблюдали, что аллель A rs110098953 в APOM и аллель A rs378530166 в CYP7A1 привели к более стабильным вторичным структурам мРНК по сравнению с аллелем G, соответственно, о чем свидетельствуют их более низкие MFE. Однако люди с генотипом AA для этих двух SNP показали значительно более высокий выход жира, процент жира и выход белка по сравнению с людьми с генотипом GG. Возможно, что другие сложные факторы в процессе транскрипции способствовали этим результатам.
Исследования показали, что интеграция информации о функциональных локусах со значительным генетическим воздействием на желаемые целевые признаки может повысить точность генетического отбора [ 33 , 34 ]. В этом исследовании H1H3 в APOM и H1H1 в CYP7A1 показали лучшие показатели надоев и состава молока. Можно рассмотреть возможность разведения в соответствии с этими комбинациями генов и добавления их в массив чипов; однако они требуют дальнейшего углубленного изучения. В будущем иммунопреципитация хроматина (ChIP), анализ сдвига электрофоретической подвижности (EMSA), мутация одного основания и другие тесты могут быть использованы для проверки того, как эти мутации регулируют биологические функции генов APOM и CYP7A1 и их влияние на образование молока, тем самым предоставляя ценные молекулярные маркеры для геномного отбора молочных коров.
5. Выводы
Наше первоначальное исследование РНК-секвенирования выявило гены APOM и CYP7A1 как перспективные кандидаты для определения признаков молока у молочного скота. В настоящем исследовании были дополнительно продемонстрированы их значимые генетические ассоциации с признаками надоя и состава молока. В частности, пять однонуклеотидных полиморфизмов (SNP) рассматривались как потенциальные причинные мутации, которые могут влиять на признаки молока, включая rs209293266 и rs110721287 в APOM , rs42765359 в CYP7A1 , которые изменили TFBS, и rs110098953 в APOM и rs378530166 в CYP7A1 , которые изменили вторичную структуру мРНК. Наши результаты послужили основой для дальнейшего изучения роли генов APOM и CYP7A1 и предоставили ценные молекулярные маркеры для программ геномной селекции у молочного скота.
Дополнительные материалы
Следующая вспомогательная информация может быть загружена по адресу: https://www.mdpi.com/article/10.3390/agriculture15010033/s1 , Таблица S1: Описательная статистика фенотипических значений признаков молочной продуктивности; Таблица S2: 26 парных ПЦР-праймеров генов APOM и CYP7A1 ; Таблица S3: Генетические ассоциации однонуклеотидных полиморфизмов (SNP) гена APOM с признаками молочной продуктивности у китайского голштина (LSM ± SE); Таблица S4: Генетические ассоциации однонуклеотидных полиморфизмов (SNP) гена CYP7A1 с признаками молочной продуктивности у китайского голштина (LSM ± SE); Таблица S5: Аддитивные, доминантные и аллельные эффекты замены 10 однонуклеотидных полиморфизмов (SNP) генов APOM и CYP7A1 на показатели надоев и состава молока у китайского голштина в течение двух лактаций.
Ссылки
1. Guinan, F.L.; Wiggans, G.R.; Norman, H.D.; Dürr, J.W.; Cole, J.B.; Van Tassell, C.P.; Misztal, I.; Lourenco, D. Changes in genetic trends in US dairy cattle since the implementation of genomic selection. J. Dairy Sci. 2023, 106, 1110–1129. [Google Scholar] [CrossRef]
2. de Las, H.S.; Lopez, B.I.; Moghaddar, N.; Park, W.; Park, J.E.; Chung, K.Y.; Lim, D.; Lee, S.H.; Shin, D.; van der Werf, J. Use of gene expression and whole-genome sequence information to improve the accuracy of genomic prediction for carcass traits in Hanwoo cattle. Genet. Sel. Evol. 2020, 52, 54. [Google Scholar] [CrossRef]
3. Liang, R.; Han, B.; Li, Q.; Yuan, Y.; Li, J.; Sun, D. Using RNA sequencing to identify putative competing endogenous RNAs (ceRNAs) potentially regulating fat metabolism in bovine liver. Sci. Rep. 2017, 1, 6396. [Google Scholar] [CrossRef] [PubMed]
4. Nielsen, L.B.; Christoffersen, C.; Ahnström, J.; Dahlbäck, B. ApoM: Gene regulation and effects on HDL metabolism. Trends Endocrin. Met. 2009, 20, 66–71. [Google Scholar] [CrossRef]
5. Ren, K.; Tang, Z.-L.; Jiang, Y.; Tan, Y.-M.; Yi, G.-Y. Apolipoprotein M. Clin. Chim. Acta 2015, 446, 21–29. [Google Scholar] [CrossRef]
6. Liu, M.; Seo, J.; Allegood, J.; Bi, X.; Zhu, X.; Boudyguina, E.; Gebre, A.K.; Avni, D.; Shah, D.; Sorci-Thomas, M.G.; et al. Hepatic Apolipoprotein M (ApoM) Overexpression Stimulates Formation of Larger ApoM/Sphingosine 1-Phosphate-enriched Plasma High Density Lipoprotein. J. Biol. Chem. 2014, 289, 2801–2814. [Google Scholar] [CrossRef] [PubMed]
7. Feingold, K.R. Lipid and Lipoprotein Metabolism. Endocrin. Metab. Clin. 2022, 3, 437–458. [Google Scholar] [CrossRef] [PubMed]
8. Hubacek, J.A.; Bobkova, D. Role of Cholesterol 7α-Hydroxylase (CYP7A1) in Nutrigenetics and Pharmacogenetics of Cholesterol Lowering. Mol. Diagn. Ther. 2006, 10, 93–100. [Google Scholar] [CrossRef]
9. Oster, H. CYP7A1: A Liver Circadian Clock Output Mediating the Metabolic Effects of Sleep Disruption. Cell. Mol. Gastroenter. 2015, 1, 574–575. [Google Scholar] [CrossRef]
10. Fuchs, M. Bile acid regulation of hepatic physiology: III. Regulation of bile acid synthesis: Past progress and future challenges. Am. J. Physiol.-Gastr. Liver 2003, 284, G551–G557. [Google Scholar] [CrossRef] [PubMed]
11. Li, T.; Francl, J.M.; Boehme, S.; Ochoa, A.; Zhang, Y.; Klaassen, C.D.; Erickson, S.K.; Chiang, J.Y. Glucose and insulin induction of bile acid synthesis: Mechanisms and implication in diabetes and obesity. J. Biol. Chem. 2012, 287, 1861–1873. [Google Scholar] [CrossRef] [PubMed]
12. Rizzolo, D.; Kong, B.; Taylor, R.E.; Brinker, A.; Goedken, M.; Buckley, B.; Guo, G.L. Bile acid homeostasis in female mice deficient in Cyp7a1 and Cyp27a1. Acta Pharm. Sin. B 2021, 11, 3847–3856. [Google Scholar] [CrossRef] [PubMed]
13. Faber, K.; Axler, O.; Dahlbäck, B.; Nielsen, L.B. Characterization of apoM in normal and genetically modified mice. J. Lipid Res. 2004, 45, 1272–1278. [Google Scholar] [CrossRef] [PubMed]
14. Cheng, G.; Zheng, L. Regulation of the apolipoprotein M signaling pathway: A review. J. Recept. Sig. Transd. 2022, 42, 285–292. [Google Scholar] [CrossRef]
15. Shimano, H.; Sato, R. SREBP-regulated lipid metabolism: Convergent physiology—Divergent pathophysiology. Nat. Rev. Endocrinol. 2017, 13, 710–730. [Google Scholar] [CrossRef] [PubMed]
16. Zhang, X.; Zhang, P.; Gao, J.; Huang, Q. Autophagy dysregulation caused by ApoM deficiency plays an important role in liver lipid metabolic disorder. Biochem. Biophys. Res. Commun. 2018, 495, 2643–2648. [Google Scholar] [CrossRef]
17. Xu, N.; Dahlbäck, B. A Novel Human Apolipoprotein (apoM). J. Biol. Chem. 1999, 274, 31286–31290. [Google Scholar] [CrossRef]
18. Wolfrum, C.; Poy, M.N.; Stoffel, M. Apolipoprotein M is required for prebeta-HDL formation and cholesterol efflux to HDL and protects against atherosclerosis. Nat. Med. 2005, 11, 418–422. [Google Scholar] [CrossRef] [PubMed]
19. Kurano, M.; Tsukamoto, K.; Shimizu, T.; Kassai, H.; Nakao, K.; Aiba, A.; Hara, M.; Yatomi, Y. Protection Against Insulin Resistance by Apolipoprotein M/Sphingosine-1-Phosphate. Diabetes 2020, 69, 867–881. [Google Scholar] [CrossRef]
20. Chambers, K.F.; Day, P.E.; Aboufarrag, H.T.; Kroon, P.A. Polyphenol Effects on Cholesterol Metabolism via Bile Acid Biosynthesis, CYP7A1: A Review. Nutrients 2019, 11, 2588. [Google Scholar] [CrossRef] [PubMed]
21. Fall, T.; Salihovic, S.; Brandmaier, S.; Nowak, C.; Ganna, A.; Gustafsson, S.; Broeckling, C.D.; Prenni, J.E.; Kastenmuller, G.; Peters, A.; et al. Non-targeted metabolomics combined with genetic analyses identifies bile acid synthesis and phospholipid metabolism as being associated with incident type 2 diabetes. Diabetologia 2016, 59, 2114–2124. [Google Scholar] [CrossRef] [PubMed]
22. Islam, M.S.; Sharif, A.; Kwan, N.; Tam, K.C. Bile Acid Sequestrants for Hypercholesterolemia Treatment Using Sustainable Biopolymers: Recent Advances and Future Perspectives. Mol. Pharm. 2022, 19, 1248–1272. [Google Scholar] [CrossRef] [PubMed]
23. Liu, F.; Wang, K.; Deng, L.; Liu, X.; Wu, M.; Wang, T.; Cui, L.; Li, Z. Transcription factor E2F3a regulates CASP8AP2 transcription and enhances sensitivity to chemotherapeutic drugs in acute lymphoblastic leukemia. Cancer Cell Int. 2018, 18, 40. [Google Scholar] [CrossRef]
24. Zhang, X.; Li, Y.; Sun, Y.; Guo, M.; Feng, J.; Wang, Y.; Zhang, Z. Regulatory effect of heat shock transcription factor-1 gene on heat shock proteins and its transcriptional regulation analysis in small abalone Haliotis diversicolor. BMC Cell Biol. 2020, 21, 1–83. [Google Scholar] [CrossRef]
25. Wang, X.; Li, A.; Raza, S.H.A.; Liang, C.; Zhang, S.; Mei, C.; Yang, W.; Zan, L. Transcription Factors ZEB1 and CREB Promote the Transcription of Bovine ABHD5 Gene. DNA Cell Biol. 2021, 40, 219–230. [Google Scholar] [CrossRef] [PubMed]
26. Li, S.; Qiu, F.; Xu, A.; Price, S.M.; Xiang, M. Barhl1 Regulates Migration and Survival of Cerebellar Granule Cells by Controlling Expression of the Neurotrophin-3 Gene. J. Neurosci. 2004, 24, 3104–3114. [Google Scholar] [CrossRef]
27. Li, R.; Chen, S.; Zhan, J.; Li, X.; Liu, W.; Sheng, X.; Lu, Z.; Zhong, R.; Chen, L.; Luo, X.; et al. Long noncoding RNA FOXD2-AS1 enhances chemotherapeutic resistance of laryngeal squamous cell carcinoma via STAT3 activation. Cell Death Dis. 2020, 11, 41. [Google Scholar] [CrossRef]
28. Wang, Y.; Kelso, A.A.; Karamafrooz, A.; Chen, Y.; Chen, W.; Cheng, C.; Qi, Y.; Gu, L.; Malkas, L.; Taglialatela, A.; et al. Arginine shortage induces replication stress and confers genotoxic resistance by inhibiting histone H4 translation and promoting PCNA ubiquitination. Cell Rep. 2023, 42, 112296. [Google Scholar] [CrossRef]
29. Robling, A.G.; Childress, P.; Yu, J.; Cotte, J.; Heller, A.; Philip, B.K.; Bidwell, J.P. Nmp4/CIZ suppresses parathyroid hormone-induced increases in trabecular bone. J. Cell. Physiol. 2009, 219, 734–743. [Google Scholar] [CrossRef] [PubMed]
30. Childress, P.; Philip, B.K.; Robling, A.G.; Bruzzaniti, A.; Kacena, M.A.; Bivi, N.; Plotkin, L.I.; Heller, A.; Bidwell, J.P. Nmp4/CIZ Suppresses the Response of Bone to Anabolic Parathyroid Hormone by Regulating Both Osteoblasts and Osteoclasts. Calcif. Tissue Int. 2011, 89, 74–89. [Google Scholar] [CrossRef] [PubMed][Green Version]
31. Wan, Y.; Kertesz, M.; Spitale, R.C.; Segal, E.; Chang, H.Y. Understanding the transcriptome through RNA structure. Nat. Rev. Genet. 2011, 12, 641–655. [Google Scholar] [CrossRef] [PubMed]
32. Dethoff, E.A.; Chugh, J.; Mustoe, A.M.; Al-Hashimi, H.M. Functional complexity and regulation through RNA dynamics. Nature 2012, 482, 322–330. [Google Scholar] [CrossRef] [PubMed]
33. Zhang, Z.; Ober, U.; Erbe, M.; Zhang, H.; Gao, N.; He, J.; Li, J.; Simianer, H. Improving the accuracy of whole genome prediction for complex traits using the results of genome wide association studies. PLoS ONE 2014, 9, e93017. [Google Scholar] [CrossRef] [PubMed]
34. Brondum, R.F.; Su, G.; Janss, L.; Sahana, G.; Guldbrandtsen, B.; Boichard, D.; Lund, M.S. Quantitative trait loci markers derived from whole genome sequence data increases the reliability of genomic prediction. J. Dairy Sci. 2015, 98, 4107–4116. [Google Scholar] [CrossRef] [PubMed]
Liu Y, Guo Z, Ni J, Yang C, Han B, Ma Y, Li J, Jiang G, Zheng W, Sun D. Identification of Genetic Markers of APOM and CYP7A1 Genes Affecting Milk Production Traits in Chinese Holstein. Agriculture. 2025; 15(1):33. https://doi.org/10.3390/agriculture15010033
Перевод статьи «Identification of Genetic Markers of APOM and CYP7A1 Genes Affecting Milk Production Traits in Chinese Holstein» авторов Liu Y, Guo Z, Ni J, Yang C, Han B, Ma Y, Li J, Jiang G, Zheng W, Sun D., оригинал доступен по ссылке. Лицензия: CC BY. Изменения: переведено на русский язык
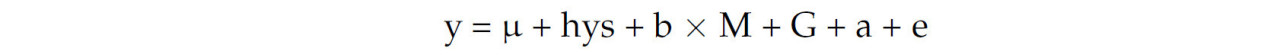































































































Комментарии (0)