Как рождается цвет перца: ученые раскрыли секреты окраски на генном уровне
Транскриптомный и метаболомный анализы выявляют различия в синтезе флавоноидов в процессе развития околоплодника Capsicum frutescens.
Аннотация
Capsicum frutescens (перец кустарниковый) является ценной экономической культурой, широко культивируемой благодаря своему уникальному вкусу и богатому питательному составу. Хотя некоторые исследования показали различия в содержании флавоноидов среди разных видов перцев, механизм, с помощью которого изменения в составе флавоноидов приводят к вариациям цвета плодов у C. frutescens, остается малоизученным. Мы провели транскриптомное и широкоцелевое метаболомное секвенирование на трех различных стадиях роста плода C. frutescens и проанализировали данные, чтобы лучше понять механизм изменения цвета. Основываясь на предыдущих исследованиях генов, регулирующих флавоноидные соединения, и MBW-комплекса, мы идентифицировали в общей сложности 28 ключевых генов, связанных с биосинтезом флавоноидов, и 8 генов, которые могут быть связаны с синтезом флавоноидов. С помощью широкоцелевого метаболомного анализа было идентифицировано 581 дифференциально экспрессируемое метаболитное вещество, включая 43 флавоноида. Было обнаружено, что большинство антоцианов, флавонолов и флавононов присутствуют в большем количестве на стадии незрелого плода, что, как мы предполагаем, связано с дифференциальной экспрессией генов, вовлеченных в биосинтез и регуляцию флавоноидов. Эти результаты представляют собой полезную основу для понимания синтеза флавоноидов и их накопления в плодах C. frutescens.
1. Введение
Окраска плодов перца меняется в процессе их роста, что представляет собой интересное явление. Часто люди определяют, созрел ли плод, именно по изменению его цвета. Механизм изменения цвета плода давно интересует селекционеров и потребителей [1]. Красивый цвет плода может значительно повысить экономическую ценность культуры [2]. Предыдущие исследования показали, что цвет плодов перца в основном связан с флавоноидами, каротиноидами и хлорофиллом [3]. Флавоноиды, как важные вторичные метаболиты, широко распространены в растениях. Благодаря технологическому прогрессу, было идентифицировано около 9000 флавоноидов в растениях [4]. Эти соединения имеют общую основную структуру, состоящую из 15 атомов углерода, и могут быть разделены на шесть основных классов в зависимости от молекулярной структуры: флавонолы, флаваноны, флавоны, изофлавоны, катехины и антоцианы [5].
Синтез флавоноидных соединений начинается с фенилаланина (L-фенилаланина) через фенилпропаноидный и флавоноидный пути. В фенилпропаноидном пути фенилаланин превращается в п-кумароил-КоА под действием трех ключевых ферментов: фенилаланин-аммиак-лиазы (PAL), 4-кумарат-КоА-лигазы (4CL) и циннамат-4-гидроксилазы (C4H) [6]. Это соединение служит предшественником для биосинтеза флавоноидов. Под действием халконсинтазы (CHS) и халконизомеразы (CHI) из п-кумароил-КоА синтезируется нарингенин [7]. Нарингенин является критическим промежуточным продуктом в пути биосинтеза флавоноидов, который подвергается ряду ферментативных реакций с образованием различных типов флавоноидных соединений. Флавоноиды, являясь полифенольными веществами, играют ключевые роли в физиологических процессах растений, включая взаимодействия между растениями и животными, привлечение опылителей, защиту от ультрафиолетового (УФ) повреждения и участие в пигментации органов растений [4,8]. Дополнительно к структурным генам, сообщалось, что транскрипционные факторы регулируют биосинтез флавоноидов, модулируя экспрессию генов их биосинтеза. Например, в этой регуляции участвуют транскрипционные факторы basic helix–loop–helix (bHLH), WD repeat (WD) и R2R3 MYB, а также белки MADS-box. Исследование Koes и др. показало, что белки bHLH и WD взаимодействуют с MYB транскрипционными факторами, образуя MBW комплекс (MYB–bHLH–WD repeat), который регулирует накопление флавоноидов в растениях [9]. Дальнейшие исследования на арабидопсисе также подтвердили тесную связь между MYB транскрипционными факторами и биосинтезом флавоноидов [10].
Цвет плодов C. frutescens меняется на различных стадиях развития. Эти изменения в основном обусловлены накоплением флавоноидов и каротиноидов [11]. Биосинтез антоцианов в основном зависит от биосинтеза флавоноидов и в основном происходит в околоплоднике перцев. Исследования антоцианов идентифицировали, что CaANT1, CaANT2, CaAN1 и CaTTG1 участвуют в их накоплении, влияя на вариацию цвета плода [12]. Howard дополнительно сообщил о значительных различиях в накоплении флавоноидов и связанных с ними метаболитов в плодах перца на разных стадиях развития и в разных условиях окружающей среды [13].
Хотя некоторые гены, связанные с цветом плода, были клонированы у C. frutescens, механизмы, с помощью которых эти гены регулируют накопление флавоноидов и влияют на изменение цвета плода, все еще остаются малоизученными. Хотя цвет плода признан ключевым признаком качества у C. frutescens, большинство исследований было сосредоточено на пигментах, таких как хлорофилл и каротиноиды. Крупномасштабная идентификация и количественное определение флавоноидов в C. frutescens остаются редкими. В этом исследовании мы использовали широкоцелевую метаболомику для обнаружения и количественного определения флавоноидов в околоплоднике C. frutescens на основе платформы анализа UPLC-MS/MS и собственной базы данных. Эукариотическая транскриптомика также была использована для секвенирования ткани околоплодника C. frutescens. Комбинируя данные транскриптомики и метаболомики, мы идентифицировали основные гены, участвующие в синтезе флавоноидов в C. frutescens, и потенциальные гены-кандидаты. Эти результаты были подтверждены с помощью количественной ПЦР в реальном времени (qRT-PCR). Интегрирование данных мульти-омики выявило изменения в накоплении флавоноидов и соответствующих уровнях экспрессии генов в процессе развития цвета плода. Результаты предоставляют важную информацию о регуляции цвета плода у C. frutescens флавоноидными соединениями.
2. Материалы и методы
2.1. Материал C. frutescens, обработка проращивания, условия роста и методы управления
Семена C. frutescens были получены из лаборатории Юго-Западного лесного университета (семена C. frutescens являются диплоидными). Семена C. frutescens промывали в проточной воде, замачивали в дистиллированной воде при 50 °C в течение 15 минут, а затем оставляли при комнатной температуре на 24 часа. Семена помещали в чашку Петри, на дно которой были положены два слоя фильтровальной бумаги для поддержания влажности. Обработанные семена помещали в инкубатор при 25 °C до их прорастания. Впоследствии их высевали в смесь торфа и вермикулита (3:1) и пересаживали в горшки диаметром 20 см. Растения выращивали на опытной базе Юго-Западного лесного университета (долгота 102.454308°, широта 25.035229°) при температуре 25 °C, относительной влажности 60–70% и световом режиме 16 часов света и 8 часов темноты. Их поливали каждые пять дней для поддержания влажности почвы, и растения не удобряли и не обрезали.
Для исследования потенциальных механизмов, стоящих за развитием цвета плода, qRT-PCR использовали для анализа экспрессии генов в кожице плодов C. frutescens на 50, 65 и 80 дней после отцветания (Рисунок 1). Первая стадия (50 дней после отцветания) представляет раннюю стадию развития плодов, на которой плоды полностью зеленые и расширение плода прекратилось, обозначается как стадия GR. Вторая стадия (65 дней после отцветания) соответствует средней стадии развития, в течение которой плод становится полностью оранжевым, обозначается как стадия OR. На третьей стадии (80 дней после отцветания) плод полностью созрел и стал красным, обозначается как стадия RE. После очистки поверхности плода ddH₂O, образцы быстро замораживали в жидком азоте и хранили при -80 °C для дальнейшего анализа.
Рисунок 1. Стадии развития C. frutescens, использованные в этом исследовании. GR: Стадия ранней зрелости плода (50 дней после отцветания); OR: Полуспелая стадия (65 дней после отцветания); RE: Полностью зрелая стадия (80 дней после отцветания).
2.2. Подготовка образца и экстракция для анализа метаболитов
Плоды C. frutescens без повреждений вредителями или болезнями собирали на 50, 65 и 80 дней после отцветания. Поверхности плодов промывали ddH₂O и быстро замораживали в жидком азоте, затем хранили при -80 °C. Образец был отправлен в Wuhan Metware Biotechnology Co., Ltd. (Ухань, Китай) для анализа флавоноидных соединений с использованием широкоцелевого метаболомного анализа на основе платформы детектирования UPLC-MS/MS и собственной базы данных. Хроматографическое разделение проводили на колонке ACQUITY BEH C18 (1.7 мкм, 2.1 мм × 100 мм, Waters Technologies Co., Ltd., Милфорд, Массачусетс, США). Подвижная фаза состояла из растворителя А (0.1% муравьиной кислоты в воде) и растворителя Б (0.1% муравьиной кислоты в ацетонитриле). Градиент элюирования был следующим: 95% растворитель А и 5% растворитель Б в начале, постепенное изменение до 5% растворитель А и 95% растворитель Б за 9 минут, выдерживание в течение 1 минуты. Затем состав возвращали к 95% растворитель А и 5% растворитель Б за 60 секунд и поддерживали в течение 180 секунд. Скорость потока была установлена на 0.35 мл/мин, а температура колонки поддерживалась на уровне 40 °C.
2.3. Анализ дифференциальных метаболитов
Ортогональный анализ проекции на латентные структуры-дискриминантный анализ (OPLS-DA) был выполнен с использованием функции OPLSR.Anal в пакете R Metabo Analyst R (Версия пакета анализа: R версия 3.5.1; Основные параметры анализа: параметры по умолчанию). Данные были логарифмически преобразованы (log2) и центрированы относительно среднего значения перед проведением OPLS-DA анализа. Чтобы избежать переобучения, были проведены тесты перестановки (200 перестановок). В OPLS-DA анализе образцов кожицы плода C. frutescens на разных стадиях развития дифференциальные метаболиты идентифицировали на основе значений VIP (VIP > 1) и абсолютного значения Log2 кратного изменения (|Log2FC| ≥ 1).
2.4. KEGG аннотация и анализ обогащения
Функциональный анализ обогащения является одной из наиболее широко используемых технологий для раскрытия применения последовательностей генов [14]. Это исследование использовало базу данных KEGG для аннотации дифференциальных метаболитов, выявленных на предыдущих этапах, следуя методу Peng и др. [15] . Эти аннотированные метаболиты затем были отображены на метаболические пути в базе данных KEGG Pathway [16] . Пути, содержащие значительно регулируемые метаболиты, были подвергнуты анализу обогащения набора метаболитов (MSEA), при этом значимость определялась по p-значениям из гипергеометрических тестов.
2.5. Транскриптомное секвенирование и анализ данных
Общую РНК экстрагировали из замороженных образцов кожицы C. frutescens на разных стадиях развития с использованием реагента TRIzol® (Thermo Fisher Scientific Inc., Уолтем, Массачусетс, США) в соответствии с инструкциями производителя. Качество и количество общей РНК, экстрагированной из околоплодника C. frutescens, оценивали с использованием флуориметра Qubit (Thermo Fisher Scientific Inc., Уолтем, Массачусетс, США) и высокопроизводительного биоанализатора фрагментов Qsep400 (Vazyme Biotech Co., Ltd., Нанкин, Китай). Для секвенирования РНК 1 мкг общей РНК от каждой из трех стадий развития (с тремя биологическими повторностями на стадию) использовали для построения секвенирующих библиотек с набором NEB Next® Ultra™ RNA Library Prep Kit for Illumina® (New England Biolabs, Inc., Ипсвич, Массачусетс, США). После подготовки библиотеки концентрацию и размер фрагментов оценивали с использованием флуориметра Qubit и анализатора Qsep400, а эффективную концентрацию библиотеки количественно определяли с помощью qRT-PCR. После прохождения контроля качества библиотеки объединяли в соответствии с их эффективными концентрациями и целевым выходом секвенирования, и секвенирование выполняли на платформе Illumina в режиме PE150 (2 × 150 п.н.). Референсный геном для этого исследования - Zhangshugang_genome.fa.gz . Количество ридов для каждого гена получали путем сравнения результатов с использованием featureCounts и статистического анализа [17]. Анализ дифференциальной экспрессии генов между разными биологическими условиями проводили с использованием DESeq2 для получения набора дифференциально экспрессируемых генов между разными биологическими условиями [18,19]. После дифференциального анализа метод Бенджамини-Хохберга использовали для коррекции вероятности проверки гипотезы (p-значение) для множественной проверки гипотез и получения частоты ложных открытий (FDR) [20]. Критериями отбора дифференциально экспрессируемых генов в этом исследовании были |log2Fold Change| ≥ 1 и FDR < 0.05.
2.6. Валидация генов-кандидатов с помощью qRT-PCR
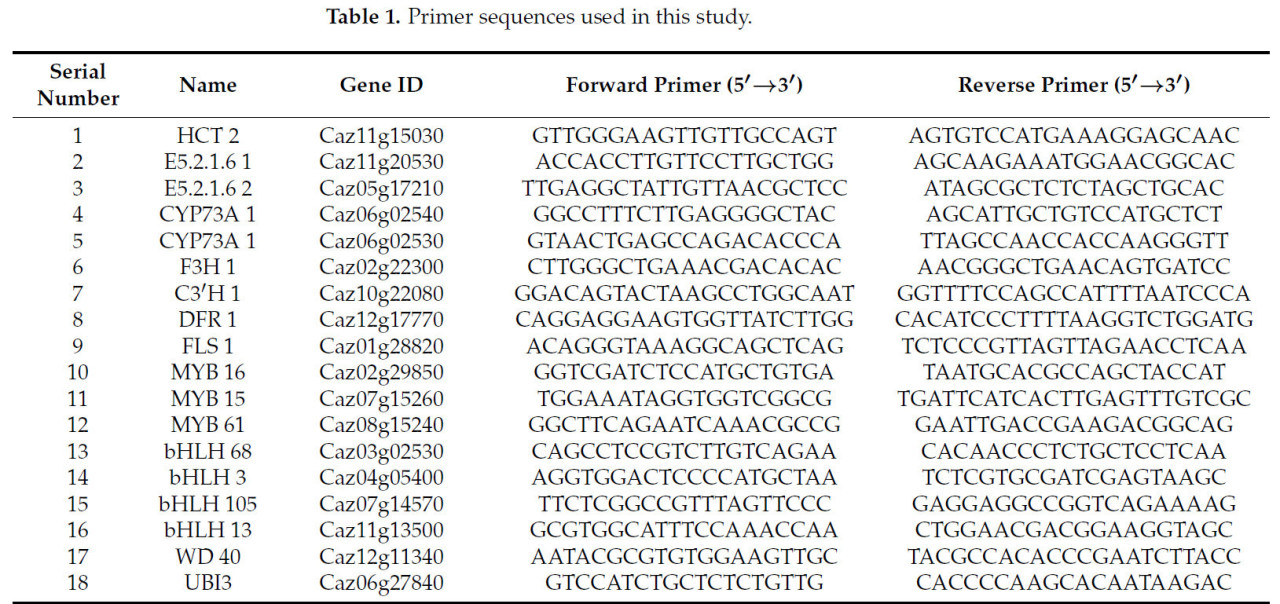
На основе комплексного транскриптомного и метаболомного анализов C. frutescens мы получили в общей сложности 8928 дифференциально экспрессируемых генов, среди которых мы идентифицировали 23 гена, тесно связанных с флавоноидами, включая CHS 1, CHS 2, HCT 2, HCT 3, E5.2.1.6 1, E5.2.1.6 2, CYP73A 1, CYP73A 2, F3H 1, C3H 1, DFR 1, DFR 2, FLS 1, FLS 2 и CYP75B1 1, вместе с транскрипционными факторами MYB 15, MYB 16, MYB 61, bHLH 68, bHLH 3, bHLH 105, bHLH 13 и WD 40. Технология qRT-PCR была использована для анализа паттернов экспрессии вышеупомянутых генов. Общую РНК экстрагировали из тканей кожицы на разных стадиях развития с использованием набора FastPure® Plant Total RNA Isolation Kit (Vazyme Biotech Co., Ltd., Нанкин, Китай) и обратно транскрибировали в кДНК с использованием набора All-In-One 5X RT MasterMix (Applied Biological Materials Inc., Шанхай, Китай). Инструмент Primer-BLAST из базы данных NCBI использовали для создания праймеров для qRT-PCR (Таблица 1). Относительную экспрессию гена рассчитывали с использованием метода 2−ΔΔCt с геном UBI в качестве внутреннего контроля [21].
3. Результаты
3.1. Анализ метаболомных данных околоплодника C. frutescens в разные основные периоды роста
3.1.1. PCA анализ метаболомных данных кожицы C. frutescens на разных стадиях роста
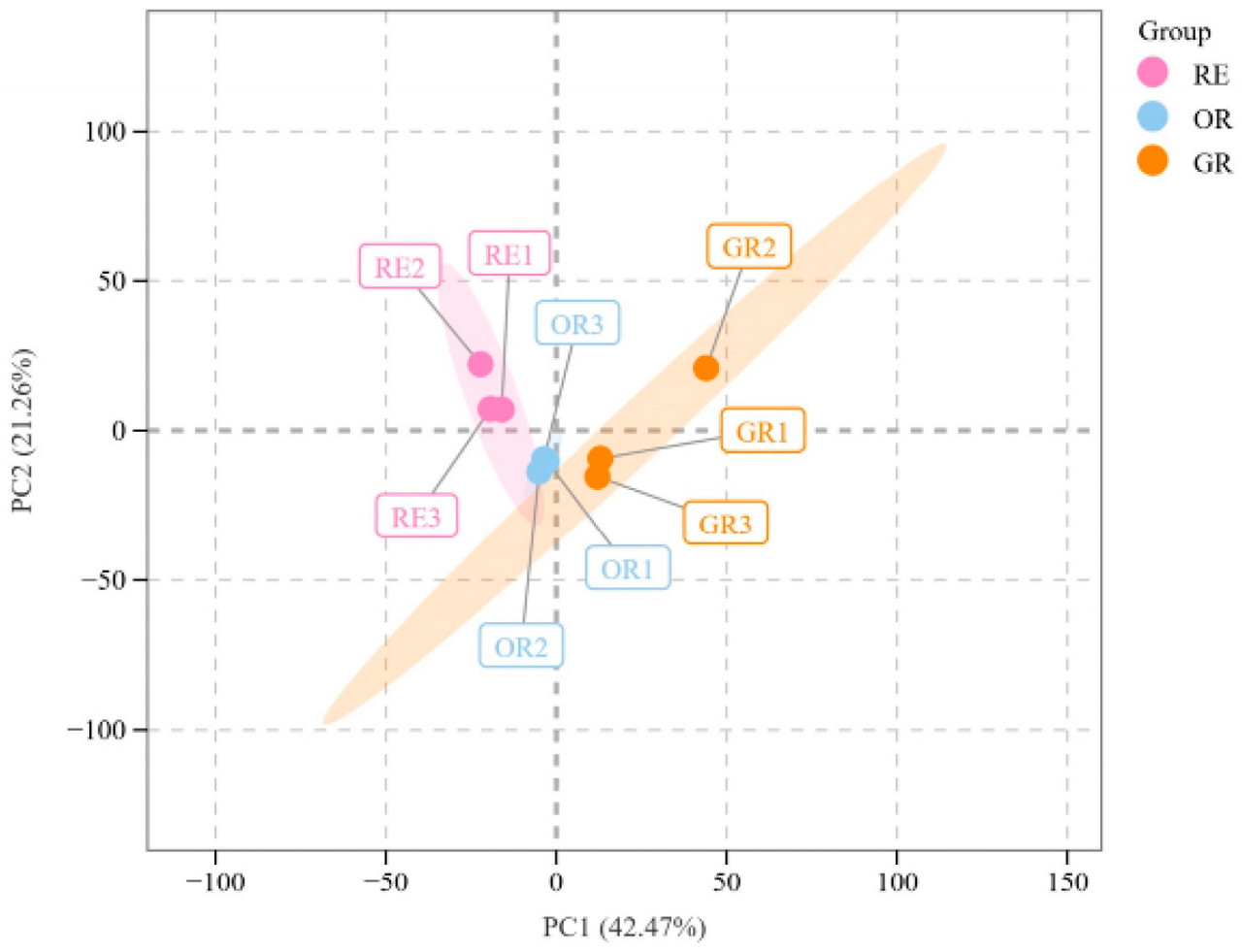
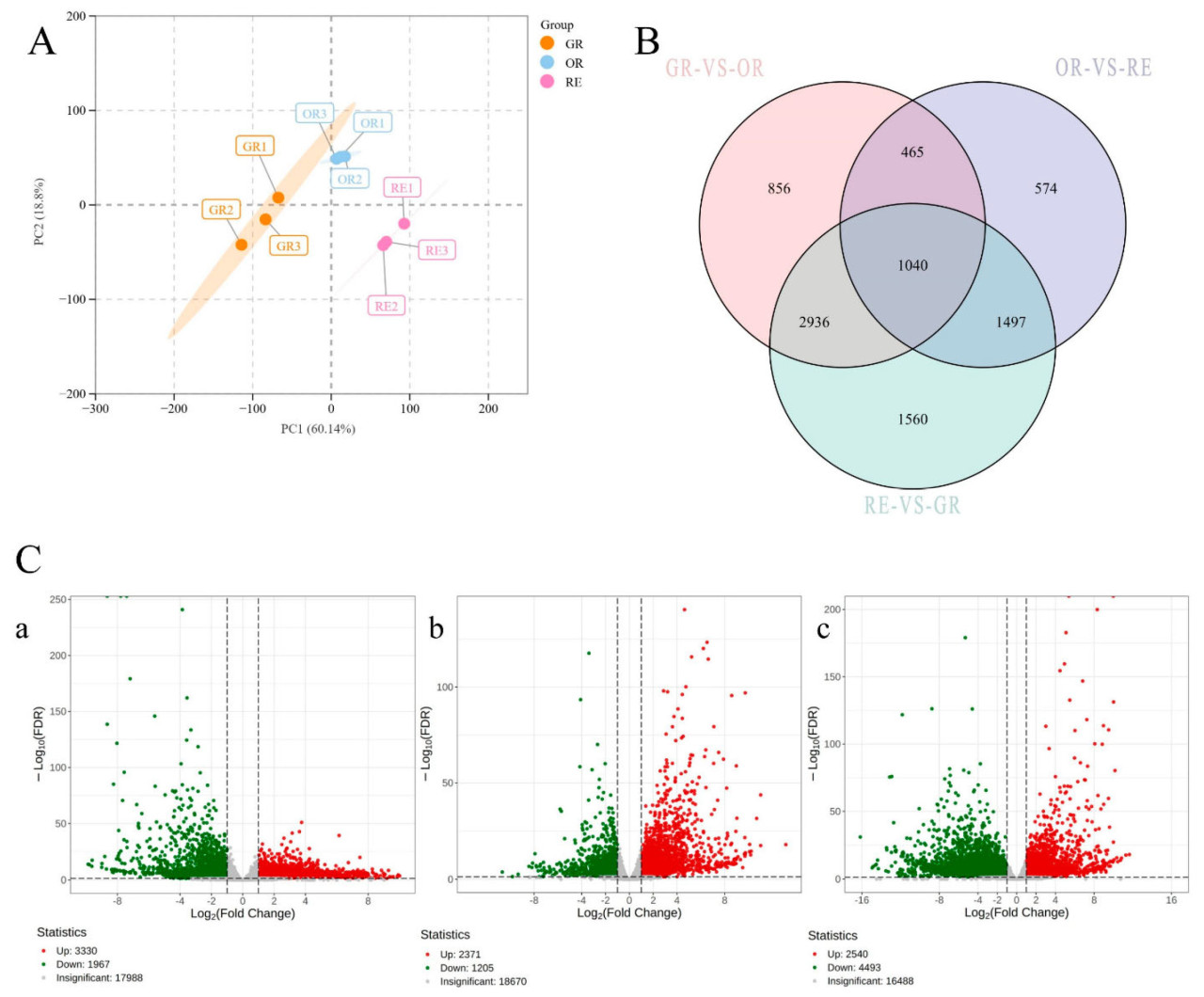
Был проведен PCA для оценки различий между группами на основе метаболомных данных. Результаты PCA показали, что PC1 и PC2 составляли 42.47% и 21.26% дисперсии соответственно (Рисунок 2). Три биологические повторности для каждой стадии развития образцов кожицы C. frutescens тесно сгруппировались вместе, что указывает на хорошую воспроизводимость и надежность внутри каждой группы. Большие расстояния, наблюдаемые между метаболитными профилями разных стадий роста, отражают отчетливые метаболические изменения, происходящие во время развития плода.
Рисунок 2. Метаболиты кожицы C. frutescens на разных стадиях развития согласно анализу главных компонент (PCA). На графике PCA GR представляет 50 дней после отцветания, OR представляет 65 дней после отцветания, а RE представляет 80 дней после отцветания.
3.1.2. Скрининг дифференциальных метаболитов в околоплоднике C. frutescens в разные периоды роста
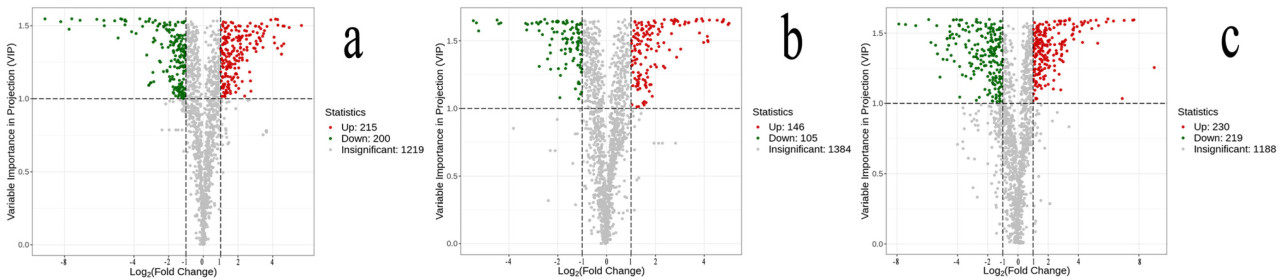
Чтобы дальнейше исследовать изменения метаболитов во время развития цвета плода у C. frutescens, UPLC-MS/MS использовали для анализа образцов кожицы с разных стадий роста. Дифференциальные метаболиты между стадиями визуализировали с использованием вулканических графиков (Рисунок 3). Всего было идентифицировано 415 дифференциальных метаболитов между незрелой стадией (GR) и полуспелой стадией (OR), из которых 215 были повышены, а 200 понижены. Между полуспелой стадией (OR) и зрелой стадией (RE) было обнаружено 251 дифференциальных метаболитов, из которых 146 были повышены и 105 понижены. Наконец, между зрелой стадией (RE) и незрелой стадией (GR) было идентифицировано 449 дифференциальных метаболитов, из которых 230 были повышены и 219 понижены.
Рисунок 3. Анализ вулканических диаграмм метаболитов околоплодника C. frutescens в течение разных периодов роста. На вулканических графиках ((a) GR против OR; (b) OR против RE; (c) RE против GR) каждая точка представляет метаболит. Зеленые точки указывают на пониженные метаболиты, красные точки указывают на повышенные метаболиты, а серые точки представляют метаболиты без значительной разницы. Ось x представляет log2 кратное изменение (log2FC) в относительных уровнях метаболитов между двумя группами образцов, причем большие абсолютные значения указывают на большие различия. Ось y представляет оценку VIP, где более высокие значения указывают на более значительные различия и более надежные дифференциальные метаболиты.
3.1.3. Идентификация флавоноидных соединений в околоплоднике C. frutescens в разные периоды роста
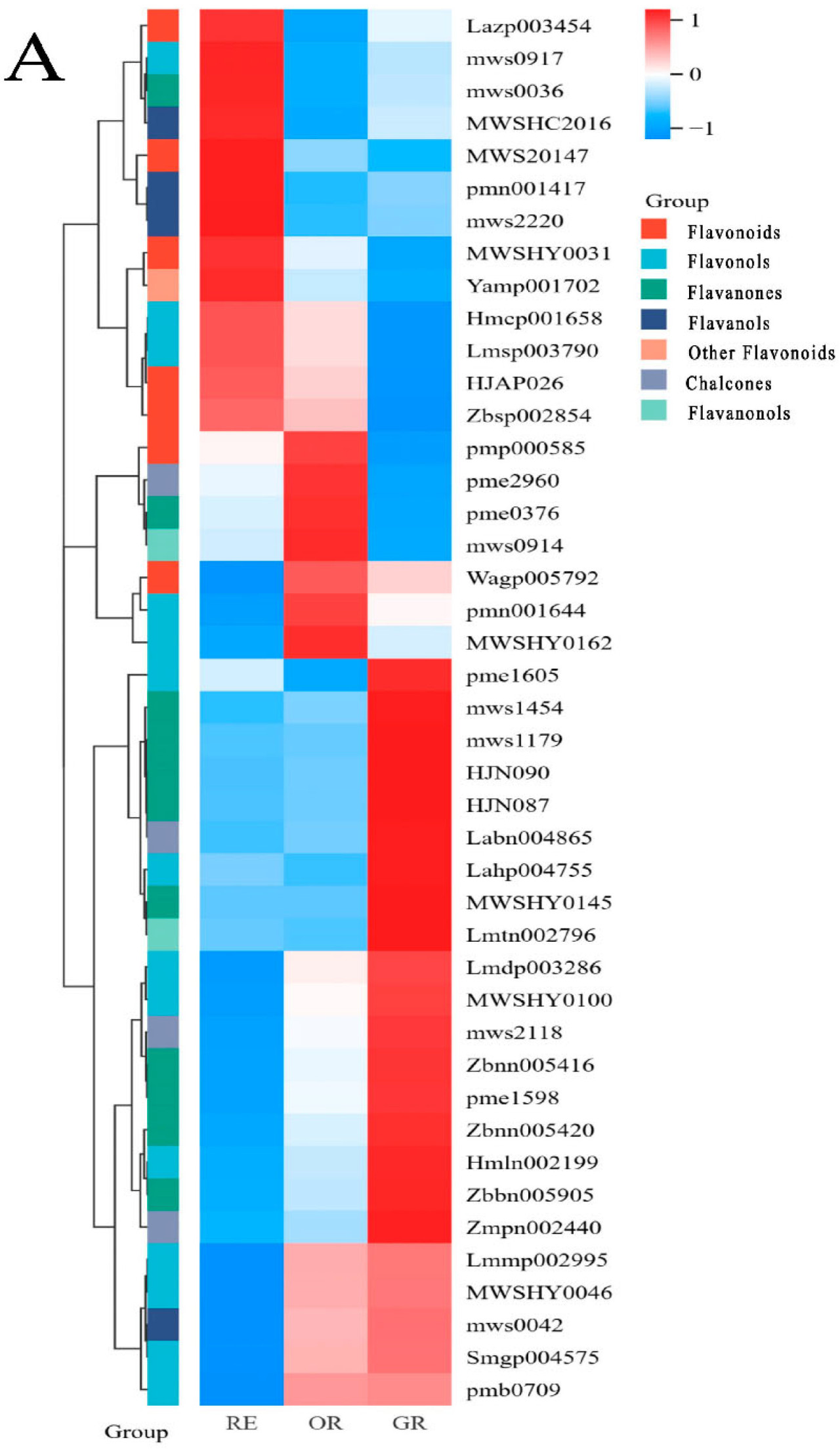
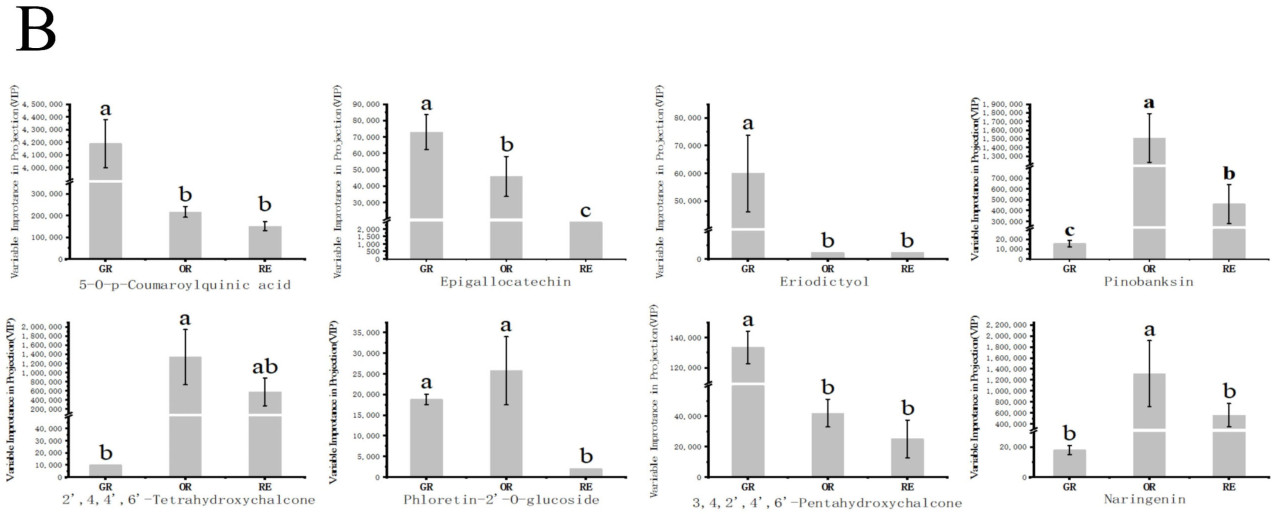
Чтобы дальнейше понять изменения флавоноидных соединений во время развития цвета плода, были проанализированы флавоноиды, обнаруженные в образцах кожицы C. frutescens на разных стадиях роста. Всего было идентифицировано 43 флавоноидных соединения, включая 14 флавонолов, 11 дигидрофлавоноидов, 7 флавонов, 4 халкона, 4 флаванола, 2 дигидрофлавонола и 1 другое флавоноидное соединение (Рисунок 4A). Среди них восемь флавоноидных метаболитов показали значительные различия, и все они демонстрировали тенденцию к снижению содержания по мере увеличения зрелости. Эриодиктиол, 5-О-п-кумароил хинная кислота и 3,4,2′,4′,6′-пентагидроксихалкон накапливались в более высоких уровнях в течение незрелой стадии (50 дней после отцветания). Пинобанксин, 2′,4,4′,6′-тетрагидроксихалкон и нарингенин были в большем избытке на полуспелой стадии (65 дней после отцветания). Эпигаллокатехин и флоретин-2′-О-глюкозид накапливались больше на незрелой и полуспелой стадиях (50 и 65 дней после отцветания), чем на зрелой стадии (80 дней после отцветания) (Рисунок 4B).
Рисунок 4. (A) Изменения в содержании флавоноидных метаболитов на разных стадиях роста C. frutescens. (B) Анализы содержания метаболитов, которые значительно меняются на разных стадиях роста (C. frutescens). На тепловой карте дифференциальные метаболиты были выбраны на основе оценки проекции важности переменных (VIP) ≥ 1 и абсолютного значения Log2 кратного изменения (|Log2FC| ≥ 1). Красный цвет указывает на повышенные уровни метаболитов, а синий указывает на пониженные уровни. Правая сторона показывает классификацию дифференциальных метаболитов. На столбчатой диаграмме ось y представляет оценку VIP, указывающую на степень вариации уровней метаболитов, а ось x представляет разные стадии роста. Значительные различия между образцами отмечены разными буквами, а полосы ошибок указывают стандартное отклонение технических повторностей.
3.2. Анализ транскриптомных данных околоплодника C. frutescens на разных стадиях роста
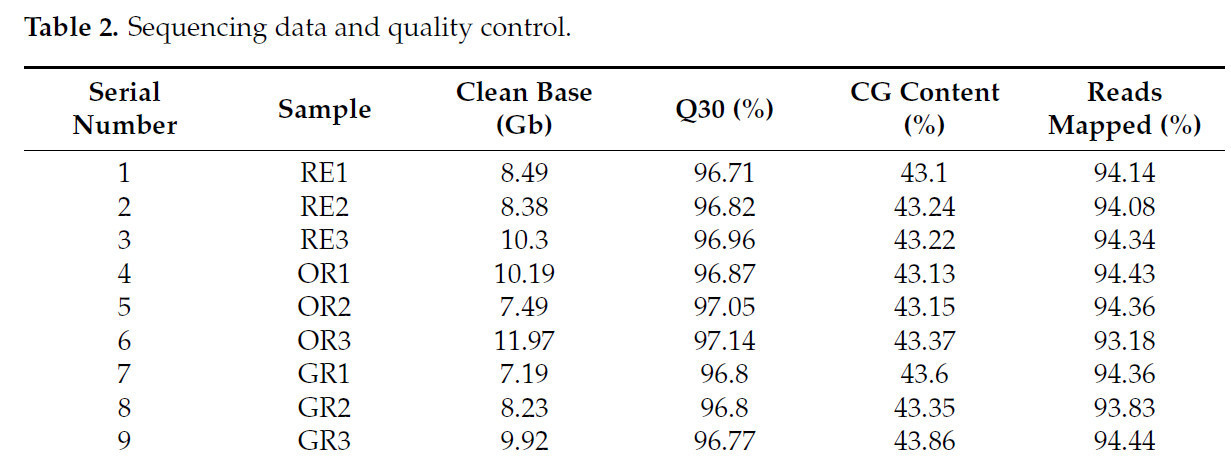
Чтобы исследовать молекулярные механизмы, лежащие в основе изменений содержания флавоноидов в кожице C. frutescens с течением времени, было проведено транскриптомное секвенирование образцов кожицы с трех стадий развития. Всего было секвенировано девять образцов плодов C. frutescens после фильтрации, предоставляя более 7 Гб чистых ридов (данные загружены в базу данных NCBI BioProject ID: PRJNA1181927; ссылка доступа: https://dataview.ncbi.nlm.nih.gov/object/PRJNA1181927?reviewer=l8g3skdieegg6f8qa8rrf6au8t; доступ осуществлен 5 ноября 2024). Эти чистые риды были сопоставлены с референсным геномом (Zhangshugang_genome.fa.gz), с процентом картирования более 93%, значением Q30 выше 96% и содержанием GC в диапазоне от 43.1% до 43.86%. Биологические повторности показали высокие коэффициенты корреляции (больше 0.8), что удовлетворяет требованиям для последующего анализа (Таблица 2). DESeq2 использовали для выполнения анализа дифференциальной экспрессии между тремя группами образцов околоплодника C. frutescens.
Таблица 2. Данные секвенирования и контроль качества.
PCA анализ дифференциально экспрессируемых генов (DEG) показал, что три биологические повторности от каждой стадии развития тесно сгруппировались вместе, что указывает на высокую воспроизводимость внутри каждой группы. Большие расстояния между образцами с разных стадий отражали значительные изменения в экспрессии генов с течением времени (Рисунок 5A). Анализ транскриптома через разные стадии C. frutescens был выполнен с использованием критериев |log2Fold Change| ≥ 1 и p < 0.05, идентифицируя в общей сложности 8928 DEG. Количество DEG между группами образцов варьировалось от 3576 до 7033. В сравнении GR против OR было идентифицировано 5297 DEG (Рисунок 5C(a)), из которых 3330 были повышены и 1967 понижены. В сравнении OR против RE было идентифицировано 3576 DEG (Рисунок 5C(b)), включая 2371 повышенных и 1205 пониженных. В сравнении RE против GR было идентифицировано 7033 DEG (Рисунок 5C(c)), из которых 2540 были повышены и 4493 понижены. Сравнение RE против GR показало наибольшее количество DEG, в то время как наименьшее количество DEG было идентифицировано в сравнении OR против RE. Всего 1040 DEG были общими для всех трех сравнений (Рисунок 5B).
Рисунок 5. Анализ данных транскриптомики околоплодника перца на разных стадиях роста. (A) Анализ главных компонент транскриптомов околоплодника перца на разных стадиях роста; (B) Анализ диаграммы Венна дифференциальных генов околоплодника перца на разных стадиях роста; (C) Анализ вулканических графиков дифференциальных генов околоплодника перца на разных стадиях роста. На графике PCA GR представляет 50 дней после отцветания, OR представляет 65 дней после отцветания, а RE представляет 80 дней после отцветания, с тремя повторностями на стадию. На диаграмме Венна розовый цвет представляет GR против OR, фиолетовый представляет OR против RE, а зеленый представляет RE против GR. Неперекрывающиеся области представляют уникальные гены, специфичные для каждого сравнения, в то время как перекрывающиеся области представляют общие дифференциально экспрессируемые гены (DEG) среди сравнений. На вулканических графиках ((a) GR против OR; (b) OR против RE; (c) RE против GR) каждая точка представляет ген. Зеленые точки указывают на пониженные гены, красные точки указывают на повышенные гены, а серые точки представляют гены без значительных различий. Ось x представляет кратное изменение экспрессии гена, причем большие абсолютные значения указывают на большие различия между двумя группами образцов. Ось y представляет уровень значимости дифференциальных генов, где более высокие значения указывают на более значительные различия, что делает идентифицированные DEG более надежными.
3.3. Совместный анализ транскриптом-метаболом
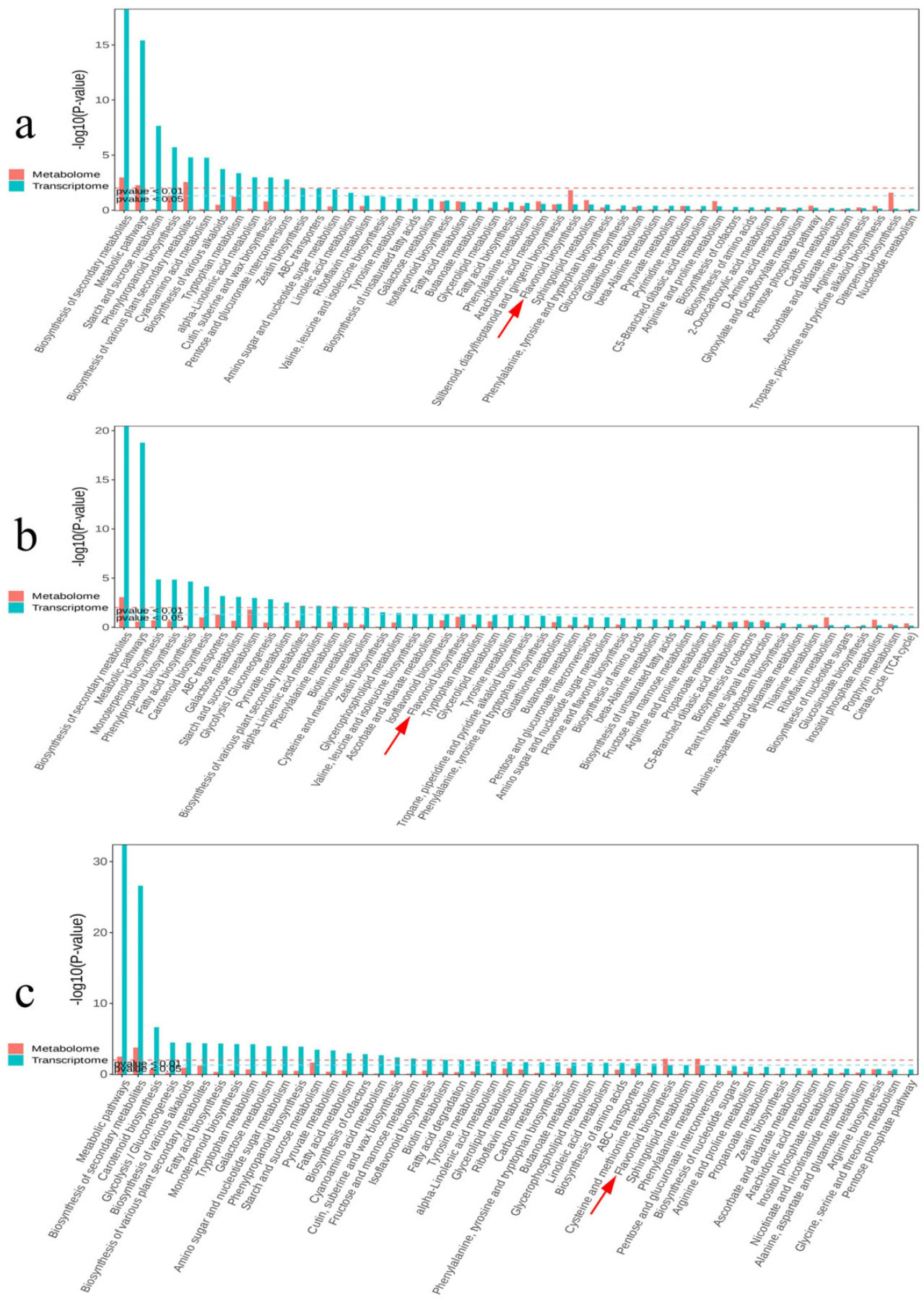
Анализ обогащения был выполнен с использованием гипергеометрических тестов для исследования генов и метаболитов в кожице C. frutescens на разных стадиях развития. Был проведен анализ обогащения путей KEGG, и тесты гипергеометрического распределения были применены к путям. Дифференциально экспрессируемые гены (DEG) были обогащены в пути биосинтеза флавоноидов во всех трех сравнениях (путь KO00941) (Рисунок 6). Основываясь на этом исследовании, разные виды C. frutescens проявляют вариации в конкретных типах и количествах флавоноидных соединений [22], что может привести к различиям в цвете плода. Поскольку дифференциальные метаболиты во всех трех сравнениях также были обогащены в пути биосинтеза флавоноидов, мы выбрали путь биосинтеза флавоноидов (путь KO00941) для дальнейшего анализа.
Рисунок 6. Совместный анализ транскриптомики и метаболомики. Столбчатая диаграмма обогащения KEGG. Ось x представляет названия путей KEGG, а ось y указывает на значимость обогащения p-значений. В графике анализа обогащения KEGG ((a) GR против OR; (b) OR против RE; (c) RE против GR) красные столбцы представляют метаболомные данные, а зеленые столбцы представляют транскриптомные данные.
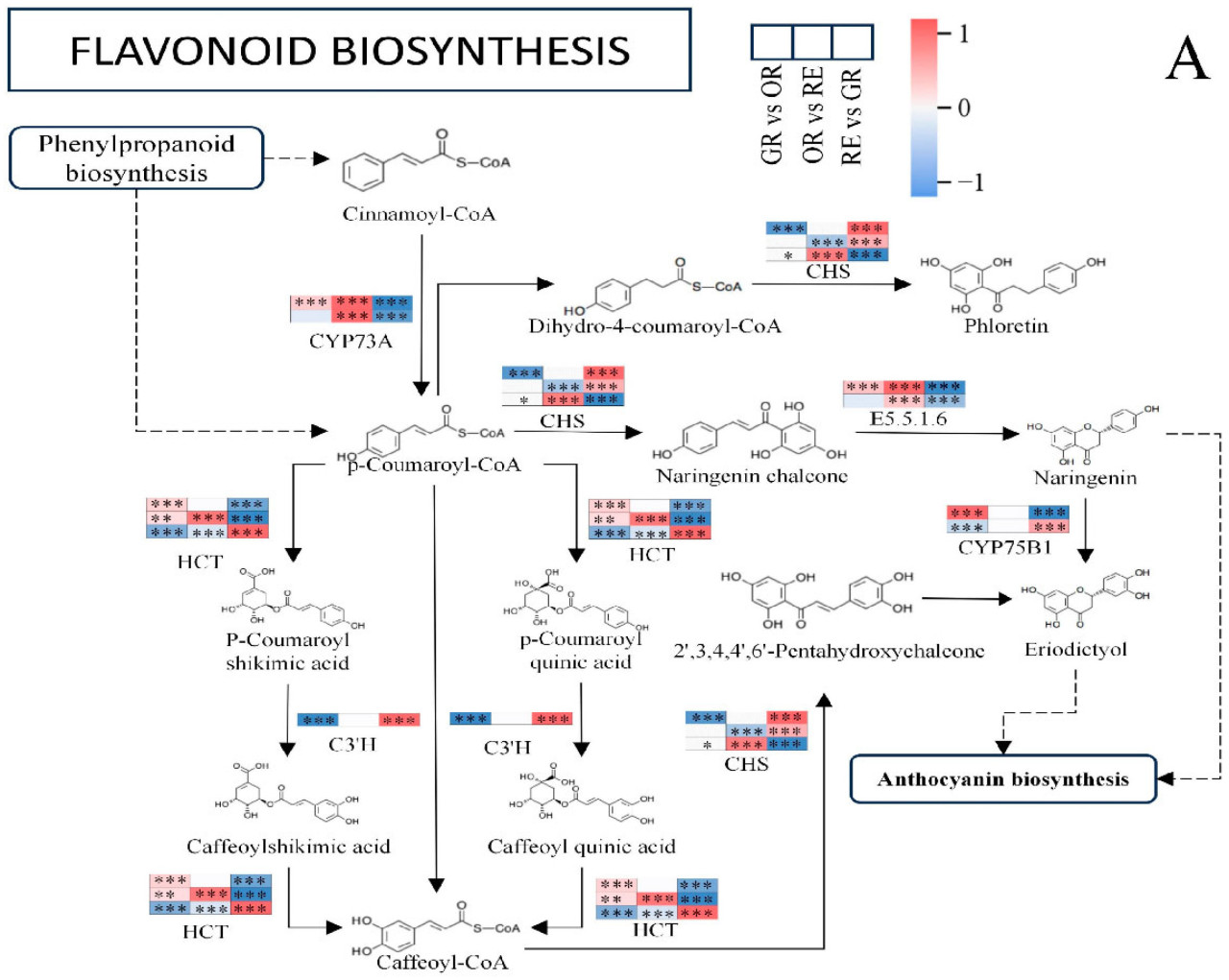
Был проведен сравнительный анализ 20 наиболее обогащенных путей KEGG для DEG через три стадии развития C. frutescens. Интегрируя метаболомные и транскриптомные данные C. frutescens, мы построили путь биосинтеза флавоноидов для плода (Рисунок 7A). На основе анализа обогащения KEGG были идентифицированы 28 ключевых генов, участвующих в биосинтезе флавоноидов (Рисунок 7C), включая 13 HCT, 3 E2.1.1.104, 2 C3’H, 2 CYP73A, 2 E5.5.1.6, 2 DFR, 1 ANS, 1 CHS, 1 CYP75B1 и 1 FLS генов. Большинство этих генов были высоко экспрессированы в незрелом плоде (50 дней после отцветания), в то время как DFR1 и CYP75B1 показали более высокую экспрессию в полуспелом плоде (65 дней после отцветания). Только ANS1 был высоко экспрессирован в зрелом плоде (80 дней после отцветания). Был выполнен корреляционный анализ Пирсона для оценки корреляции между восемью значительно различающимися флавоноидными метаболитами и уровнями экспрессии 28 DEG в пути биосинтеза флавоноидов (Рисунок 7B). Результаты выявили 111 значительных положительных корреляций и восемь значительных отрицательных корреляций. Анализ показал, что гены ANS, C3’H, CHS, CYP75B1, DFR, E2.1.1.104, FLS и HCT были значительно скоррелированы с идентифицированными флавоноидными соединениями, показывающими дифференциальную экспрессию.
Рисунок 7. (A) Анализ пути биосинтеза флавоноидов, визуализированный с использованием тепловой карты на основе log2 кратных изменений. Звездочки указывают на уровень значимости, где «» p < 0.05, «» p < 0.01 и «» p < 0.001. Красный цвет указывает на увеличение кратного изменения гена, а синий указывает на уменьшение. Каждая строка данных представляет транскрипт. (B) Корреляционный анализ между генами биосинтеза флавоноидов и значительно различающимися флавоноидными метаболитами. Ось y показывает гены, кодирующие флавоноидные соединения, а ось x показывает значительно различающиеся флавоноидные метаболиты. Звездочки указывают на уровень значимости, где «» p < 0.05, «» p < 0.01 и «» p < 0.001. Красный цвет указывает на положительные корреляции, а синий указывает на отрицательные корреляции. (C) Анализ экспрессии генов биосинтеза флавоноидов, с использованием средних значений FPKM из трех биологических повторностей. Ось x представляет разные стадии развития (GR: 50 дней после отцветания, OR: 65 дней после отцветания, RE: 80 дней после отцветания), а ось y показывает названия генов.
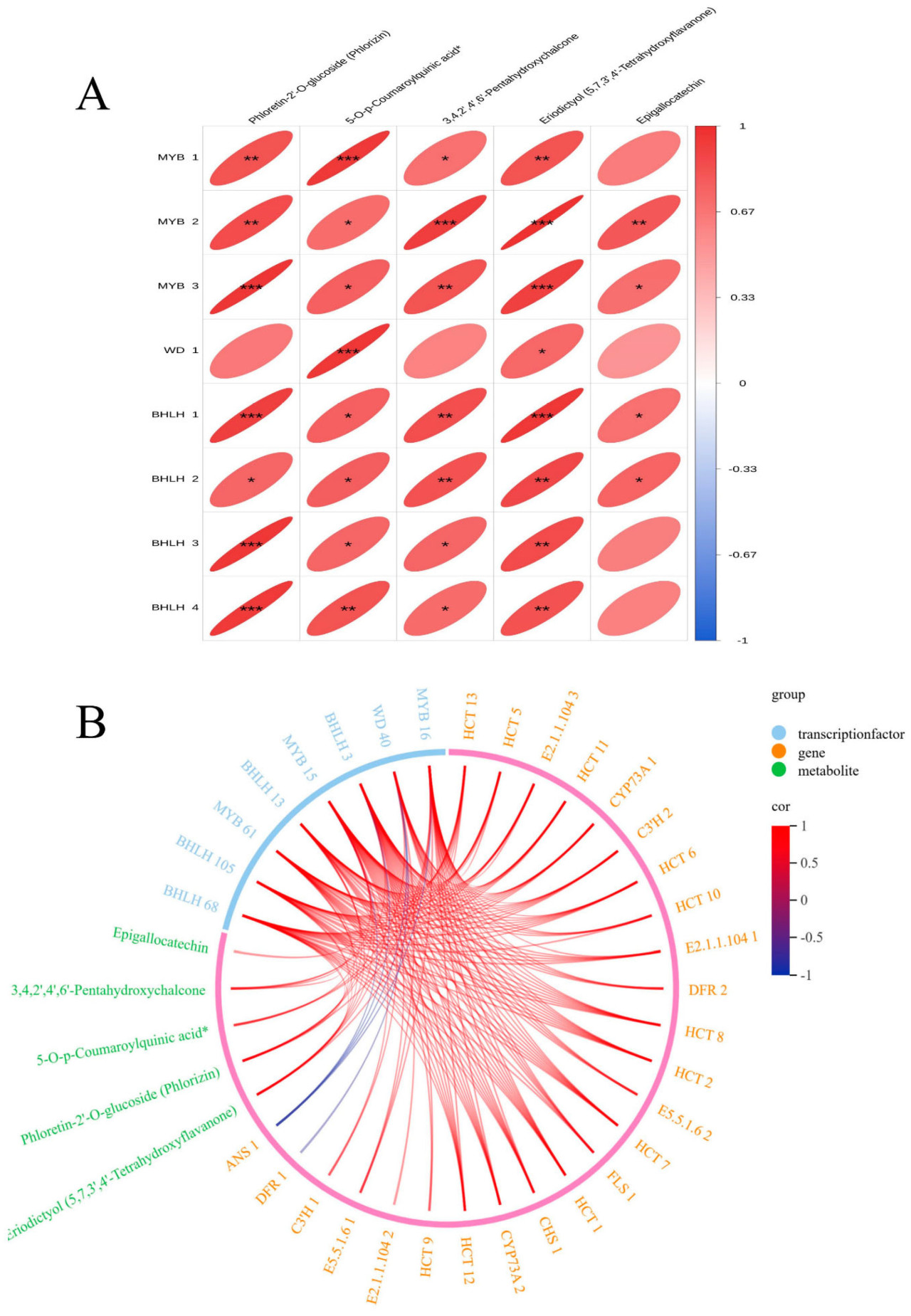
Основываясь на предыдущих исследованиях, предполагающих участие MBW комплекса в биосинтезе флавоноидов [9,10], мы ранжировали 100 наиболее дифференциально экспрессируемых генов по кратному изменению (|Log2FC|) между незрелой стадией (50 дней после отцветания) и зрелой стадией (80 дней после отцветания), в течение которых произошли наиболее значительные изменения цвета. Из них было идентифицировано восемь транскрипционных факторов, из которых три принадлежат к семейству MYB, четыре принадлежат к семейству bHLH и один принадлежит к семейству WD. Корреляционный анализ между этими транскрипционными факторами и значительно различающимися флавоноидными метаболитами выявил сильную корреляцию (Рисунок 8A). Дополнительно, корреляционный анализ между этими восемью транскрипционными факторами и 28 ключевыми генами, участвующими в биосинтезе флавоноидов, показал, что за исключением отрицательной корреляции между ANS1 и DFR1, все другие транскрипционные факторы демонстрировали положительные корреляции (Рисунок 8B).
Рисунок 8. (A) Корреляционный анализ между транскрипционными факторами и значительно различающимися флавоноидными метаболитами на основе корреляции Пирсона. Ось y представляет названия транскрипционных факторов, а ось x показывает значительно различающиеся флавоноидные метаболиты. Звездочки указывают на уровень значимости, где «» p < 0.05, «» p < 0.01 и «» p < 0.001. (B) Корреляционный анализ между транскрипционными факторами и генами, кодирующими биосинтез флавоноидов. Синий представляет транскрипционные факторы, зеленый представляет значительно различающиеся флавоноидные метаболиты, а оранжевый представляет гены биосинтеза флавоноидов. Красные линии указывают на положительные корреляции, а синие линии указывают на отрицательные корреляции.
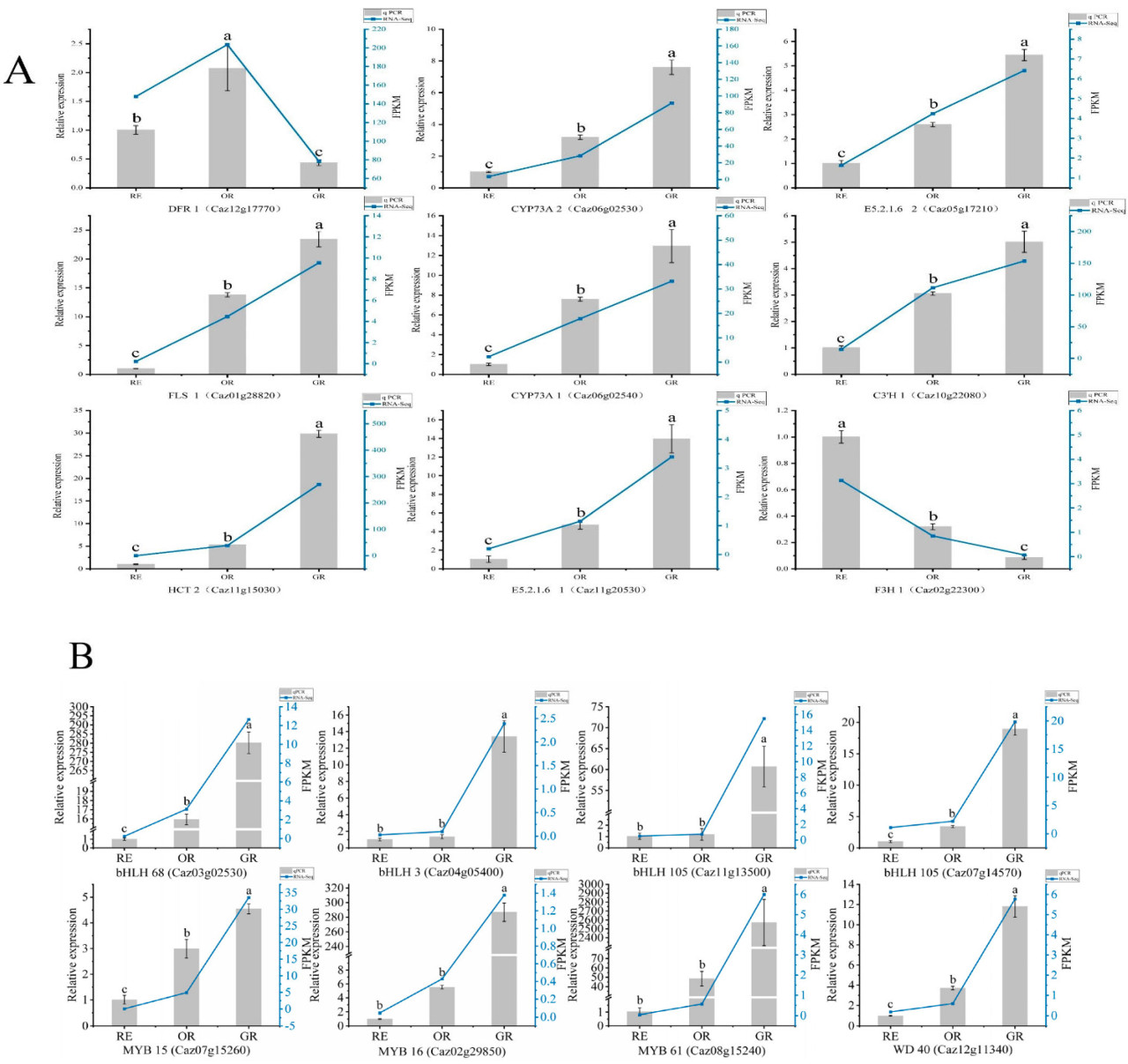
Чтобы дальнейше понять биосинтез флавоноидов, мы случайным образом выбрали девять основных генов, участвующих в биосинтезе флавоноидов, и восемь потенциально связанных генов для анализа qRT-PCR, чтобы оценить их изменения экспрессии через разные стадии развития плода. Этот анализ также служил для проверки транскриптомных данных. Результаты qRT-PCR показали, что тенденции экспрессии девяти выбранных генов были согласованы со значениями FPKM из транскриптомного анализа, дальнейше подтверждая точность и надежность транскриптомных данных (Рисунок 9A). Дополнительно, общее снижение относительных уровней экспрессии по мере созревания плода перца наблюдалось в результатах qRT-PCR для транскрипционных факторов (Рисунок 9B).
Рисунок 9. (A) Анализ тенденций, сравнивающий данные транскриптомики и результаты qRT-PCR. Ось x представляет разные стадии развития (GR: 50 дней после отцветания, OR: 65 дней после отцветания, RE: 80 дней после отцветания). Левая ось y (черная) представляет относительную экспрессию, а правая ось y (синяя) представляет значения FPKM. Полосы ошибок представляют ошибки технических повторностей. Значимость qPCR указана изменением строчных букв. (B) Анализ паттернов экспрессии транскрипционных факторов. Ось x представляет разные стадии развития (GR: 50 дней после отцветания, OR: 65 дней после отцветания, RE: 80 дней после отцветания), левая ось y (черная) представляет относительную экспрессию, а правая ось y (синяя) представляет значения FPKM. Полосы ошибок представляют ошибки технических повторностей. Значимость qPCR указана изменением строчных букв.
4. Обсуждение
Цвет плода часто считался важным признаком качества [23], причем исследования показывали, что изменения в накоплении флавоноидов в растениях часто приводят к изменениям цвета плода [24]. Флавоноиды были ключевыми вторичными метаболитами, широко распространенными в растениях, включая овощи и фрукты [25,26,27]. Биосинтез флавоноидов давно является фокусом исследований физиологии растений, поскольку флавоноиды играют важную роль в нескольких физиологических процессах растений [28]. Исследования на видах пасленовых продемонстрировали сильную корреляцию между накоплением флавоноидов и переходом цвета плода [24,29,30]. В недавнем исследовании хризантемы (сорт «Fencui») также сообщалось, что CmMYB012 может регулировать CmFNS, чтобы ингибировать биосинтез флавоноидов, что в конечном итоге влияет на цвет цветка растения [31]. В этом исследовании мы проанализировали плод C. frutescens на разных стадиях развития, используя метаболомные и транскриптомные данные. Наши результаты показали, что флавоноидные соединения и гены, кодирующие биосинтез флавоноидов, меняются по мере развития плода. Анализ обогащения KEGG данных транскриптомики и метаболомики показал, что оба были обогащены в пути метаболизма флавоноидов; основываясь на предыдущих исследованиях, мы предположили, что флавоноидные соединения в кожице со временем деградируют, что приводит к переходу цвета плода от зеленого к красному.
Метаболомный анализ идентифицировал 43 флавоноидных метаболита в кожице C. frutescens, состоящих из семи подкатегорий, что указывает на богатое присутствие флавоноидных соединений в кожице C. frutescens. Среди обнаруженных флавоноидов значительные изменения наблюдались в флавонолах и дигидрофлавоноидах во время развития плода. В частности, 3,5,7-тригидроксифлаванон (пинобанксин), халкон нарингенина, нарингенин, флоризин, 5-О-п-кумароилхинная кислота, 3,4,2′,4′,6′-пентагидроксихалкон, эриодиктиол и эпигаллокатехин проявляли наиболее выраженные различия. В растениях флавоноиды накапливались различными способами и были ключевыми биомолекулами, влияющими на пигментацию, часто встречаясь в эпидермальных тканях [32,33]. В этом исследовании значительно различающиеся флавоноидные соединения показали тенденцию к снижению содержания по мере созревания плода, что позволяет предположить, что флавоноиды могут постепенно разлагаться на другие вещества, способствуя изменениям цвета во время развития плода. Это открытие согласуется с результатами исследования Allwood и др. [34] на смородине черной (Ribes nigrum). Были четкие различия как в типе, так и в содержании метаболитов на разных стадиях развития C. frutescens, причем наиболее заметные изменения происходили, когда плод переходил от оранжевой стадии (65 дней после отцветания) к красной стадии (80 дней после отцветания). Это наблюдение было согласовано с выводами Heng и др. [35], которые сообщили о значительных различиях в содержании метаболитов у C. frutescens между полуспелой и зрелой стадиями. Основываясь на этих результатах, мы предположили, что распад и трансформация этих метаболитов в основном происходят во время перехода от полуспелой к зрелой стадиям.
Анализ RNA-Seq идентифицировал 28 ключевых генов, участвующих в основном пути биосинтеза флавоноидов. Эти гены были похожи на гены биосинтеза флавоноидов, участвующие в окраске кожицы у дыни [36], хотя 4CL, IFS и UFGT не показали значительной дифференциальной экспрессии в этом исследовании, что, как мы предположили, может быть связано с межвидовыми различиями. CHS был важнейшим ферментом в синтезе флавоноидных соединений в растениях и играл ключевую роль в производстве производных флавоноидов и поддержке роста и развития растений [37,38,39]. В нашем исследовании п-кумароил-КоА катализировался CHS с образованием халкона нарингенина. Аналогично, другое исследование обнаружило значительное увеличение содержания антоцианов во время созревания перца, что было связано с переходом цвета от зеленого к красному [3]. Их исследование подчеркнуло повышающую регуляцию генов, участвующих в биосинтезе антоцианов, таких как CHS и DFR, подчеркивая роль флавоноидов в окраске перца на более поздних стадиях развития плода. Исследования на Marchantia polymorpha показали, что PabHLH1 может катализировать синтез флавоноидов [40], в то время как CmMYB012 в хризантеме негативно регулирует флавоноиды [31]. Исследования на люцерне также упоминали, что MtWD40 может регулировать производство антоцианов [41]. В этом исследовании мы отобрали в общей сложности восемь генов MBW комплекса (MYB–bHLH–WD repeat) по коэффициенту корреляции Пирсона и проанализировали их паттерны экспрессии. Мы обнаружили, что все восемь генов показали высокую экспрессию во время раннего роста растений и снижение экспрессии по мере роста растений. Корреляционный анализ восьми отобранных генов и флавоноидных метаболитов с значительными различиями обнаружил, что все они были положительно скоррелированы. Мы предполагаем, что регуляция биосинтеза флавоноидов может быть вовлечена в эти восемь генов.
Дополнительно, исследования на других плодоносящих растениях, таких как виноград (Vitis vinifera) и голубика (Vaccinium spp.), показали сходные паттерны накопления флавоноидов. В винограде содержание антоцианов значительно увеличивалось с созреванием плода и приводило к сдвигу цвета от красного к фиолетовому [42]. Аналогично, это исследование сообщило, что рост уровней антоцианов заставлял голубику переходить от зеленого к темно-синему, с содержанием антоцианов, достигающим пика при полной зрелости плода [43]. Однако при сравнении наших выводов с другими исследованиями по C. frutescens возникают определенные различия. В частности, конкретные типы и количества флавоноидных соединений варьировались через разные виды перца, потенциально приводя к отчетливому цвету плода [22]. Это предполагает, что, хотя общая тенденция увеличения содержания флавоноидов, связанная с созреванием плода, была сохранена через C. frutescens, конкретные изменения в составе флавоноидов и их эффекты на цвет плода могут варьироваться между разными сортами перца.
Хотя это исследование предоставило ценные идеи, следует признать несколько ограничений. Во-первых, наше исследование было сосредоточено на одном сорте C. frutescens. Учитывая генетическое разнообразие внутри рода Capsicum, повторение этого исследования на разных сортах перца помогло бы определить, являются ли наблюдаемые паттерны накопления флавоноидов и изменений цвета последовательными. Это помогло бы идентифицировать любые сортовые различия и расширить наше понимание флавоноидных соединений в C. frutescens. С другой стороны, это исследование было проведено в контролируемых условиях окружающей среды. Биосинтез флавоноидов и пигментация плода могут значительно зависеть от таких факторов, как интенсивность света, температура и почвенные питательные вещества [44]. Будущие исследования должны изучить влияние разных условий окружающей среды на накопление флавоноидов и окраску плода у C. frutescens. Такие исследования могли бы предоставить идеи о том, как экологические стрессоры регулируют пути флавоноидов и влияют на качество плода. Дополнительно, наше исследование не изучало потенциальные взаимодействия между флавоноидами и другими метаболитами, которые могут влиять на цвет плода. Метаболиты, такие как каротиноиды и хлорофилл, также влияют на цвет плода и могут взаимодействовать с флавоноидами сложными способами [45]. Наконец, питательные и оздоровительные преимущества флавоноидов были хорошо установлены, с многочисленными исследованиями, подчеркивающими их антиоксидантные, противовоспалительные и противораковые свойства [46]. Понимание изменений в содержании флавоноидов в плодах перца могло бы направлять усилия по улучшению их питательной и экономической ценности.
5. Выводы
В течение развития плода C. frutescens от незрелых до зрелых стадий мы идентифицировали 28 ключевых генов, участвующих в биосинтезе флавоноидов, и 8 дополнительных генов с потенциальными регуляторными ролями. Также были идентифицированы восемь флавоноидных соединений, которые проявляли значительные изменения во время роста плода. За исключением отрицательной корреляции между ANS1 и DFR1, все другие генно-метаболитные корреляции были положительными, указывая на их важные роли в процессе созревания плода C. frutescens. Дополнительно, восемь отобранных транскрипционных факторов все проявляли высокие уровни экспрессии во время ранней стадии роста растения. В сочетании с изменяющимися тенденциями флавоноидных метаболитов, мы предполагаем, что эти транскрипционные факторы могут быть вовлечены в регуляцию биосинтеза флавоноидных соединений и что изменения в содержании флавоноидных соединений в теле растения привели к изменениям в цвете околоплодника плода C. frutescens. Эти находки способствовали более широкому пониманию биосинтеза флавоноидов и его динамических изменений в C. frutescens.
Ссылки
1. Timberlake, C.F. Plant pigments for colouring food. Nutr. Bull. 1989, 14, 113–125. [Google Scholar] [CrossRef]
2. Facteau, T.; Chestnut, N.; Rowe, K. Relationship between fruit weight, firmness, and leaf/fruit ratio in Lambert and Bing sweet cherries. Can. J. Plant Sci. 1983, 63, 763–765. [Google Scholar] [CrossRef]
3. Lightbourn, G.J.; Griesbach, R.J.; Novotny, J.A.; Clevidence, B.A.; Rao, D.D.; Stommel, J.R. Effects of anthocyanin and carotenoid combinations on foliage and immature fruit color of Capsicum annuum L. J. Hered. 2008, 99, 105–111. [Google Scholar] [CrossRef] [PubMed]
4. Ferrer, J.-L.; Austin, M.; Stewart Jr, C.; Noel, J. Structure and function of enzymes involved in the biosynthesis of phenylpropanoids. Plant Physiol. Biochem. 2008, 46, 356–370. [Google Scholar] [CrossRef]
5. Chen, W.; Gao, Y.; Xie, W.; Gong, L.; Lu, K.; Wang, W.; Li, Y.; Liu, X.; Zhang, H.; Dong, H. Genome-wide association analyses provide genetic and biochemical insights into natural variation in rice metabolism. Nat. Genet. 2014, 46, 714–721. [Google Scholar] [CrossRef]
6. Cheng, A.X.; Han, X.J.; Wu, Y.F.; Lou, H.X. The function and catalysis of 2-oxoglutarate-dependent oxygenases involved in plant flavonoid biosynthesis. Int. J. Mol. Sci. 2014, 15, 1080–1095. [Google Scholar] [CrossRef] [PubMed]
7. Wang, D.; Wang, K.; Sun, S.; Yan, P.; Lu, X.; Liu, Z.; Li, Q.; Li, L.; Gao, Y.; Liu, J. Transcriptome and Metabolome Analysis Reveals Salt-Tolerance Pathways in the Leaves and Roots of ZM-4 (Malus zumi) in the Early Stages of Salt Stress. Int. J. Mol. Sci. 2023, 24, 3638. [Google Scholar] [CrossRef] [PubMed]
8. Forkmann, G. Flavonoids as Flower Pigments; The Formation of the Natural Spectrum and its Extension by Genetic Engineering. Plant Breed. 1991, 106, 1–26. [Google Scholar] [CrossRef]
9. Koes, R.; Verweij, W.; Quattrocchio, F. Flavonoids: A colorful model for the regulation and evolution of biochemical pathways. Trends Plant Sci. 2005, 10, 236–242. [Google Scholar] [CrossRef] [PubMed]
10. Dubos, C.; Stracke, R.; Grotewold, E.; Weisshaar, B.; Martin, C.; Lepiniec, L. MYB transcription factors in Arabidopsis. Trends Plant Sci. 2010, 15, 573–581. [Google Scholar] [CrossRef] [PubMed]
11. Liu, Y.; Lv, J.; Liu, Z.; Wang, J.; Yang, B.; Chen, W.; Ou, L.; Dai, X.; Zhang, Z.; Zou, X. Integrative analysis of metabolome and transcriptome reveals the mechanism of color formation in pepper fruit (Capsicum annuum L.). Food Chem. 2020, 306, 125629. [Google Scholar] [CrossRef] [PubMed]
12. Tang, B.; Li, L.; Hu, Z.; Chen, Y.; Tan, T.; Jia, Y.; Xie, Q.; Chen, G. Anthocyanin accumulation and transcriptional regulation of anthocyanin biosynthesis in purple pepper. J. Agric. Food Chem. 2020, 68, 12152–12163. [Google Scholar] [CrossRef] [PubMed]
13. Howard, L.R.; Talcott, S.T.; Brenes, C.H.; Villalon, B. Changes in phytochemical and antioxidant activity of selected pepper cultivars (Capsicum species) as influenced by maturity. J. Agric. Food Chem. 2000, 48, 1713–1720. [Google Scholar] [CrossRef] [PubMed]
14. Dozmorov, M.G. Epigenomic annotation-based interpretation of genomic data: From enrichment analysis to machine learning. Bioinformatics 2017, 33, 3323–3330. [Google Scholar] [CrossRef] [PubMed][Green Version]
15. Shu, P.; Zhang, Z.; Wu, Y.; Chen, Y.; Li, K.; Deng, H.; Zhang, J.; Zhang, X.; Wang, J.; Liu, Z. A comprehensive metabolic map reveals major quality regulations in red-flesh kiwifruit (Actinidia chinensis). New Phytol. 2023, 238, 2064–2079. [Google Scholar] [CrossRef] [PubMed]
16. Kanehisa, M.; Furumichi, M.; Sato, Y.; Kawashima, M.; Ishiguro-Watanabe, M. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 2023, 51, D587–D592. [Google Scholar] [CrossRef] [PubMed]
17. Liao, Y.; Smyth, G.K.; Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics 2014, 30, 923–930. [Google Scholar] [CrossRef]
18. Love, M.I.; Huber, W.; Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq. Genome Biol. 2014, 15, 1–21. [Google Scholar] [CrossRef] [PubMed]
19. Varet, H.; Brillet-Guéguen, L.; Coppée, J.-Y.; Dillies, M.-A. SARTools: A DESeq2-and EdgeR-based R pipeline for comprehensive differential analysis of RNA-Seq data. PLoS ONE 2016, 11, e0157022. [Google Scholar] [CrossRef] [PubMed]
20. Yang, L.; Wang, P.; Chen, J. 2dGBH: Two-dimensional group Benjamini–Hochberg procedure for false discovery rate control in two-way multiple testing of genomic data. Bioinformatics 2024, 40, btae035. [Google Scholar] [CrossRef]
21. Livak, K.J.; Schmittgen, T.D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCT method. Methods 2001, 25, 402–408. [Google Scholar] [CrossRef]
22. Wahyuni, Y.; Ballester, A.R.; Sudarmonowati, E.; Bino, R.J.; Bovy, A.G. Metabolite biodiversity in pepper (Capsicum) fruits of thirty-two diverse accessions: Variation in health-related compounds and implications for breeding. Phytochemistry 2011, 72, 1358–1370. [Google Scholar] [CrossRef] [PubMed]
23. Bashir, M.A.; Alvi, A.M.; Khan, K.A.; Rehmani, M.I.A.; Ansari, M.J.; Atta, S.; Ghramh, H.A.; Batool, T.; Tariq, M. Role of pollination in yield and physicochemical properties of tomatoes (Lycopersicon esculentum). Saudi J. Biol. Sci. 2018, 25, 1291–1297. [Google Scholar] [CrossRef] [PubMed]
24. Kang, S.I.; Hwang, I.; Goswami, G.; Jung, H.J.; Nath, U.K.; Yoo, H.J.; Lee, J.M.; Nou, I.S. Molecular Insights Reveal Psy1, SGR, and SlMYB12 Genes are Associated with Diverse Fruit Color Pigments in Tomato (Solanum lycopersicum L.). Molecules 2017, 22, 2180. [Google Scholar] [CrossRef] [PubMed]
25. Wang, H.J.; Pao, L.H.; Hsiong, C.H.; Shih, T.Y.; Lee, M.S.; Hu, O.Y. Dietary flavonoids modulate CYP2C to improve drug oral bioavailability and their qualitative/quantitative structure-activity relationship. Aaps J. 2014, 16, 258–268. [Google Scholar] [CrossRef] [PubMed]
26. Khoo, B.Y.; Chua, S.L.; Balaram, P. Apoptotic effects of chrysin in human cancer cell lines. Int. J. Mol. Sci. 2010, 11, 2188–2199. [Google Scholar] [CrossRef] [PubMed]
27. Liu, J.; Li, C.; Ding, G.; Quan, W. Artificial intelligence assisted ultrasonic extraction of total flavonoids from Rosa sterilis. Molecules 2021, 26, 3835. [Google Scholar] [CrossRef] [PubMed]
28. Liu, W.; Feng, Y.; Yu, S.; Fan, Z.; Li, X.; Li, J.; Yin, H. The Flavonoid Biosynthesis Network in Plants. Int. J. Mol. Sci. 2021, 22, 12824. [Google Scholar] [CrossRef] [PubMed]
29. Zhang, Y.; Ma, C.; Liu, C.; Wei, F. Luteolin attenuates doxorubicin-induced cardiotoxicity by modulating the PHLPP1/AKT/Bcl-2 signalling pathway. PeerJ 2020, 8, e8845. [Google Scholar] [CrossRef] [PubMed]
30. Dymarska, M.; Janeczko, T.; Kostrzewa-Suslow, E. Glycosylation of Methoxylated Flavonoids in the Cultures of Isaria fumosorosea KCH J2. Molecules 2018, 23, 2578. [Google Scholar] [CrossRef] [PubMed]
31. Zhou, L.-J.; Geng, Z.; Wang, Y.; Wang, Y.; Liu, S.; Chen, C.; Song, A.; Jiang, J.; Chen, S.; Chen, F. A novel transcription factor CmMYB012 inhibits flavone and anthocyanin biosynthesis in response to high temperatures in chrysanthemum. Hortic. Res. 2021, 8, 248. [Google Scholar] [CrossRef]
32. Adato, A.; Mandel, T.; Mintz-Oron, S.; Venger, I.; Levy, D.; Yativ, M.; Domínguez, E.; Wang, Z.; De Vos, R.C.; Jetter, R.; et al. Fruit-surface flavonoid accumulation in tomato is controlled by a SlMYB12-regulated transcriptional network. PLoS Genet. 2009, 5, e1000777. [Google Scholar] [CrossRef] [PubMed]
33. Zhou, X.-J.; Hu, C.-T.; Yan, Y.; Wu, S.; Wang, J. A novel microfluidic aqueous two-phase system with immobilized enzyme enhances cyanidin-3-O-glucoside content in red pigments from mulberry fruits. Biochem. Eng. J. 2020, 158. [Google Scholar] [CrossRef]
34. Allwood, J.W.; Woznicki, T.L.; Xu, Y.; Foito, A.; Aaby, K.; Sungurtas, J.; Freitag, S.; Goodacre, R.; Stewart, D.; Remberg, S.F.; et al. Application of HPLC-PDA-MS metabolite profiling to investigate the effect of growth temperature and day length on blackcurrant fruit. Metabolomics 2019, 15, 12. [Google Scholar] [CrossRef] [PubMed]
35. Heng, Z.; Xu, X.; Xu, X.; Li, Y.; Wang, H.; Huang, W.; Yan, S.; Li, T. Integrated transcriptomic and metabolomic analysis of C frutescens fruits provides new insight into the regulation of the branched chain esters and capsaicin biosynthesis. Food Res. Int. 2023, 169, 112856. [Google Scholar] [CrossRef] [PubMed]
36. Zhang, A.; Zheng, J.; Chen, X.; Shi, X.; Wang, H.; Fu, Q. Comprehensive Analysis of Transcriptome and Metabolome Reveals the Flavonoid Metabolic Pathway Is Associated with Fruit Peel Coloration of Melon. Molecules 2021, 26, 2830. [Google Scholar] [CrossRef] [PubMed]
37. Aksamit-Stachurska, A.; Korobczak-Sosna, A.; Kulma, A.; Szopa, J. Glycosyltransferase efficiently controls phenylpropanoid pathway. BMC Biotechnol. 2008, 8, 25. [Google Scholar] [CrossRef] [PubMed]
38. Kong, X.; Khan, A.; Li, Z.; You, J.; Munsif, F.; Kang, H.; Zhou, R. Identification of chalcone synthase genes and their expression patterns reveal pollen abortion in cotton. Saudi J. Biol. Sci. 2020, 27, 3691–3699. [Google Scholar] [CrossRef] [PubMed]
39. Wang, Z.; Yu, Q.; Shen, W.; El Mohtar, C.A.; Zhao, X.; Gmitter, F.G., Jr. Functional study of CHS gene family members in citrus revealed a novel CHS gene affecting the production of flavonoids. BMC Plant Biol. 2018, 18, 189. [Google Scholar] [CrossRef] [PubMed]
40. Zhao, Y.; Zhang, Y.-Y.; Liu, H.; Zhang, X.-S.; Ni, R.; Wang, P.-Y.; Gao, S.; Lou, H.-X.; Cheng, A.-X. Functional characterization of a liverworts bHLH transcription factor involved in the regulation of bisbibenzyls and flavonoids biosynthesis. BMC Plant Biol. 2019, 19, 1–13. [Google Scholar] [CrossRef]
41. Pang, Y.; Wenger, J.P.; Saathoff, K.; Peel, G.J.; Wen, J.; Huhman, D.; Allen, S.N.; Tang, Y.; Cheng, X.; Tadege, M. A WD40 repeat protein from Medicago truncatula is necessary for tissue-specific anthocyanin and proanthocyanidin biosynthesis but not for trichome development. Plant Physiol. 2009, 151, 1114–1129. [Google Scholar] [CrossRef]
42. Castellarin, S.D.; Matthews, M.A.; Di Gaspero, G.; Gambetta, G.A. Water deficits accelerate ripening and induce changes in gene expression regulating flavonoid biosynthesis in grape berries. Planta 2007, 227, 101–112. [Google Scholar] [CrossRef] [PubMed]
43. Prior, R.; Cao, G.; Martin, A.; Sofi, E.; Mcewen, J.; O’Brien, C.; Lischner, N.; Ehlenfeldt, M.; Kalt, W.; Krewer, G. Antioxidant capacity as influenced by total phenolic and anthocyanin content, maturity, and variety of Vaccinium species. J. Agric. Food Chem. 1998, 46, 2686–2693. [Google Scholar] [CrossRef]
44. Zoratti, L.; Karppinen, K.; Luengo Escobar, A.; Häggman, H.; Jaakola, L. Light-controlled flavonoid biosynthesis in fruits. Front. Plant Sci. 2014, 5, 534. [Google Scholar] [CrossRef] [PubMed]
45. Ralley, L.; Enfissi, E.M.; Misawa, N.; Schuch, W.; Bramley, P.M.; Fraser, P.D. Metabolic engineering of ketocarotenoid formation in higher plants. Plant J. 2004, 39, 477–486. [Google Scholar] [CrossRef] [PubMed]
46. Zanoaga, O.; Braicu, C.; Jurj, A.; Rusu, A.; Buiga, R.; Berindan-Neagoe, I. Progress in Research on the Role of Flavonoids in Lung Cancer. Int. J. Mol. Sci. 2019, 20, 4291. [Google Scholar] [CrossRef]
Yang Y, Cai Q, Wang X, Yang Y, Li L, Sun Z, Li W. Transcriptomic and Metabolomic Analyses Reveal Differences in Flavonoid Synthesis During Fruit Development of Capsicum frutescens pericarp. Agriculture. 2025; 15(2):222. https://doi.org/10.3390/agriculture15020222
Перевод статьи «Transcriptomic and Metabolomic Analyses Reveal Differences in Flavonoid Synthesis During Fruit Development of Capsicum frutescens pericarp» авторов Yang Y, Cai Q, Wang X, Yang Y, Li L, Sun Z, Li W., оригинал доступен по ссылке. Лицензия: CC BY. Изменения: переведено на русский язык
































































































Комментарии (0)