Новый генетический ключ к цинку в пшенице: как маркер KAZn-2D-3 поможет выращивать более полезные сорта
Цинк является важным микроэлементом для ферментов и белков как в зерне
пшеницы, так и в организме человека. Недостаток цинка может привести к снижению
урожайности пшеницы и низкому содержанию цинка в зерне, что, в свою очередь,
ведет к недостаточному потреблению цинка с пищей.
Аннотация
Для картирования количественных trait loci (QTL), связанных с содержанием цинка в зерне (GZnC), была использована популяция рекомбинантных инбредных линий (RIL), полученная от скрещивания Avocet/Huites (популяция AH), с применением технологии DArT (diversity array technology). Было идентифицировано девять QTL на хромосомах 2D, 3B, 4A, 4D, 5A, 5B, 6A, 7A и 7D. Среди них QTL QGZn.haust-AH-2D выявлялся в нескольких условиях окружающей среды и объяснял от 5,61% до 11,27% фенотипической изменчивости, располагаясь в физическом интервале от 13,62 Mb до 17,82 Mb.
Параллельно, полногеномный анализ ассоциаций (GWAS), проведенный на панели из 243 сортов или продвинутых линий (популяция CH) с использованием 660K SNP-чипа пшеницы, также выявил генетические локусы, ассоциированные с содержанием цинка. Один из значимых сигналов был обнаружен в физическом интервале от 13,61 Mb до 15,12 Mb на хромосоме 2D и объяснял от 8,99% до 11,58% фенотипической изменчивости в пяти моделях.
Был разработан высокопроизводительный KASP-маркер (competitive allele specific PCR), который подтвердил ассоциацию в естественной популяции пшеницы (популяция NA). Локус QGZn.haust-AH-2D был точно картирован в узкую область, названную TaZn-2DS, между маркерами KAZn-2D-3 и 1111273 на физическом расстоянии 2,70 Mb. Генетический эффект локуса TaZn-2DS составил 11,43%.
Данное исследование показывает, что локус TaZn-2DS ассоциирован с содержанием цинка, и разработанный маркер KAZn-2D-3 может быть использован для генетического улучшения питательного качества пшеницы.
1. Введение
Пшеница (Triticum aestivum L.) не только удовлетворяет примерно 20% потребностей человека в энергии и растительном белке, но и обеспечивает многочисленными необходимыми питательными веществами, включая микроэлементы, пищевые волокна и липиды. Это одна из важнейших продовольственных культур в мире [1, 2]. Дефицит цинка (Zn) может привести к проявлению скрытого голода, от которого страдают около 200 миллионов человек в развивающихся странах [3]. Некоторые предыдущие исследования показывают, что примерно 17% населения мира испытывают недостаток цинка [4, 5]. Дефицит цинка в организме человека может привести к нарушению роста и развития, ослаблению иммунной функции, а также к дефициту в обучении и памяти [6]. Предыдущие исследования показали, что организм человека может получать цинк из ежедневной пищи, включая пшеницу [7]. Содержание цинка в зерне пшеницы (GZnC) составляло в среднем 37 мг/кг в развитых странах, а в Китае — более 30 мг/кг [2]. Следовательно, генетическое улучшение является экономичным и эффективным методом повышения содержания цинка в зерне пшеницы с целью смягчения дефицита цинка у человека [8, 9].
В связи с растущим вниманием к селекции на повышение питательной ценности различные международные организации и страны запустили исследовательские программы. В 2022 году Национальная комиссия по развитию и реформам Китая опубликовала «План развития биологической экономики на период 14-й пятилетки» (https://www.ndrc.gov.cn/xxgk/jd/jd/202205/t20220509_1324417.html).
Международный центр улучшения кукурузы и пшеницы (CIMMYT) планирует усилить селекцию на содержание GZnC примерно в течение 10 лет [10]. Однако выращивание превосходных сортов с высоким содержанием GZnC сталкивается со многими трудностями [11]. Благодаря быстрому развитию биотехнологий исследователи значительно повысили генетическое содержание цинка и улучшили питательные качества, используя полногеномное ассоциативное исследование (GWAS) или анализ сцепления. Баейсовская информация о неравновесии сцепления итеративно вложенных ключей (BLINK) была использована для проведения многолокусного полногеномного ассоциативного исследования (ML-GWAS) на 161 заменителе пшеницы и Aegilops, и было идентифицировано 44 ассоциации маркер–признак (MTA) [12]. Чжоу и др. [13] использовали три модели, включая GLM, смешанную линейную модель (MLM) и модель циркулирующей вероятности с фиксированными и случайными параметрами (FarmCPU), чтобы идентифицировать 29 локусов GZnC на хромосоме 3D. Рен и др. [14] обнаружили 11 QTL GZnC на хромосомах 1B, 2B, 3B, 4A, 4B, 5A, 5D, 6A и 7A с использованием популяции RIL Avocet/Chilero, с фенотипическими эффектами в диапазоне от 1,07% до 16,62%, и один KASP-маркер, разработанный для QGZn.haust-5DL. В дополнение, четыре QTL, связанные с GZnC, были обнаружены с использованием популяции RIL Zhou 8425B/Chinese Spring. Локусы на хромосомах 3B, 4A, 5A и 5B объясняли от 3,5% до 8,7% фенотипической изменчивости. Шесть KASP-маркеров, разработанных для QGZnCzc.caas-3BL, QGZnCzc.caas-5AL и QGZnCzc.caas-5BL, были проверены на 125 повторно секвенированных сортах пшеницы. Среди них доля благоприятных аллелей в местных сортах была выше, чем в современных сортах [15]. Предыдущее картирование QTL и разработка KASP-маркеров считаются отличными инструментами. В данном исследовании мы поставили перед собой следующие цели: (1) идентифицировать стабильные локусы, связанные с GZnC, на основе картирования сцепления и анализа GWAS; (2) разработать KASP-маркеры, ассоциированные с новыми локусами; (3) провести точное картирование стабильных локусов, связанных с GZnC, методом клонирования на основе карты; (4) повысить эффективность точного картирования локусов GZnC и проанализировать их генетические эффекты. Это исследование дополнительно предоставит некоторые новые гены и маркеры для генетического улучшения питательных качеств пшеницы. Насколько нам известно, это первое сообщение о поиске генов для содержания цинка в пшенице с использованием комбинации GWAS, анализа сцепления и разработки KASP-маркеров.
2. Материалы и методы
2.1. Растительный материал
Популяция RIL была получена путем скрещивания Avocet/Huites (175 линий F6) («популяция AH»). Подробности об этой популяции описаны в предыдущем исследовании нашей группы [16].
Растительный материал включал 243 сорта («популяция CH»), предоставленные профессором Чен Фэном из Хэнаньского сельскохозяйственного университета. Все сорта были из долины Хуанхуай. Подробное описание этой популяции приведено в предыдущем исследовании нашей группы (Таблица S1) [17, 18].
Для проверки KASP-маркеров была использована одна природная популяция из 110 сортов («популяция NA»). Из них 40 сортов происходят из отечественных источников, а остальные 70 сортов — из зарубежных стран, таких как европейские страны, США, Мексика и Австралия. Материалы были предоставлены в рамках проекта по инновациям в области генофонда пшеницы и молекулярной селекции Хэнаньского университета науки и технологий в Китае (Таблица S2).
2.2. Планирование полевого эксперимента
Популяция AH выращивалась в трех условиях: на экспериментальном поле Хэнаньского университета науки и технологий (112°42′ в.д., 34°60′ с.ш.) в сезоне 2019–2020 гг. (E1), а также на экспериментальном поле Хэнаньского университета науки и технологий (E2) и в уезде Мэнцзинь (112°59′ в.д., 34°82′ с.ш.) (E3) в сезоне 2020–2021 гг. Популяция CH выращивалась в двух условиях: в уезде Мэнцзинь (E5) и на экспериментальном поле Хэнаньского университета науки и технологий (E6) в сезоне 2020–2021 гг. Популяция NA была высажена на поле сельскохозяйственного колледжа Хэнаньского университета науки и технологий в сезоне 2019–2020 гг. (E8). На каждом участке посевы проводились по схеме рандомизированных полных блоков с тремя повторениями. Каждый участок состоял из одного ряда длиной 2,0 м, расположенного на расстоянии 20 см друг от друга, с расстоянием между растениями 8 см и 25 семенами в каждом ряду. Все полевые испытания проводились в соответствии со стандартными методами. Содержание щелочно-гидролизуемого азота и доступного цинка на экспериментальном поле Хэнаньского университета науки и технологий составляло 64,75 мг/кг и 1,73 мг/кг соответственно, а содержание щелочно-гидролизуемого азота и доступного цинка в уезде Мэнцзинь — 55,85 мг/кг и 1,36 мг/кг соответственно.
2.3. Фенотипическая оценка
GZnC определяли с использованием метода, описанного Реном и др. [14], с небольшими оптимизациями и корректировками.
(1) Когда пшеница созрела, мы собрали и обмолотили ее вручную. Примерно 2 г нормального зерна были отобраны случайным образом, помещены в бумажный пакет, запечатаны и высушены при температуре 80 °C до достижения постоянного веса.
(2) После сушки образцы измельчали с помощью шаровой мельницы из карбида вольфрама (Retsch MM400, Haan, Германия). Измельчитель был установлен на 20 оборотов в секунду и работал в течение 30 секунд для получения цельнозерновой пшеничной муки.
(3) В пробирку для разложения (HVE56) было взвешено 0,2 г сухой цельнозерновой пшеничной муки. Добавили 5 мл раствора HNO₃ (аналитической чистоты). Пробирку поместили в микроволновую печь для разложения (Anton-Paar, Multiwave 4000, Грац, Австрия) и выдерживали до полной прозрачности. После охлаждения объем довели до 25 мл.
(4) Содержание GZnC определяли с помощью оптического эмиссионного спектрометра с индуктивно связанной плазмой (Agilent 5110 ICP-OES, Санта-Клара, Калифорния, США). Длина волны детектирования была установлена на 213,857 нм.
(5) Формула для расчета GZnC выглядит следующим образом:
В формуле X, C, C₀, m и V представляют содержание цинка в образце, определенное содержание цинка в образце, содержание цинка в контрольном образце, массу образца и общий объем обработки образца соответственно.
2.4. Статистический анализ
Статистический анализ проводился с использованием программного обеспечения Statistical Product and Service Solutions («SPSS») 17.0 (IBM, Нью-Йорк, штат Нью-Йорк, США). Для оценки эффектов генотипа (G), окружающей среды (E) и взаимодействия G × E использовался дисперсионный анализ (ANOVA). Наилучшие линейные несмещенные оценки (BLUE) и коэффициент корреляции Пирсона (r) рассчитывались с помощью инструмента «AOV» в программном обеспечении QTL IciMapping v4.2 и R соответственно [19, 20]. Диаграммы фенотипической корреляции строились с помощью программного обеспечения Origin 22b. Наследуемость в широком смысле (H²) для GZnC рассчитывалась по следующей формуле:
В формуле σ²g, σ²ge, σ²ε и n представляют, соответственно, дисперсию генотипа, дисперсию взаимодействия генотипа и окружающей среды, дисперсию окружающей среды и количество условий окружающей среды.
Наилучшие линейные несмещенные оценки (BLUE) GZnC в популяции AH и популяции CH были обозначены как E4 (AH-BLUE) и E7 (CH-BLUE) соответственно.
2.5. Полногеномное ассоциативное исследование (GWAS)
Генотипирование природной популяции проводилось с использованием массива SNP пшеницы 660 K в Пекинской компании CapitalBio Technology (Пекин, Китай) (https://www.capitalbiotech.com/, дата обращения: 10 августа 2018 г.). Молекулярные данные были предоставлены исследовательской группой профессора Чен Фэна из Хэнаньского сельскохозяйственного университета. Контроль качества и анализ главных компонент (PCA) данных генотипирования были взяты из работы Ли и др. [17]. Анализ контроля качества, анализ родства, анализ популяционной структуры и анализ неравновесия сцепления природной популяции подробно описаны в работах Ян и др. [21] и Лю и др. [22]. При этом расстояния распада LD для геномов A, B и D составляли 3 Мб, 10 Мб и 3 Мб соответственно.
GWAS проводился с использованием программного обеспечения R 4.4.1, и для анализа были приняты модели General Linear Model (GLM), Mixed Linear Model (MLM), Fixed and random model Circulating Probability Unification (FarmCPU) [23], Bayesian-information and Linkage-disequilibrium Iteratively Nested Keyway (BLINK) [24] и Settlement of MLMs Under Progressively Exclusive Relationship (SUPER) [25].
Во всех пяти моделях в качестве фиксированного эффекта использовался метод главных компонент (PCA), а в качестве случайного эффекта — матрица K. Они были объединены с GZnC в двух средах, и в анализе ассоциаций использовались значения BLUE. Для выявления более значимых маркеров пороговое значение значимости SNP установлено на уровне 1,0 × 10⁻⁴. Диаграмма Манхэттена и квантильно-квантильная диаграмма (Q-Q диаграмма) визуализируются с помощью пакета CMplot в программном обеспечении R 4.4.1 (https://github.com/YinLiLin/R-CMplot, дата обращения: 20 мая 2024 г.).
2.6. Построение карты генетического сцепления
В эксперименте использовалась высокоплотная генетическая карта сцепления популяции AH, построенная на маркерах DArT [26]. Молекулярные данные популяции AH были предоставлены группой Ван Сюэ из Нанкинского сельскохозяйственного университета и группой доктора Рави П. Сингха из CIMMYT соответственно. Картирование QTL проводилось с использованием метода составного интервального картирования (Inclusive Composite Interval Mapping, ICIM) в QTL IciMapping v4.2. Порог LOD был установлен на 2,5. QTL с расстоянием менее 10 сМ или имеющие общие фланкирующие маркеры считались одним и тем же локусом. QTL с фенотипическим вкладом >10% считался основным QTL [26]. Метод именования QTL описан McCouch и др. [27], а карта генетической связи была построена с использованием MapChart2.3 (https://www.wur.nl/en/show/Mapchart.htm).
2.7. Разработка KASP-маркеров
Маркеры KASP были разработаны с использованием базовых консенсусных последовательностей SNP, полученных из массива 660 K, и были загружены в PolyMarker (http://www.polymarker.info/).
Процедура амплификации описана в работе Ли и др. [17]. Значимость фенотипических различий между генотипами KASP-маркеров рассчитывалась с использованием «t-критерия».
2.8. Идентификация генов-кандидатов
Последовательности молекулярных маркеров, тесно связанные с QTL, были подвергнуты выравниванию BLAST на веб-сайте Ensemble Plants v57 (https://plants.ensembl.org/index.html, дата обращения: 24 мая 2024 г.) для получения физических положений маркеров. Высокодостоверно аннотированные гены в пределах целевого физического местоположения были получены с помощью веб-сайта IWGSC RefSeq v1.1 (https://wheat-urgi.versailles.inra.fr/, дата обращения: 25 мая 2024 г.). Уровни экспрессии и паттерны генов-кандидатов были проанализированы с помощью базы данных экспрессии гексаплоидной пшеницы на веб-сайте WheatOmics 1.0 (http://202.194.139.32/expression/index.html, дата обращения: 23 июня 2024 г.) [28].
3. Результаты
3.1. Фенотипическая изменчивость GZnC в популяции AH и популяции CH
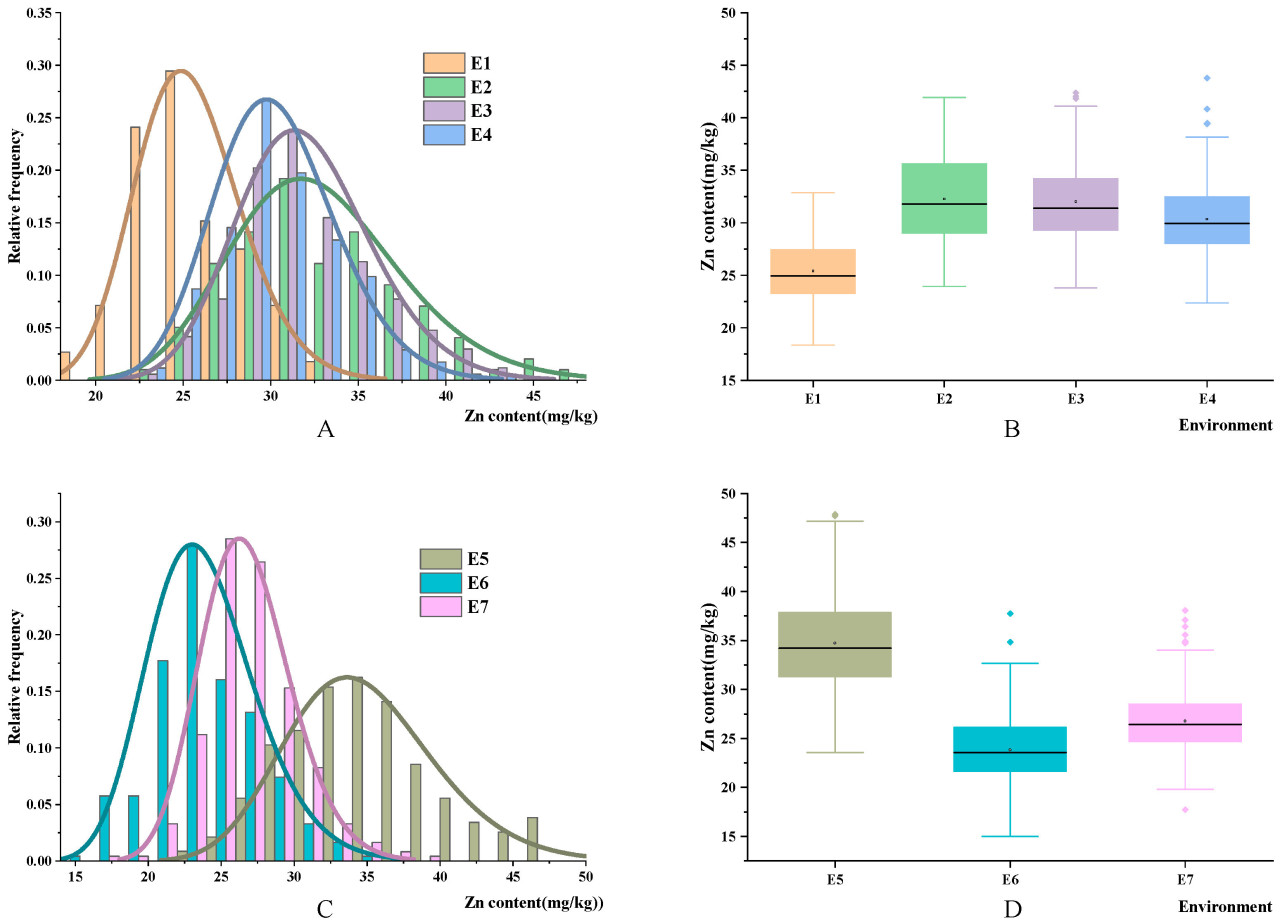
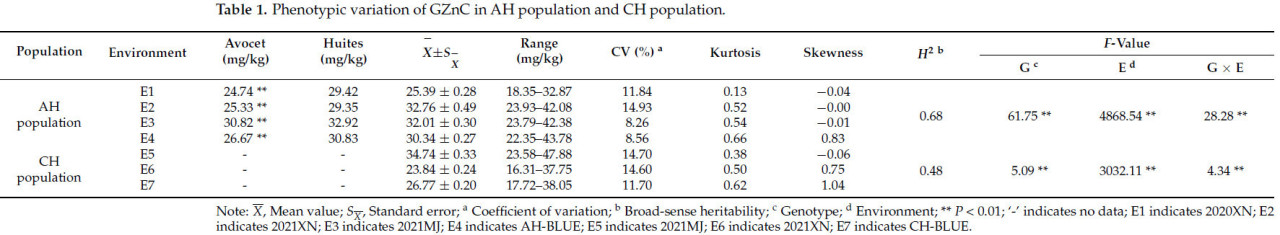
В популяции AH наблюдалась значительная вариация концентрации GZnC, варьирующаяся от 18,35 до 43,78 мг/кг, а коэффициент вариации составлял от 8,26% до 14,93%. Концентрация GZnC у Huites была значительно выше (P < 0,01), чем концентрация GZnC у Avocet (Рисунок 1, Таблица 1). Согласно фенотипическому распределению, асимметрия и эксцесс GZnC в трех полевых экспериментах были меньше 1,0, что указывает на непрерывное нормальное распределение GZnC с широким диапазоном вариаций.
Рисунок 1. Распределение GZnC в популяции AH и популяции CH. Примечание: E1 обозначает 2020XN; E2 обозначает 2021XN; E3 обозначает 2021MJ; E4 обозначает AH-BLUE; E5 обозначает 2021MJ; E6 обозначает 2021XN; E7 обозначает CH-BLUE. (A): диаграмма нормального распределения GZnC в популяции AH; (B): распределение содержания Zn в GZnC в популяции AH, точки на рисунке представляют собой выбросы; (C): диаграмма нормального распределения GZnC в популяции CH; (D): распределение содержания Zn в GZnC в популяции CH. Точки на рисунке представляют собой выбросы.
Таблица 1. Фенотипическая изменчивость GZnC в популяции AH и популяции CH.
Фенотипическая изменчивость GZnC была проанализирована в популяции CH (Рисунок 1, Таблица 1). В двух условиях выращивания и при значениях BLUE содержание GZnC в популяции CH было значимым, варьируя от 16,31 мг/кг до 47,88 мг/кг, а коэффициент вариации составлял от 11,70% до 14,70%. Абсолютные значения эксцесса и асимметрии составляли 0,38–0,62 и 0,06–1,04 соответственно, и GZnC демонстрировал непрерывное нормальное распределение с широким диапазоном вариаций. Генетическое разнообразие популяции CH было достаточно богатым для проведения GWAS.
GZnC оказывал очень значительное (P < 0,01) влияние на генотип (G), окружающую среду (E) и взаимодействие между генотипом и окружающей средой (G × E) (Таблица 1). Наследуемость в широком смысле (H²) популяции AH и популяции CH составила 0,68 и 0,48 соответственно.
3.2. Анализ сцепления и анализ генетического эффекта QTL гена GZnC в популяции AH
Для GZnC был проведен анализ сцепления при значениях AH-BLUE и в трех условиях окружающей среды (E1, E2, E3, E4). В общей сложности было идентифицировано девять QTL на хромосомах 2D, 3B, 4A, 4D, 5A, 5B, 6A, 7A и 7D для GZnC, что составляет от 4,31% до 17,36% фенотипической изменчивости (Таблица 2). Кроме того, QGZn.haust-AH-2D и QGZn.haust-AH-6A оказались стабильными QTL, которые можно было обнаружить в условиях окружающей среды E1/E2 и E2/E4 соответственно (Рисунок 2 и Рисунок S1). QTL QGZn.haust-AH-2D был тесно связан с маркерами DArT 1111273 и SNP4993218, расположенными на участке от 13,62 Мб до 17,82 Мб, и составлял от 5,61% до 11,27% фенотипической изменчивости. Аллель с возрастающим эффектом QGZn.haust-AH-2D был получен от мужского родителя Huites. QTL QGZn.haust-AH-6A был тесно связан с маркерами DArT 100462039 и 100592272, расположенными на участке от 484,85 Мб до 601,40 Мб, и составлял от 14,20% до 17,36% фенотипической изменчивости. Ген с возрастающим эффектом QGZn.haust-AH-6A был получен от женского родителя Avocet (Таблица 2).
Рисунок 2. Картирование QTL для GZnC. Примечание: E1 обозначает 2020XN; E2 обозначает 2021XN; E3 обозначает 2021MJ; E4 обозначает AH-BLUE. Коричневым цветом обозначены левый и правый маркеры локусов.
Таблица 2. QTL гена GZnC, выявленные методом ICIM-ADD в популяции AH.
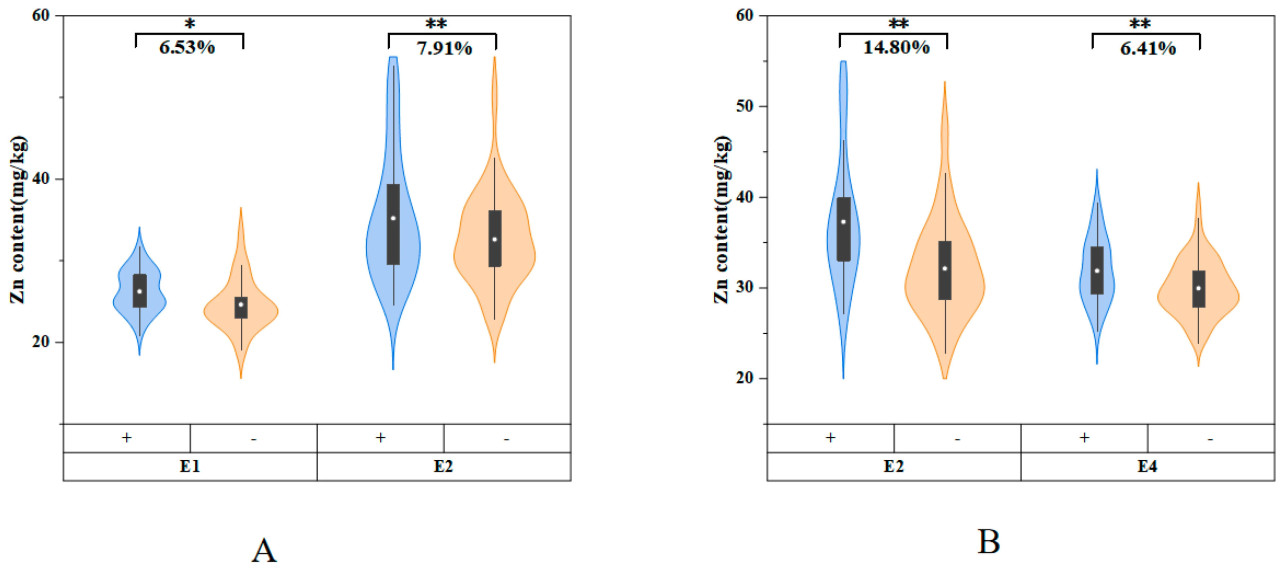
Анализ генетического эффекта QGZn.haust-AH-2D и QGZn.haust-AH-6A проводился в вышеупомянутых условиях (Рисунок 3). Средний генетический эффект материалов, несущих QGZn.haust-AH-2D, составил 6,53% в E1, что значительно выше, чем у линий без QGZn.haust-AH-2D, и 7,91% в E2, что чрезвычайно значительно выше, чем у линий без эффекта QGZn.haust-AH-2D. Генетический эффект GZnC линий, несущих QGZn.haust-AH-6A, составил от 6,41% до 14,80% как в условиях E2, так и в условиях E4, что чрезвычайно значительно выше, чем у линий без QGZn.haust-AH-6A.
Рисунок 3. Генетические эффекты QGZn.haust-AH-2D (A) и QGZn.haust-AH-6A (B), обеспечивающие наличие GZnC. обозначает P < 0,05. обозначает P < 0,01.
3.3. GWAS QGZn.haust-AH-2D в популяции CH
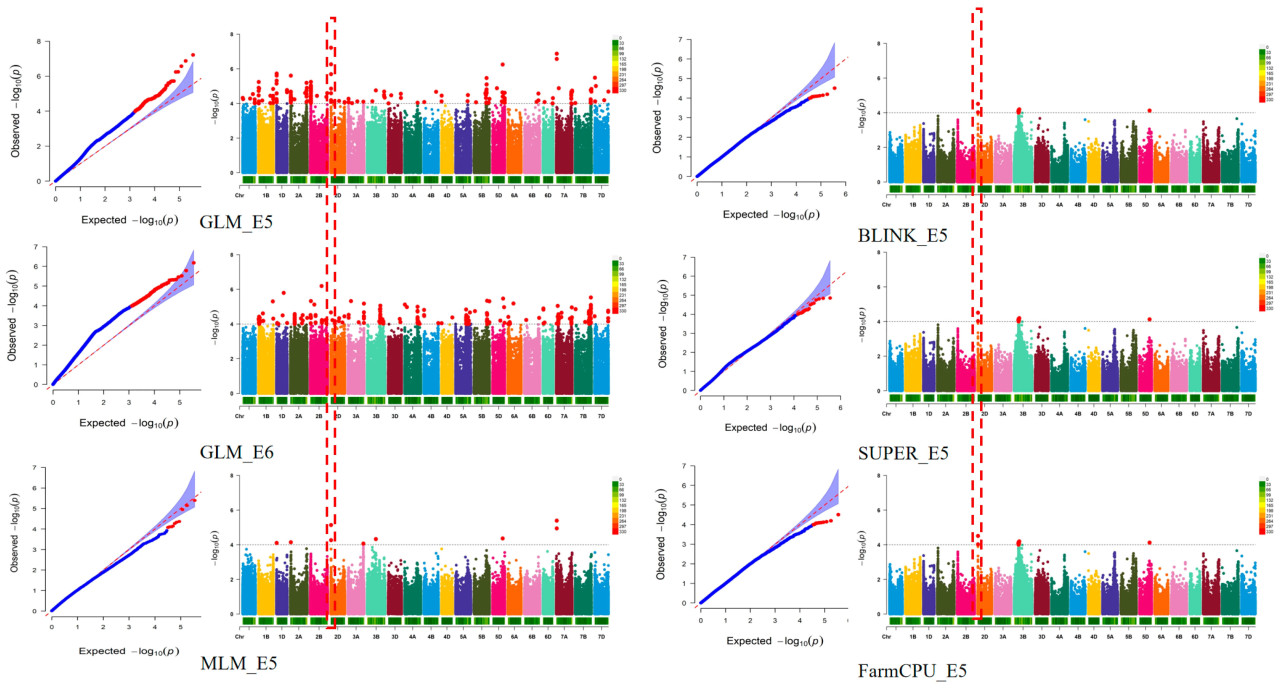
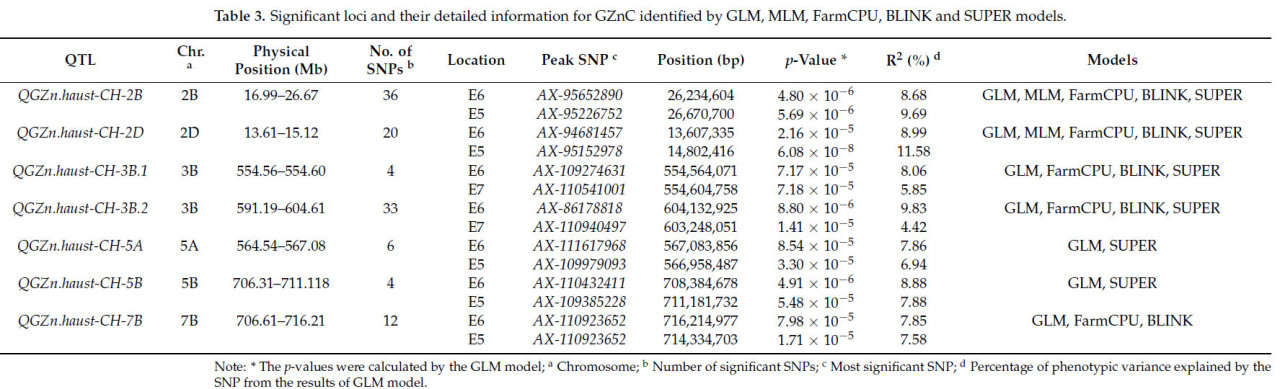
Для дальнейшей проверки QGZn.haust-AH-2D были использованы пять моделей GWAS (GLM, MLM, FarmCPU, BLINK и SUPER) для анализа GZnC при значениях CH-BLUE и в двух условиях окружающей среды. В общей сложности было выявлено 591 значимый SNP (P < 1,0 × 10⁻⁴) на 21 хромосоме (Рисунок 4), при этом 115 значимых SNP были обнаружены в нескольких моделях и в нескольких условиях окружающей среды. Согласно значению неравновесия сцепления (LD), оно было разделено на семь локусов (Таблица 3), на которые приходится от 4,42% до 11,58% фенотипической изменчивости. Кроме того, было обнаружено, что QGZn.haust-CH-2D присутствует в двух средах E5/E6 и расположен в пределах физического интервала от 13,61 Мб до 15,12 Мб на коротком плече хромосомы 2D, составляя от 8,99% до 11,58% фенотипической изменчивости (Таблица 3, Рисунок 4).
Рисунок 4. Диаграммы Манхэттена и QQ-диаграммы для GZnC, построенные с использованием моделей GLM, MLM, FarmCPU, BLINK и SUPER в QGZn.haust-CH-2D. Примечание: на QQ-диаграмме красным цветом обозначены новые локусы мутаций. Пунктирные прямоугольники представляют диаграмму Манхэттена для QGZn.haust-AH-2D. Пунктирная горизонтальная линия обозначает пороговое значение значимости −log10(P) = 4,0. SNP выше красной пунктирной линии значимо ассоциированы с Zn.
Таблица 3. Значимые локусы и подробная информация о них для GZnC, выявленные с помощью моделей GLM, MLM, FarmCPU, BLINK и SUPER.
Гаплотипический анализ QGZn.haust-CH-2D выявил два превосходных гаплотипа (Рисунок 5(Bd)), содержащих 20 SNP, значительно связанных с GZnC (Таблица 3). В популяции CH генетический эффект QGZn.haust-CH-2D_H1 составил 14,65%, а QGZn.haust-CH-2D_H2 — 8,92% (Рисунок 5(Bd)).
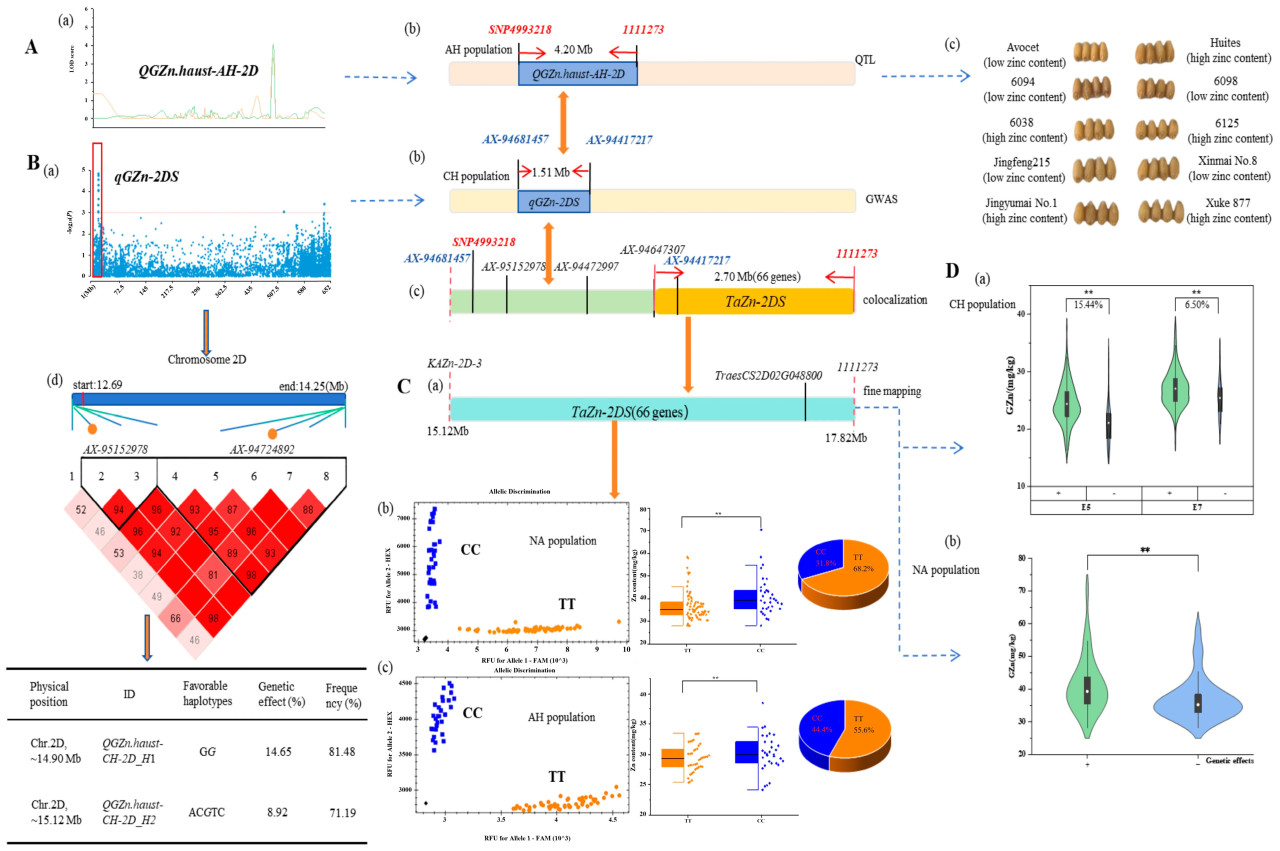
Рисунок 5. Детальное картирование TaZn-2DS для GZnC. (A): Картирование QTL популяции AH на хромосоме 2D. Примечание: (a) LOD популяции AH на хромосоме 2D, оранжевым цветом обозначено значение LOD для 2020XN, а зеленым — значение LOD для 2021XN; (b) Физическое расположение и длина QGZn.haust-AH-2D в популяции AH; (c) Семена между сортами (линиями) с низким содержанием GZnC и сортами с высоким содержанием GZnC в популяции AH и популяции CH. (B): Интервал совместного расположения на хромосоме 2D в популяции AH и популяции CH. Примечание: (a) GWAS-анализ популяции CH на хромосоме 2D; (b) Физическое расположение и длина QGZn.haust-CH-2D в популяции CH; (c) Интервал совместного расположения на хромосоме 2D в популяции AH и популяции CH; (d): Гаплотипический анализ QGZn.haust-CH-2D. (C): Детальное картирование и генотипирование TaZn-2DS в популяции NA и популяции AH. Примечание: (a) Детальное картирование TaZn-2DS; (b) Генотипирование TaZn-2DS с помощью KAZn-2D-3 в популяции NA; (c) Генотипирование TaZn-2DS с помощью KAZn-2D-3 в популяции AH. (D): Генетический эффект TaZn-2DS. (a) Генетический эффект TaZn-2DS в популяции CH; (b) Генетический эффект TaZn-2DS в популяции NA. Примечание: обозначает P < 0,01.
3.4. Анализ совместной локализации QGZn.haust-AH-2D и QGZn.haust-CH-2D
В данном исследовании локус QGZn.haust-AH-2D был локализован в диапазоне от 13,62 Мб до 17,82 Мб (E1, E2) с помощью анализа сцепления, а локус QGZn.haust-CH-2D — в диапазоне от 13,61 Мб до 15,12 Мб (E5, E6) с использованием GWAS. Интервал ко-локализации этих локусов составил от 13,61 Мб до 17,82 Мб и был назван TaZn-2DS (Рисунок 5 A,B).
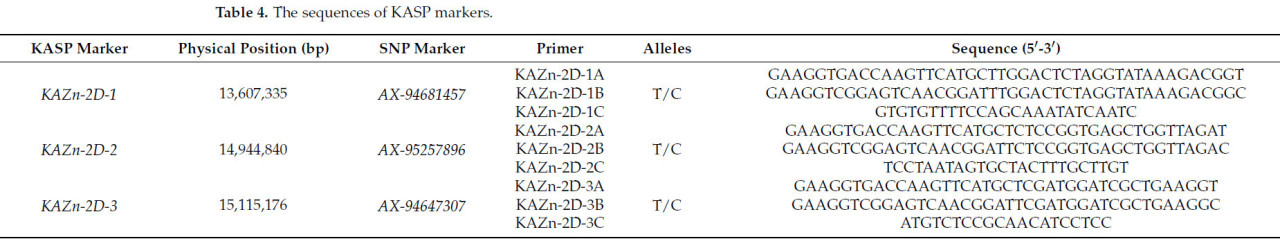
3.5. Разработка и валидация KASP-маркеров
Для повышения эффективности отбора GZnC при обогащении пшеницы питательными веществами мы разработали три KASP-маркера. Один из KASP-маркеров, названный KAZn-2D-3, был тесно связан с TaZn-2DS (как показано в Таблице 4 и на Рисунке 5 (Cb)) и был обнаружен в результате валидации популяции NA, состоящей из 110 выдающихся сортов и линий пшеницы. Гаплотипы были TT (неблагоприятный) и CC (благоприятный), что подтверждается наличием 34 и 73 сортов в популяции NA для KAZn-2D-3. Значение GZnC у сортов с аллельной вариацией TT было очень значительно ниже, чем у другого сорта с аллельной вариацией CC. Аллельная вариация CC увеличила значение GZnC на 11,43% на Рисунке 5 (Cb).
Таблица 4. Последовательности KASP-маркеров.
3.6. Точное картирование и анализ генетического эффекта TaZn-2DS
Локус, названный TaZn-2DS, был локализован между KAZn-2D-3 и 1111273 с помощью точного картирования (Рисунок 5 (Ca,Cc)). Генотипирование KAZn-2D-3 было проведено в популяции AH на Рисунке 5 (Cc), и KAZn-2D-3, маркер DArT 1111273 и SNP4993218 были использованы для реконструкции генетического картирования сцепления. В итоге, 66 высокодостоверно аннотированных генов находились в пределах кандидатного региона TaZn-2DS (15,12–17,82 Мб).
Анализ генетического эффекта TaZn-2DS был проведен в популяции NA и популяции CH (Рисунок 5 (Da,Db)). В популяции NA генотип GZnC сортов или перспективных линий, несущих доминантный аллель TaZn-2DS, был на 11,34% выше, чем у сортов без доминантного аллеля TaZn-2DS в условиях E8 (P < 0,01). В популяции CH генотип GZnC сортов, несущих доминантный аллель TaZn-2DS, был на 6,50–15,44% выше, чем у сортов без доминантного аллеля TaZn-2DS в условиях E5 и E7 (P < 0,01).
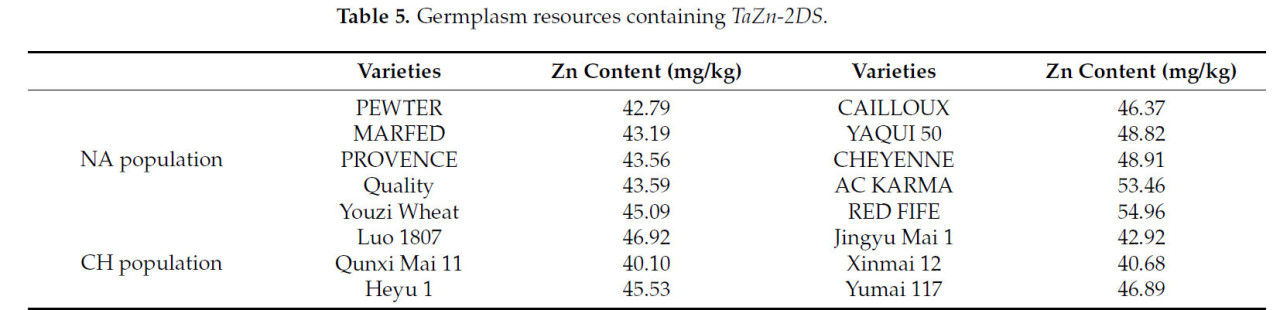
Тем временем были идентифицированы генетические ресурсы, содержащие доминантный аллель TaZn-2DS. Некоторые сорта, такие как PEWTER, MARFED, PROVENCE, Quality, Youzi Wheat, CAILLOUX, YAQUI 50, CHEYENNE, AC KARMA и RED FIFE, соответственно, содержали доминантный аллель TaZn-2DS и были исследованы в популяции NA. Некоторые сорта, такие как Qunxi Mai 11, Heyu 1, Luo 1807, Jingyu Mai 1, Xinmai 12 и Yumai 117, содержали TaZn-2DS и были обнаружены в популяции CH (Таблица 5).
Таблица 5. Генетические ресурсы, содержащие TaZn-2DS.
4. Обсуждение
Цинк является ключевым компонентом зерна и играет жизненно важную роль в росте и развитии всех живых организмов, включая человека [29]. Наша цель состояла в том, чтобы использовать естественные генетические вариации GZnC для увеличения содержания цинка в зерне пшеницы. Предыдущие исследования показали, что GZnC современных сортов пшеницы был ниже и менее изменчив, чем у диких образцов пшеницы [30]. Наше исследование показало, что содержание цинка в древних сортах было выше, чем в современных сортах пшеницы.
Предыдущие исследования выявили многочисленные локусы количественных признаков (QTL), оказывающие значительное влияние на GZnC [2, 12, 13, 15, 31, 32, 33], но существует лишь несколько сообщений об исследовании новых локусов с помощью GWAS и анализа сцепления. В настоящем исследовании локус TaZn-2DS был локализован с помощью GWAS и анализа сцепления, и был обнаружен узкий интервал ко-локализации от 13,61 Мб до 17,82 Мб. Кроме того, были разработаны и успешно проверены KASP-маркеры с использованием генотипов популяции NA с богатым генетическим разнообразием. Наше исследование предлагает новый, надежный и стабильный ген и маркер для улучшения GZnC и селекции на повышение питательной ценности пшеницы.
4.1. Сравнительный анализ значимых локусов в GZnC
GWAS и анализ сцепления были двумя важными методами исследования QTL. Хотя анализ сцепления может выявлять редкие вариации в популяциях RIL, целевой ген будет расположен в большом хромосомном регионе. В отличие от этого, анализ ассоциаций имеет больше преимуществ в картировании целевого гена в меньшем хромосомном регионе [34, 35]. Однако он сталкивается с большими трудностями в обнаружении редких вариаций [36]. Используя GWAS и анализ сцепления, мы идентифицировали локус, значительно связанный с GZnC (QGZn.haust-AH-2D, QGZn.haust-CH-2D) в пределах физического интервала от 13,61 Мб до 17,82 Мб на коротком плече хромосомы 2D. Для проверки QTL были разработаны и использованы три KASP-маркера. Интересно, что генетическое картирование сцепления популяции AH было перерисовано путем объединения маркера KASP с тесно связанным маркером DArT 1111273, и SNP4993218 смог дополнительно сузить физическое расстояние QTL до 2,70 Мб.
В ходе исследования локусов GZnC ученые также обнаружили другие QTL на 2D-хромосомах. Новый локус, QGZnzx.caas-2DL, был обнаружен на 2DL в двух популяциях RIL, а именно ZM175/XY60 и ZM175/LX987, на физическом интервале от 562,7 Мб до 596,2 Мб, что составляет от 3,6% до 9,0% фенотипической изменчивости [2]. В популяции RIL Triticum dicoccon PI94624/Aegilops tauschii[409]//BCN на 2DL были идентифицированы два локуса, QGZn.iari-2D.1 и QGZn.iari-2D.2, на 73,5 Мб – 104,5 Мб и 52,5 Мб – 87,5 Мб физических интервалов соответственно, что составляет 5,05% и 8,11% фенотипической изменчивости соответственно [37]. В популяции RIL HD3086/HI1500 на 2D был обнаружен новый локус, QGZnC.iari-2D, на 843 Мб физического интервала, что составляет 6,10% фенотипической изменчивости [31]. Популяция Hanxuan10/Lumai 14 DH была использована для идентификации нового локуса на 2D, на долю которого приходится 13,4% фенотипической изменчивости [38]; этот локус не совпадал с TaZn-2DS. Поэтому мы предположили, что TaZn-2DS является новым локусом GZnC.
4.2. Анализ генов-кандидатов TaZn-2DS
Результаты выявили 66 генов с высокой степенью достоверности в TaZn-2DS (от 15,12 Мб до 17,82 Мб хромосомы 2D). В зависимости от аннотации генов и прогнозирования экспрессии генов-кандидатов (EnsemblPlants, https://plants.ensembl.org/index.html, дата обращения: 24 мая 2024 г.; WheatOmics 1.0, http://202.194.139.32/, дата обращения: 24 мая 2024 г.), три из 66 генов с высокой степенью достоверности были выбраны в качестве кандидатов: TraesCS2D02G045600, TraesCS2D02G045700 и TraesCS2D02G048800, которые относятся к семейству белков фосфатных транспортеров и семейству белков ABC-транспортеров соответственно. Белки, кодируемые семейством фосфатных транспортеров, в основном участвовали в транспорте ионов металлов, фосфора (0055085 BP) и сахара (PF00083) и могли использовать градиент концентрации ионов водорода на плазматической мембране для обеспечения энергии, необходимой для поглощения ионов металлов растениями [39]. Белки, кодируемые семейством ABC-транспортеров, представляли собой суперсемейство мембранных белков, широко распространенное как у прокариот, так и у эукариот, и выполняли множество функций. Их основная функция заключалась в использовании энергии, генерируемой гидролизом АТФ, для транспортировки субстратов через мембрану против градиента концентрации, главным образом для транспортировки органических и неорганических малых молекул, таких как ионы металлов и сахар [40]. Однако TraesCS2D02G045600 и TraesCS2D02G045700 не экспрессировались в корнях, стеблях, листьях и зернах пшеницы. TraesCS2D02G048800 имел самую высокую экспрессию в листьях через 2 дня после цветения пшеницы. Используя базу данных eFP Browser пшеницы, мы предположили период экспрессии и тканевое обогащение гена TraesCS2D02G048800 на стадиях роста и развития пшеницы. TraesCS2D02G048800 имел самую высокую экспрессию в «семенной оболочке через два дня после цветения» и активно экспрессировался в процессе развития эндосперма (от позднего листового эндосперма до двухклеточного зародыша) [41]. В заключение мы предположили, что ген TraesCS2D02G048800 может быть тесно связан с метаболическим процессом GZnC.
4.3. KASP-маркеры для селекции с использованием молекулярных маркеров
В предыдущих исследованиях Рен и др. [14] разработали KASP-маркеры на хромосоме 5DL. Результаты показали, что Kasp_5D_QGZnFe может эффективно использоваться для молекулярно-маркерной селекции (MAS) GZnC и GFeC. Сунь и др. [15] трансформировали шесть значимых SNP-маркеров, обнаруженных на 3BL, 5AL и 5BL, в KASP-маркеры в 125 сортах, чтобы определить генетические эффекты трех стабильных QTL на GZnC. В то же время шесть KASP-маркеров были отнесены к той же генетической позиции, что и исходный SNP-чип. Результаты показали, что три QTL на GZnC в разных генетических условиях были эффективны. Был разработан KASP-маркер KAZn-2D-3, тесно связанный с QTL QGZn.haust-AH-2D, и лучшие гаплотипы были проверены в двух природных популяциях (популяция CH и популяция NA). Кроме того, маркер KAZn-2D-3 может использоваться в качестве функционального маркера для маркер-ориентированной селекции с целью повышения питательной ценности пшеницы в процессе селекции, а также может помочь селекционерам эффективно и точно отбирать генетические ресурсы по содержанию цинка.
5. Выводы
В данном исследовании мы обнаружили новый QTL, TaZn-2DS, определяющий содержание цинка в зерне пшеницы, и провели детальное картирование области TaZn-2DS, сузив ее до 2,70 Мб. KAZn-2D-3, KASP-маркер, может быть использован в молекулярно-ориентированной селекции для генетического улучшения питательных качеств пшеницы.
Дополнительные материалы
Следующие дополнительные материалы можно загрузить по адресу: https://www.mdpi.com/article/10.3390/agriculture15020124/s1, Рисунок S1: Картирование QTL GZnC; Таблица S1: Названия сортов популяции CH; Таблица S2: Названия сортов популяции NA.
Ссылки
1. Fan, J.; Jin, Y.; Liu, J.; Fei, X. Studies on the Degradation Pattern of Wheat Malt Endo-1,4-β-Xylanase on Wheat-Derived Arabinoxylans. J. Sci. Food Agric. 2024, 104, 4278–4285. [Google Scholar] [CrossRef] [PubMed]
2. Sun, M.; Luo, Q.; Zheng, Q.; Tong, J.; Wang, Y.; Song, J.; Zhang, Y.; Pu, Z.; Zheng, J.; Liu, L.; et al. Molecular Characterization of Stable QTL and Putative Candidate Genes for Grain Zinc and Iron Concentrations in Two Related Wheat Populations. Theor. Appl. Genet. 2023, 136, 217. [Google Scholar] [CrossRef]
3. Kandwal, P.; Fujiwara, T.; Kamiya, T. OsVIT2 Mutation Increases Fe and Zn of Grain without Compromising the Growth in Paddy Field. Front. Plant Sci. 2022, 13, 868661. [Google Scholar] [CrossRef] [PubMed]
4. Kumar, S.; Hash, C.; Nepolean, T.; Mahendrakar, M.; Satyavathi, C.; Singh, G.; Rathore, A.; Yadav, R.; Gupta, R.; Srivastava, R. Mapping Grain Iron and Zinc Content Quantitative Trait Loci in an Iniadi-Derived Immortal Population of Pearl Millet. Genes 2018, 9, 248. [Google Scholar] [CrossRef]
5. Semba, R.D.; Askari, S.; Gibson, S.; Bloem, M.W.; Kraemer, K. The Potential Impact of Climate Change on the Micronutrient-Rich Food Supply. Adv. Nutr. 2021, 13, 80–100. [Google Scholar] [CrossRef]
6. Yasuda, H.; Tsutsui, T. Infants and Elderlies Are Susceptible to Zinc Deficiency. Sci. Rep. 2016, 6, 21850. [Google Scholar] [CrossRef]
7. Li, X.; Wang, F.; Feng, X.; Xiao, Q.; Zheng, Q.; Xu, J.; Ma, J.; Ji, J.; Lu, S. A Nationwide Investigation of Trace Elements in Rice and Wheat Flour in China: Levels, Spatial Distributions and Implications for Human Exposure. Environ. Sci. Pollut. Res. 2023, 30, 75235–75246. [Google Scholar] [CrossRef]
8. Andersson, M. Progress Update: Crop Development of Biofortified Staple Food Crops under HarvestPlus. Afr. J. Food Agric. Nutr. Dev. 2017, 17, 11905–11935. [Google Scholar] [CrossRef]
9. Ma, J.; Qi, S.; Yuan, M.; Zhao, D.; Zhang, D.; Feng, J.; Wang, J.; Li, W.; Song, C.; Wang, T.; et al. A Genome-Wide Association Study Revealed the Genetic Variation and Candidate Genes for Grain Copper Content in Bread Wheat (Triticum aestivum L.). Food Funct. 2022, 13, 5177–5188. [Google Scholar] [CrossRef]
10. Govindan, V.; Singh, R.P.; Juliana, P.; Mondal, S.; Bentley, A.R. Mainstreaming Grain Zinc and Iron Concentrations in CIMMYT Wheat Germplasm. J. Cereal Sci. 2022, 105, 103473. [Google Scholar] [CrossRef]
11. Gupta, P.K.; Balyan, H.S.; Sharma, S.; Kumar, R. Biofortification and Bioavailability of Zn, Fe and Se in Wheat: Present Status and Future Prospects. Theor. Appl. Genet. 2020, 134, 1–35. [Google Scholar] [CrossRef] [PubMed]
12. Kaur, H.; Sharma, P.; Kumar, J.; Singh, V.K.; Vasistha, N.K.; Gahlaut, V.; Tyagi, V.; Verma, S.K.; Singh, S.; Dhaliwal, H.S.; et al. Genetic Analysis of Iron, Zinc and Grain Yield in Wheat-Aegilops Derivatives Using Multi-Locus GWAS. Mol. Biol. Rep. 2023, 50, 9191–9202. [Google Scholar] [CrossRef]
13. Zhou, Z.; Shi, X.; Zhao, G.; Qin, M.; Ibba, M.I.; Wang, Y.; Li, W.; Yang, P.; Wu, Z.; Lei, Z.; et al. Identification of Novel Genomic Regions and Superior Alleles Associated with Zn Accumulation in Wheat Using a Genome-Wide Association Analysis Method. Int. J. Mol. Sci. 2020, 21, 1928. [Google Scholar] [CrossRef]
14. Ren, P.; Zhao, D.; Zeng, Z.; Yan, X.; Zhao, Y.; Lan, C.; Wang, C. Pleiotropic Effect Analysis and Marker Development for Grain Zinc and Iron Concentrations in Spring Wheat. Mol. Breed. 2022, 42, 49. [Google Scholar] [CrossRef] [PubMed]
15. Sun, M.; Tong, J.; Dong, Y.; Pu, Z.; Zheng, J.; Zhang, Y.; Zhang, X.; Hao, C.; Xu, X.; Cao, Q.; et al. Molecular Characterization of QTL for Grain Zinc and Iron Concentrations in Wheat Landrace Chinese Spring. Theor. Appl. Genet. 2024, 137, 148. [Google Scholar] [CrossRef]
16. Bi, J.; Zeng, Z.; Li, Q.; Hong, Z.; Yan, Q.; Zhao, Y.; Wang, C. QTL Mapping and KASP Marker Development of Grain Quality-Relating Traits in Two Wheat RIL Populations. Acta Agron. Sin. 2024, 50, 669–1683. [Google Scholar] [CrossRef]
17. Li, Q.; Zeng, Z.; Zhao, Y.; Li, J.; Chen, F.; Wang, C. Genome-Wide Association Study and Linkage Mapping Reveal TaqW-6B Associated with Water-Extractable Arabinoxylan Content in Wheat Grain. Theor. Appl. Genet. 2024, 137, 166. [Google Scholar] [CrossRef]
18. Jia, Y.; Xu, N.; Zhang, J.; Ren, K.; Wu, J.; Wang, C.; Huang, M.; Li, Y. Combined Genome-Wide Association Studies (GWAS) and Linkage Mapping Identifies Genomic Regions Associated with Seedling Root System Architecture (RSA) under Different Nitrogen Conditions in Wheat (Triticum aestivum L.). Agriculture 2024, 14, 1652. [Google Scholar] [CrossRef]
19. Meng, L.; Li, H.; Zhang, L.; Wang, J. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations. Crop J. 2015, 3, 269–283. [Google Scholar] [CrossRef]
20. Ma, J.; Cao, Y.; Li, H. Genome-wide association study of ear cob diameter in maize. Acta Agron. Sin. 2021, 47, 1228–1238. [Google Scholar] [CrossRef]
21. Yang, X.; Pan, Y.; Singh, P.K.; He, X.; Ren, Y.; Zhao, L.; Zhang, N.; Cheng, S.; Chen, F. Investigation and Genome-Wide Association Study for Fusarium Crown Rot Resistance in Chinese Common Wheat. BMC Plant Biol. 2019, 19, 153. [Google Scholar] [CrossRef]
22. Lv, G.; Dong, Z.; Wang, Y.; Geng, J.; Li, J.; Lv, X.; Sun, C.; Ren, Y.; Zhang, J.; Chen, F. Identification of Genetic Loci of Black Point in Chinese Common Wheat by Genome-Wide Association Study and Linkage Mapping. Plant Dis. 2020, 104, 2005–2013. [Google Scholar] [CrossRef]
23. Liu, X.; Huang, M.; Fan, B.; Buckler, E.S.; Zhang, Z. Iterative Usage of Fixed and Random Effect Models for Powerful and Efficient Genome-Wide Association Studies. PLoS Genet. 2016, 12, e1005767. [Google Scholar] [CrossRef] [PubMed]
24. Huang, M.; Liu, X.; Zhou, Y.; Summers, R.M.; Zhang, Z. BLINK: A Package for the next Level of Genome-Wide Association Studies with Both Individuals and Markers in the Millions. Gigascience 2019, 8, giy154. [Google Scholar] [CrossRef] [PubMed]
25. Wang, Q.; Tian, F.; Pan, Y.; Buckler, E.S.; Zhang, Z. A SUPER Powerful Method for Genome Wide Association Study. PLoS ONE 2014, 9, e107684. [Google Scholar] [CrossRef] [PubMed]
26. Wang, Y.; Zeng, Z.; Li, J.; Zhao, D.; Zhao, Y.; Peng, C.; Lan, C.; Wang, C. Identification and Validation of New Quantitative Trait Loci for Spike-Related Traits in Two RIL Populations. Mol. Breed. 2023, 43, 64. [Google Scholar] [CrossRef]
27. Mccouch, S.; Cho, Y.; Yano, M.; Paul, E.; Blinstrub, M.; Morishima, H. Report on qtl nomenclature. Rice Genet. Newsl. 1997, 14, 11–13. [Google Scholar]
28. Ma, S.; Wang, M.; Wu, J.; Guo, W.; Chen, Y.; Li, G.; Wang, Y.; Shi, W.; Xia, G.; Fu, D.; et al. WheatOmics: A Platform Combining Multiple Omics Data to Accelerate Functional Genomics Studies in Wheat. Mol. Plant 2021, 14, 1965–1968. [Google Scholar] [CrossRef]
29. Welch, R.M.; Graham, R.D. Breeding for Micronutrients in Staple Food Crops from a Human Nutrition Perspective. J. Exp. Bot. 2004, 55, 353–364. [Google Scholar] [CrossRef]
30. Cakmak, I.; Torun, A.; Millet, E.; Feldman, M.; Fahima, T.; Korol, A.; Nevo, E.; Braun, H.J.; Özkan, H. Triticum dicoccoides: An Important Genetic Resource for Increasing Zinc and Iron Concentration in Modern Cultivated Wheat. Soil Sci. Plant Nutr. 2004, 50, 1047–1054. [Google Scholar] [CrossRef]
31. Manjunath, K.K.; Krishna, H.; Devate, N.B.; Sunilkumar, V.P.; Chauhan, D.; Singh, S.; Mishra, C.N.; Singh, J.B.; Sinha, N.; Jain, N.; et al. Mapping of the QTLs Governing Grain Micronutrients and Thousand Kernel Weight in Wheat (Triticum aestivum L.) Using High Density SNP Markers. Front. Nutr. 2023, 10, 1105207. [Google Scholar] [CrossRef]
32. Soriano, J.M.; Colasuonno, P.; Marcotuli, I.; Gadaleta, A. Meta-QTL Analysis and Identification of Candidate Genes for Quality, Abiotic and Biotic Stress in Durum Wheat. Sci. Rep. 2021, 11, 11877. [Google Scholar] [CrossRef] [PubMed]
33. Krishnappa, G.; Khan, H.; Krishna, H.; Kumar, S.; Mishra, C.N.; Parkash, O.; Devate, N.B.; Nepolean, T.; Rathan, N.D.; Mamrutha, H.M.; et al. Genetic Dissection of Grain Iron and Zinc, and Thousand Kernel Weight in Wheat (Triticum aestivum L.) Using Genome-Wide Association Study. Sci. Rep. 2022, 12, 12444. [Google Scholar] [CrossRef] [PubMed]
34. He, J.; Liu, F.; Wang, W.; Xing, G.; Guan, R.; Gai, J. Restricted two-stage multi-locus genome-wide association analysis and its applications to genetic and breeding studies. Sci. Agric. Sin. 2020, 53, 1704–1716. [Google Scholar] [CrossRef]
35. Xu, J.; Ni, Z.; Chen, F.; Fu, X.; Yu, F. Integrated Linkage Mapping and Genome-Wide Association Study to Dissect the Genetic Basis of Zinc Deficiency Tolerance in Maize at Seedling Stage. Crop J. 2022, 10, 1807–1818. [Google Scholar] [CrossRef]
36. Brachi, B.; Morris, G.P.; Borevitz, J.O. Genome-Wide Association Studies in Plants: The Missing Heritability Is in the Field. Genome Biol. 2011, 12, 232. [Google Scholar] [CrossRef]
37. Krishnappa, G.; Rathan, N.D.; Sehgal, D.; Ahlawat, A.K.; Singh, S.K.; Singh, S.K.; Shukla, R.B.; Jaiswal, J.P.; Solanki, I.S.; Singh, G.P.; et al. Identification of Novel Genomic Regions for Biofortification Traits Using an SNP Marker-Enriched Linkage Map in Wheat (Triticum aestivum L.). Front. Nutr. 2021, 8, 669444. [Google Scholar] [CrossRef]
38. Shi, R.; Li, H.; Tong, Y.; Jing, R.; Zhang, F.; Zou, C. Identification of Quantitative Trait Locus of Zinc and Phosphorus Density in Wheat (Triticum aestivum L.) Grain. Plant Soil 2008, 306, 95–104. [Google Scholar] [CrossRef]
39. Deng, M.; Wang, F.; Mao, C. Plant phosphate transporters and its molecular regulation mechanism. Plant Physiol. J. 2017, 53, 377–387. [Google Scholar] [CrossRef]
40. Liu, Y.; Zhao, F. Structure and mechanism of ABC transporter. Chin. Bull. Life Sci. 2017, 29, 223–229. [Google Scholar] [CrossRef]
41. Winter, D.; Vinegar, B.; Nahal, H.; Ammar, R.; Wilson, G.V.; Provart, N.J. An “Electronic Fluorescent Pictograph” Browser for Exploring and Analyzing Large-Scale Biological Data Sets. PLoS ONE 2007, 2, e718. [Google Scholar] [CrossRef] [PubMed]
Hong Z, Zeng Z, Li J, Yan X, Song J, Yan Q, Li Q, Zhao Y, Liu C, Jing X, et al. Gene Mining and Genetic Effect Analysis Reveal Novel Loci, TaZn-2DS Associated with Zinc Content in Wheat Grain. Agriculture. 2025; 15(2):124. https://doi.org/10.3390/agriculture15020124
Перевод статьи « Gene Mining and Genetic Effect Analysis Reveal Novel Loci,TaZn-2DSAssociated with Zinc Content in Wheat Grain» авторов Hong Z, Zeng Z, Li J, Yan X, Song J, Yan Q, Li Q, Zhao Y, Liu C, Jing X, et al., оригинал доступен по ссылке. Лицензия: CC BY. Изменения: переведено на русский язык

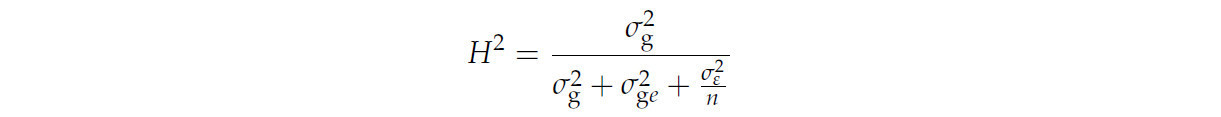
































































































Комментарии (0)