Bacillus velezensis TCS001 - новый защитник гикори: подавляет патогенную гниль и формирует полезное почвенное сообщество
Phytophthora cinnamomi вызывает серьезную корневую гниль у гикори, приводя к значительным потерям урожая. Хотя бактерии рода Bacillus признаны полезными ризосферными микроорганизмами, их применение против корневой гнили гикори и их влияние на микробные сообщества ризосферы остаются малоизученными. Данное исследование показало, что Bacillus velezensis TCS001 значимо подавлял рост P. cinnamomi ST402 in vitro и продемонстрировал 71%-ную эффективность в борьбе с корневой гнилью.
Аннотация
Сканирующая электронная микроскопия (СЭМ) выявила, что фильтрат культуральной жидкости TCS001 вызывал деформации мицелия P. cinnamomi. Анализ α- и β-разнообразия показал значительное влияние TCS001 на богатство и разнообразие бактериального сообщества ризосферы, в то время как на грибное сообщество влияние было минимальным. Более того, TCS001 изменил ко-встречаемость микробиома ризосферы гикори. Анализ дифференциальной численности предполагает, что TCS001 способствует рекрутированию полезных микроорганизмов, ассоциированных с устойчивостью к болезням, тем самым подавляя развитие заболевания. Эти результаты подчеркивают влияние TCS001 на микробиом ризосферы гикори в присутствии патогена, предоставляя ценные данные для будущих исследований и разработки эффективных биоконтрольных стратегий против корневой гнили гикори.
1. Введение
Гикори (Carya cathayensis) — распространенный питательный орех, производимый лиственным деревом с перистыми сложными листьями и орехами, принадлежащим к семейству Juglandaceae, широко произрастающий в провинциях Чжэцзян и Аньхой в Китае [1]. В настоящее время в провинции Чжэцзян, Китай, возделывается более 15 000 га гикори, что обеспечивает доход местным фермерам и способствует экологической защите в горных районах восточного Китая. Однако традиционные практики выращивания, такие как монокультура, чрезмерное удобрение и чрезмерное применение гербицидов, привели к значительным фитосанитарным проблемам [2]. В последнее время усыхающая болезнь, вызываемая Phytophthora cinnamomi, стала представлять серьезную угрозу для возделывания гикори. Было проведено различие между почвенными видами Phytophthora, которые в первую очередь вызывают потерю мелких корней, корневую и корневую шейковую гниль, а также некрозы коры, и воздушно-капельными видами Phytophthora, которые ответственны за некроз листьев, увядание побегов и гниль плодов, а также некрозы коры, в зависимости от того, происходит ли их жизненный цикл в основном над или под землей [3]. Заболевание, вызываемое P. cinnamomi, вспыхнуло в нескольких садах по всему уезду Линань, основному производственному району Китая [4]. Листья гикори желтеют, увядают и в конечном итоге опадают, что приводит к гибели растений.
P. cinnamomi — почвенный патоген, жизненный цикл которого включает как половую, так и бесполую стадии. Он может расти сапротрофно на мертвом органическом веществе, а также паразитировать на восприимчивых хозяевах. Как правило, патоген поражает мелкие боковые корни, но также может проникать в древесные стебли, особенно через раны или естественные трещины в эпидермисе. Разрастание внутри корневой системы может привести к корневой гнили, нарушая поглощение и транспорт воды к ветвям, что приводит к увяданию и пожелтению листьев. Растения могут погибнуть быстро или выживать в течение многих лет, но симптомы болезни часто не проявляются. Способность P. cinnamomi к сапротрофному росту в почве или бессимптомному существованию в зараженных растениях является ключевым фактором его долгосрочного выживания. Пораженные растения демонстрировали некротические симптомы на той же стороне основания стебля, что и опавшие листья, что приводило к значительным потерям урожая и серьезно сказывалось на доходах местных фермеров и экологической безопасности.
Ризосферный микробиом предоставляет растениям многочисленные преимущества, включая улучшенное усвоение питательных веществ, устойчивость к стрессам и подавление болезней [5,6]. Микроорганизмы, ассоциированные с растениями, все чаще признаются за их ключевую роль в устойчивости к болезням [7,8,9]. Это влияние опосредовано такими механизмами, как микробная конкуренция и антагонизм по отношению к патогенам [10,11,12], а также рекрутирование полезных микробов для усиления колонизации корней и смягчения атак патогенов [13]. Растущий интерес к ризосферным микробным сообществам отражает все большее внимание к экологическому сельскому хозяйству и устойчивому развитию. Среди полезных бактерий Bacillus velezensis стал ключевым объектом исследований благодаря своему значительному влиянию на здоровье растений и устойчивость к болезням [14,15]. Надежная способность B. velezensis к спорообразованию обеспечивает выживание в различных средах, в то время как производство различных метаболитов, включая антибиотики и противогрибковые соединения, способствует повышению устойчивости растений к болезням [16,17]. Хотя потенциал биоконтроля и эффекты, способствующие росту растений, у видов Bacillus хорошо документированы, влияние B. velezensis на усыхающую болезнь гикори и его воздействие на ризосферный микробиом остаются неисследованными.
Микробные инокулянты оказывают свое влияние не только напрямую на растения-хозяева, но и косвенно, модулируя состав почвенного микробиома, тем самым способствуя пролиферации полезных микробов и внося вклад в подавление болезней и стимуляцию роста. В данном исследовании мы подтвердили противогрибковый эффект B. velezensis TCS001 против патогена Phytophthora cinnamomi ST402 путем обработки растений гикори TCS001. Мы также проанализировали влияние TCS001 на микробное сообщество в ризосфере гикори с помощью высокопроизводительного секвенирования ризосферной почвы, предоставив ценные данные для будущей разработки агентов биоконтроля против патогенов, таких как Phytophthora, у важных лесных культур, таких как гикори.
2. Материалы и методы
2.1. Растительные материалы, патоген и отбор почвы
Саженцы гикори выращивали в теплице при дневных/ночных температурах 24 °C в течение 16 ч/сутки при влажности 80% в теплице. Bacillus velezensis TCS001 был выделен и идентифицирован в нашей лаборатории (CGMCC № 8921).
Phytophthora cinnamomi ST402 (получен от проф. Юнцзюнь Вана, Чжэцзянский университет сельского и лесного хозяйства) использовался в данном исследовании. Phytophthora cinnamomi ST402 культивировали на агаре V8 (3 г/л CaCO3, 100 мл/л 100% овощного сока V8, 20 г/л агар) при 25 °C в темноте. Рост мицелия и фенотипические изменения оценивали на чашках Петри с агаром V8 через семь дней.
После посева штрихами штамма B. velezensis TCS001 на агар LB и инкубации в течение 48 ч была отобрана отдельная колония и инокулирована в 80 мл посевной среды MLB (7 г/л пептон, 2 г/л дрожжевой экстракт, 6 г/л NaCl, 2 г/л глюкоза, 0,06 г/л KCl, 0,5 г/л MgCl2·6H2O) и инкубирована при 27 °C и 145 об/мин в течение 16 ч. Эта посевная культура впоследствии использовалась в качестве 3% инокулята для среды производства липопептидов (10,5 г/л растворимый крахмал, 18,5 г/л арахисовая мука, 3 г/л NaCl, 32% об./об., pH 7,0) и инкубировалась при 31 °C и 164 об/мин в течение 48 ч. Полученную культуральную жидкость центрифугировали (7830 об/мин, 4 °C, 30 мин), а затем четыре раза стерильно фильтровали (фильтр 0,22 мкм) с использованием стерильного шприца и фильтровальной мембраны для получения супернатанта.
Супернатант вносили в расплавленный картофельно-декстрозный агар (PDA) при температуре 45–50 °C, достигая конечной концентрации 20% (об./об.). Мицелиальный диск диаметром 5 мм от каждого патогенного штамма помещали в центр чашек Петри с PDA, содержащим бактериальный фильтрат. Затем чашки инкубировали при 25 °C в течение пяти дней. Для сравнения готовили контрольные чашки без добавления бактериального фильтрата. Каждое экспериментальное условие было продублировано трижды для обеспечения надежности. Ингибирующий эффект количественно оценивали по следующей формуле: y = (A − B)/A × 100% (A: радиус роста патогена в контроле; B: радиус роста патогена в различных обработках).
Для контроля неоднородности почвы использовали смесь автоклавированного питательного субстрата и высушенной на воздухе почвы (<4 мм). У саженцев Carya равномерно промывали корни стерильной водой перед пересадкой в горшки объемом 0,5 л, заполненные стерилизованным почвенным субстратом. Через неделю после пересадки контрольная группа (CK) получала стерильную воду, а группа обработки (T6) получала корневой полив суспензией B. velezensis TCS001 (1 × 106 КОЕ/мл). Объем полива как стерильной воды, так и суспензии TCS001 составлял 200 мл. Еще через неделю обе группы инокулировали P. cinnamomi. Для инокуляции Phytophthora cinnamomi использовали метод полива спорами. Чашки Петри с Phytophthora cinnamomi на V8, культивируемые в темноте при 22 °C в течение 3–5 дней, замачивали в 500 мл ddH2O. Воду заменяли каждые 12 часов для стимулирования образования спорангиев. Стерильные зубочистки использовали для соскабливания гиф со среды V8, содержащей спорангии, в 500 мл ddH2O, что служило поливным раствором. Каждый горшок с гикори инокулировали поливом 200 мл суспензии спор. Заболеваемость = количество зараженных растений / общее количество растений × 100%.
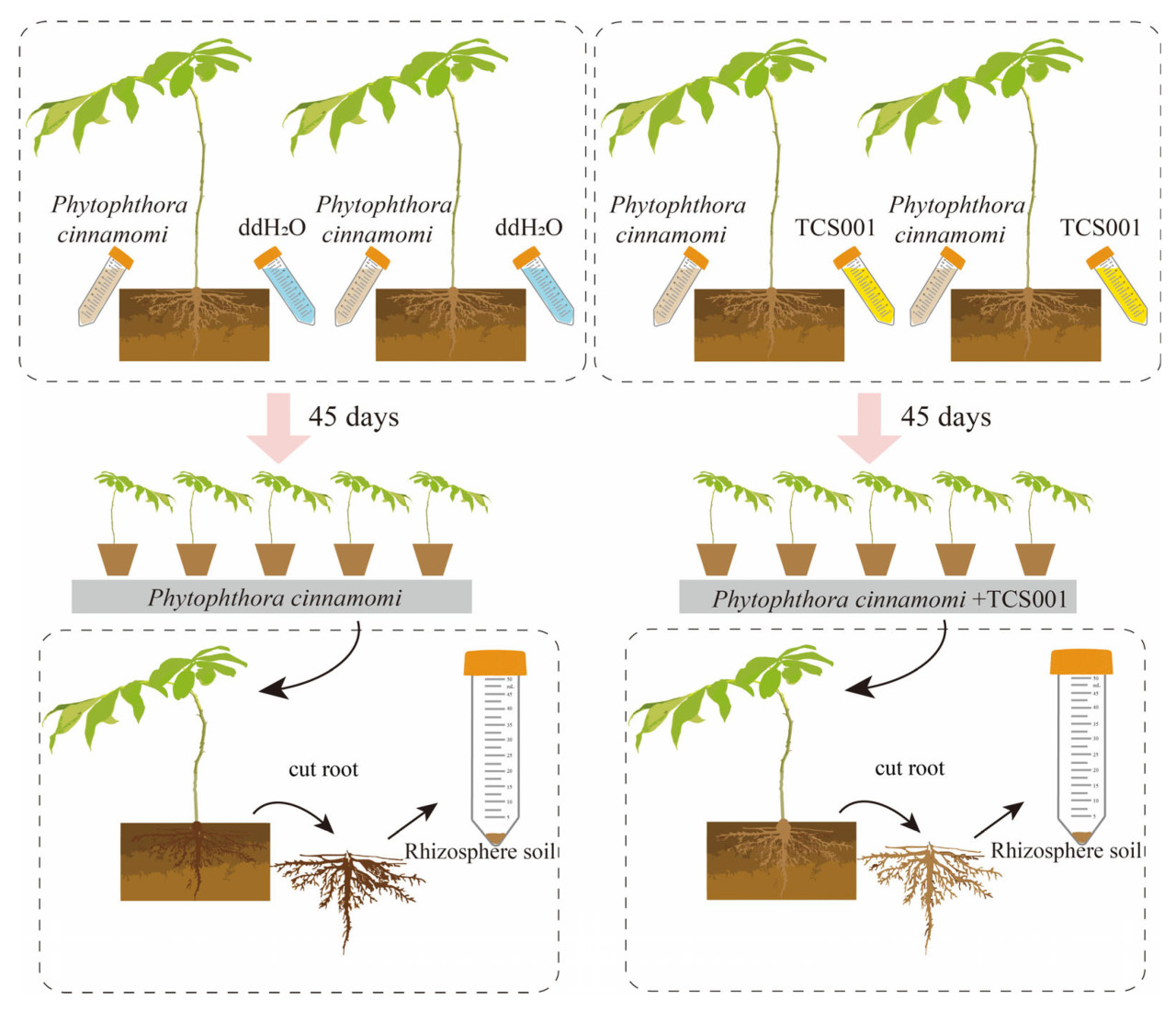
Саженцы выращивали в теплице с регулируемыми параметрами окружающей среды, включая температуру 24 °C, 16-часовой световой цикл и влажность 80%. Их поливали стерильной дважды дистиллированной водой (ddH2O). Стратегия полива заключалась в поддержании поверхностного слоя почвы в горшках во влажном состоянии. Образцы ризосферной почвы собирали через 45 дней после инокуляции Phytophthora cinnamomi. После тщательного удаления поверхностной почвы растения аккуратно выкапывали. Любую почву, прилипшую к корням, затем счищали стерильной кистью. Образцы ризосферной почвы помещали в полипропиленовые пробирки объемом 50 мл, быстро замораживали с помощью жидкого азота и затем хранили при −80 °C. Каждое экспериментальное условие включало три биологических повторности, по пять растений в каждом повторении (Рисунок 1).
Рисунок 1. Схематическая диаграмма. Растения контрольной группы (CK) получали полив стерильной водой, в то время как растения группы обработки (T6) поливали суспензией B. velezensis TCS001. Через семь дней после полива обе группы инокулировали в корни P. cinnamomi ST402. Ризосферную почву отбирали для анализа микробиома через 45 дней после инокуляции.
2.2. Экстракция ДНК, ПЦР-амплификация и высокопроизводительное секвенирование
Геномную ДНК экстрагировали из шести образцов почвы с использованием набора E.Z.N.A.® Soil DNA Kit (Omega Bio-tek, Гуанчжоу, Китай) в соответствии с протоколом производителя. Качество и концентрацию ДНК определяли с помощью электрофореза в 1% агарозном геле и спектрофотометрии NanoDrop® ND-2000 (Thermo Scientific, Уилмингтон, Северная Каролина, США) перед хранением при −80 °C. Ампликоны бактериальной 16S рДНК и грибной ITS рДНК получали с использованием пар праймеров (5′-ACTCCTACGGGAGGCAGCAG-3′) и 806R (5′-GGACTACHVGGGTWTCTAAT-3′), а для гена ITS рДНК амплификацию проводили с праймерами ITS1-F (CTTGGTCATTTAGAGGAAGTAA) и ITS4-R (TCCTCCGCTTATTGATATGC) на термоциклере для ПЦР ABI GeneAmp® 9700 (Thermo Fisher Scientific, Юджин, Орегон, США). Каждая реакция ПЦР объемом 20 мкл содержала 4 мкл 5× буфера Fast Pfu, 2 мкл 2,5 мМ dNTP, по 0,8 мкл каждого праймера (5 мкМ), 0,4 мкл полимеразы Fast Pfu, 10 нг матричной ДНК и ddH2O. Условия термического циклирования были следующими: 95 °C в течение 3 минут, затем 27 циклов по 95 °C в течение 30 с, 55 °C в течение 30 с и 72 °C в течение 45 с, с окончательной элонгацией при 72 °C в течение 10 минут и выдерживанием при 4 °C. Все образцы амплифицировали в трехкратной повторности. Продукты ПЦР экстрагировали из геля (2% агароза) с использованием набора AxyPrep DNA Gel Extraction Kit (Axygen Biosciences, Ханчжоу, Китай) и количественно определяли с помощью флуорометра Quantus™ (Promega, Пекин, Китай).
Очищенные ампликоны секвенировали на платформе Illumina NovaSeq PE250 компанией Majorbio Bio-Pharm Technology Co. Ltd. (Шанхай, Китай) с использованием стандартных протоколов. Необработанные данные были депонированы в архиве чтений последовательностей NCBI (SRA).
2.3. Обработка данных и биоинформатический анализ
Необработанные файлы FASTQ демультиплексировали с помощью пользовательского скрипта Perl, разработанного собственными силами. Фильтрацию по качеству и объединение парных чтений проводили с использованием fastp v0.19.6 и FLASH v1.2.7 соответственно, применяя следующие критерии: (i) чтения обрезали при средних показателях качества <20 в скользящем окне 50 п.н., и те, которые были <50 п.н., отбрасывали, вместе с чтениями, содержащими неоднозначные основания; (ii) перекрывающиеся последовательности ≥10 п.н. с максимальным коэффициентом несоответствия 0,2 в области перекрытия объединяли; (iii) образцы демультиплексировали на основе последовательностей штрих-кода и праймера (точное совпадение штрих-кода, ≤2 несоответствия нуклеотидов в праймере). Операционные таксономические единицы (OTU) были получены из проанализированных последовательностей с использованием UPARSE v7.1 [18,19] с порогом сходства последовательностей 97%. Наиболее часто встречающуюся последовательность в каждой OTU выбирали в качестве ее представительной. Для нормализации глубины секвенирования последовательности гена 16S рРНК разрежали до 44 980 чтений, а последовательности ITS рДНК — до 84 995 чтений, что привело к среднему охвату Гуда 99,09%.
Таксономическую классификацию репрезентативных последовательностей OTU проводили с помощью RDP Classifier v2.2 по базе данных SILVA (v138.1 для бактерий) и базе данных UNITE (v138.1 для грибов) с порогом достоверности 0,7. Функциональный потенциал метагенома предсказывали с использованием PICRUSt2 (Phylogenetic Investigation of Communities by Reconstruction of Unobserved States) [20]. Анализ использовал его интегрированный конвейер, который включает HMMER для выравнивания последовательностей, EPA-NG и Gappa для филогенетического размещения, castor для нормализации числа копий 16S гена и MinPath для предсказания семейств генов и путей. Все этапы соответствовали стандартному протоколу PICRUSt2.
Биоинформатический анализ микробиоты почвы проводили на платформе Majorbio Cloud (https://cloud.majorbio.com). На основе информации об OTU рассчитывали кривые разрежения и индексы альфа-разнообразия, включая наблюдаемые OTU, богатство Chao1, индекс Шеннона и охват Гуда, с помощью Mothur v1.30.1 [21]. Сходство между микробными сообществами в разных образцах определяли с помощью анализа главных координат (PCoA) на основе несходства Брея-Кертиса с использованием пакета Vegan v2.5-3. Для расчета значимости различий между двумя обработками использовали t-критерий Уэлча с помощью STAMP. Вариации относительной численности OTU в различных обработках анализировали с помощью критериев отношения правдоподобия в пакете "EdgeR". Для визуализации создавали диаграмму Манхэттена с использованием пакета "ggplot2".
2.4. Анализ ко-встречаемости сети
Сети ко-встречаемости для бактериальных и грибных сообществ в контрольной (CK) и обработанной (T6) группах были построены для исследования изменений во взаимодействиях микробных сообществ. Сети генерировали с помощью R (v4.3.1) на основе коэффициентов корреляции Спирмена; включали только значимые (p < 0,05) корреляции с |R| > 0,6 [22]. Для визуализации сети использовали макет Фрюхтермана-Рейнгольда в Gephi.
2.5. Наблюдение с помощью сканирующей электронной микроскопии (СЭМ)
Свежий мицелий P. cinnamomi ST402 на V8, обработанный фильтратом культуральной жидкости TCS001, собирали и тщательно фиксировали в 2,5% глутаральдегиде в фосфатном буфере в течение как минимум 4 ч. Затем образцы обрабатывали и визуализировали с помощью просвечивающего электронного микроскопа (Hitachi H-7650) в Чжэцзянском университете.
3. Результаты
3.1. Биоконтрольный эффект TCS001 на болезни гикори
Bacillus velezensis TCS001 и Phytophthora cinnamomi ST402 использовали для оценки эффективности биоконтроля. Двухкультуральный тест продемонстрировал значительное ингибирование роста P. cinnamomi ST402 с помощью TCS001 (Рисунок 2A). Дополнительно, эксперименты на растениях (Рисунок 1) показали, что корневой полив TCS001 (обработка T6) значительно снизил заболеваемость (63%) в присутствии P. cinnamomi ST402 по сравнению с неинокулированным контролем (CK), где заболеваемость составляла более 86,5% (Рисунок 2B). Больные листья желтеют, а их корни загнивают, становясь черными (Рисунок 2C). Эти данные продемонстрировали эффективный биоконтроль вызываемой P. cinnamomi ST402 болезни гикори с помощью TCS001 (Рисунок 2C,D и Рисунок S1).
Рисунок 2. Эффективность биоконтроля B. velezensis TCS001 против P. cinnamomi ST402. (A) TCS001 ингибировал рост Phytophthora cinnamomi ST402. (B) Уровень заболеваемости для ST402 и ST402+TCS001. Рост гикори при различных обработках. Разные заглавные буквы указывают на значительные различия между показателями (p < 0,01, t-критерий). (C) Контрольная группа была инокулирована только Phytophthora cinnamomi ST402. (D) Корни поливали бактериальным раствором TCS001 и впоследствии инокулировали Phytophthora cinnamomi ST402.
3.2. Влияние фильтрата культуральной жидкости TCS001 на рост Phytophthora cinnamomi
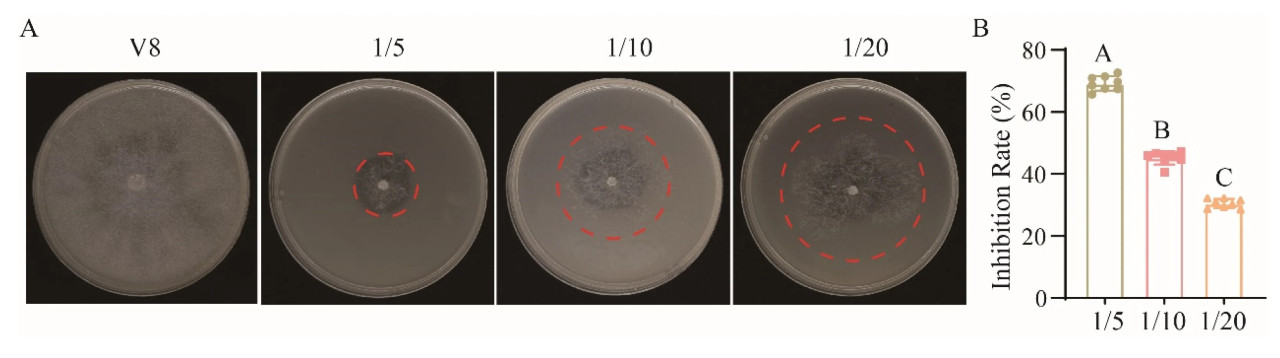
Противогрибковую активность различных концентраций фильтрата культуральной жидкости TCS001 против P. cinnamomi ST402 оценивали на среде V8 (Рисунок 3A). Как показано на Рисунке 3, различные концентрации фильтрата культуральной жидкости оказывают различное ингибирующее действие на патоген P. cinnamomi ST402 со значениями 68%, 49% и 36% соответственно (Рисунок 3B). Кроме того, оптимальным условием обработки фильтратом культуральной жидкости TCS001 было определено соотношение разбавления жидкость:вода 1:5. Результаты показали, что фильтрат культуральной жидкости TCS001 может ингибировать рост и развитие Phytophthora cinnamomi.
Рисунок 3. Тест на ингибирующую активность против Phytophthora cinnamomi ST402. (A) Противогрибковая активность фильтрата культуральной жидкости TCS001 in vitro оценивалась при серийных коэффициентах разбавления 1/5, 1/10 и 1/20 (фильтрат культуральной жидкости к среде). (B) Рассчитывали процент ингибирования для чашек Петри, обработанных фильтратом культуральной жидкости TCS001 при указанных разведениях. Значительные различия между показателями обозначены разными заглавными буквами (p < 0,01, t-критерий).
3.3. Наблюдение с помощью сканирующей электронной микроскопии (СЭМ) гиф, культивированных на среде, разбавленной в пять раз фильтратом культуральной жидкости
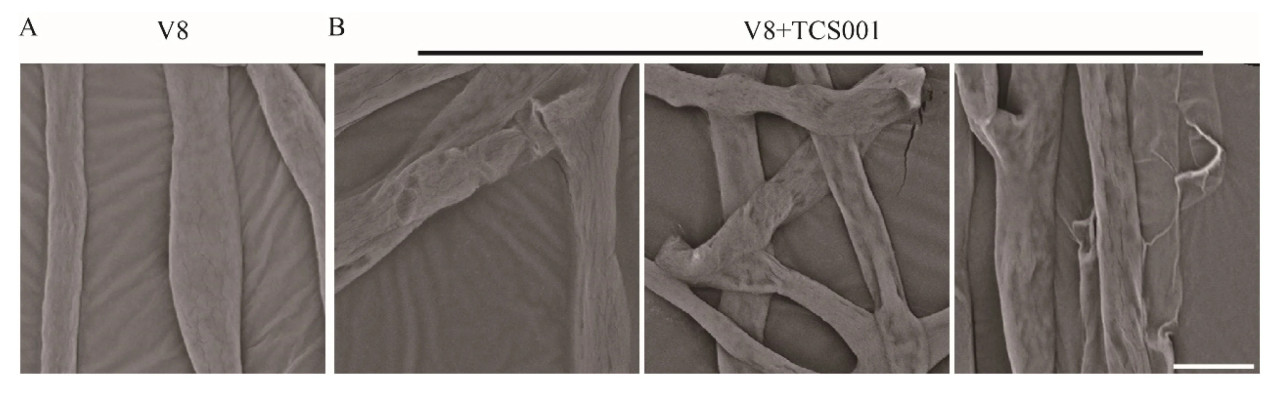
Сканирующую электронную микроскопию (СЭМ) использовали для исследования гиф P. cinnamomi ST402, выращенных на среде V8, содержащей пятикратное разведение фильтрата культуральной жидкости TCS001. Контрольные гифы имели гладкую поверхность (Рисунок 4A), в то время как гифы, обработанные фильтратом TCS001, демонстрировали разрывы, скручивание и деформацию (Рисунок 4B). Эти данные свидетельствовали о том, что фильтрат культуральной жидкости TCS001 изменял морфологию гиф P. cinnamomi ST402.
Рисунок 4. Морфологические изменения мицелия P. cinnamomi, выращенного на V8 с добавлением фильтрата культуральной жидкости TCS001. (A) контроль; (B) группа обработки.
3.4. Характеристика наборов данных ампликонного секвенирования
Чтобы изучить влияние B. velezensis TCS001 на ризосферный микробиом гикори, было проведено высокопроизводительное секвенирование шести образцов ризосферной почвы. Кривые разрежения для ампликонов гена 16S рРНК и ITS показали достаточную глубину секвенирования для охвата разнообразия сообщества (Рисунок S1). Всего было получено 379 897 высококачественных последовательностей 16S рРНК (от 59 430 до 69 804 на образец; средняя длина 412,40 п.н.) и 559 998 высококачественных последовательностей ITS (от 85 193 до 97 923 на образец; средняя длина 241,52 п.н.) (Таблица S1). Таксономическую принадлежность определяли с использованием баз данных SILVA (бактерии) и UNITE (грибы) с порогом сходства 97%, используя байесовский классификатор и BLAST. В результате было получено 1283 бактериальных и 522 грибных OTU, представляющих 35 бактериальных и 15 грибных типов соответственно. Дополнительный анализ был сосредоточен на OTU с относительной численностью >0,05%, в результате чего было получено 401 бактериальная и 474 грибных OTU. Эти данные использовали для оценки влияния TCS001 на ризосферный микробиом гикори.
3.5. CK и T6 имеют значительно различающиеся ризосферные микробные сообщества
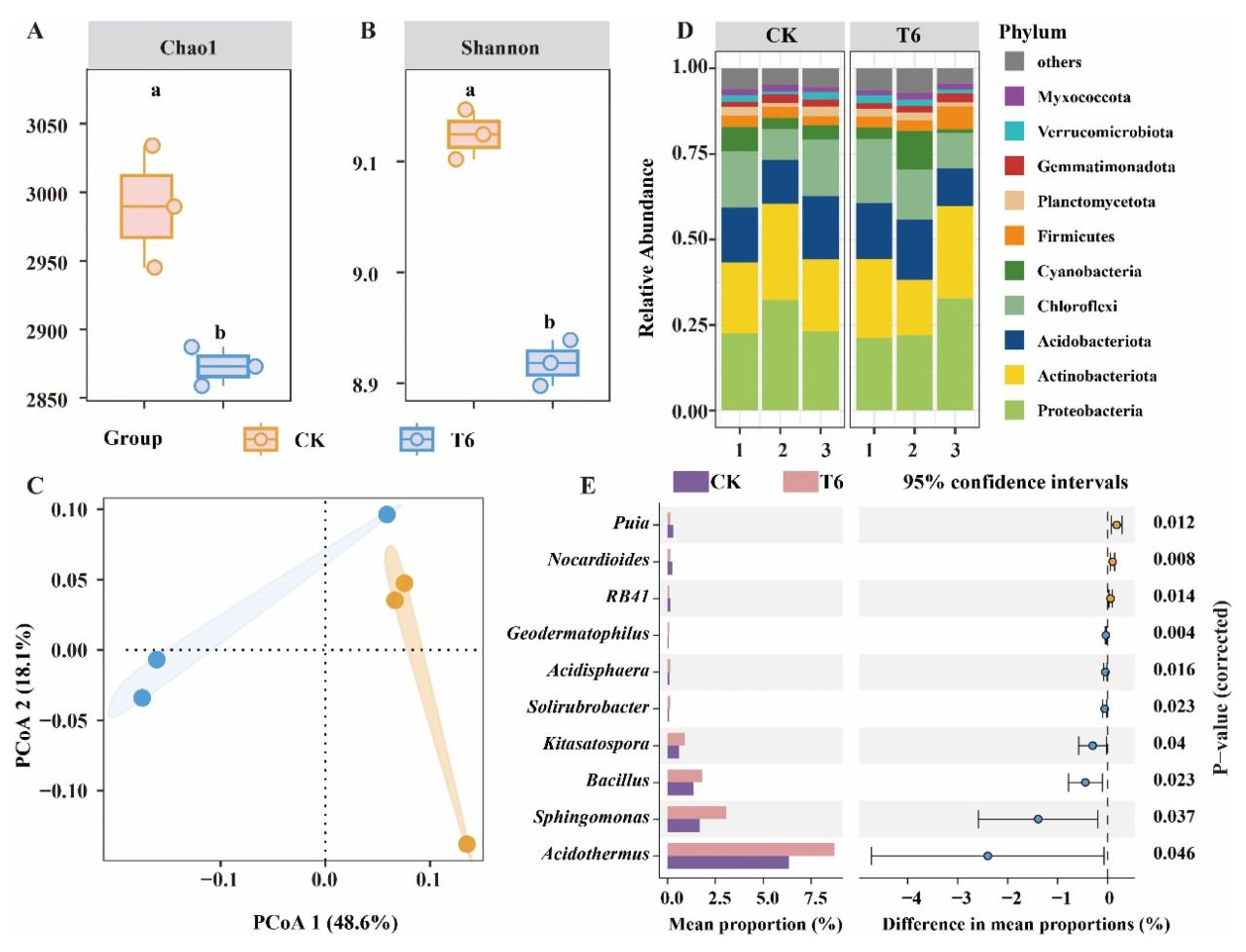
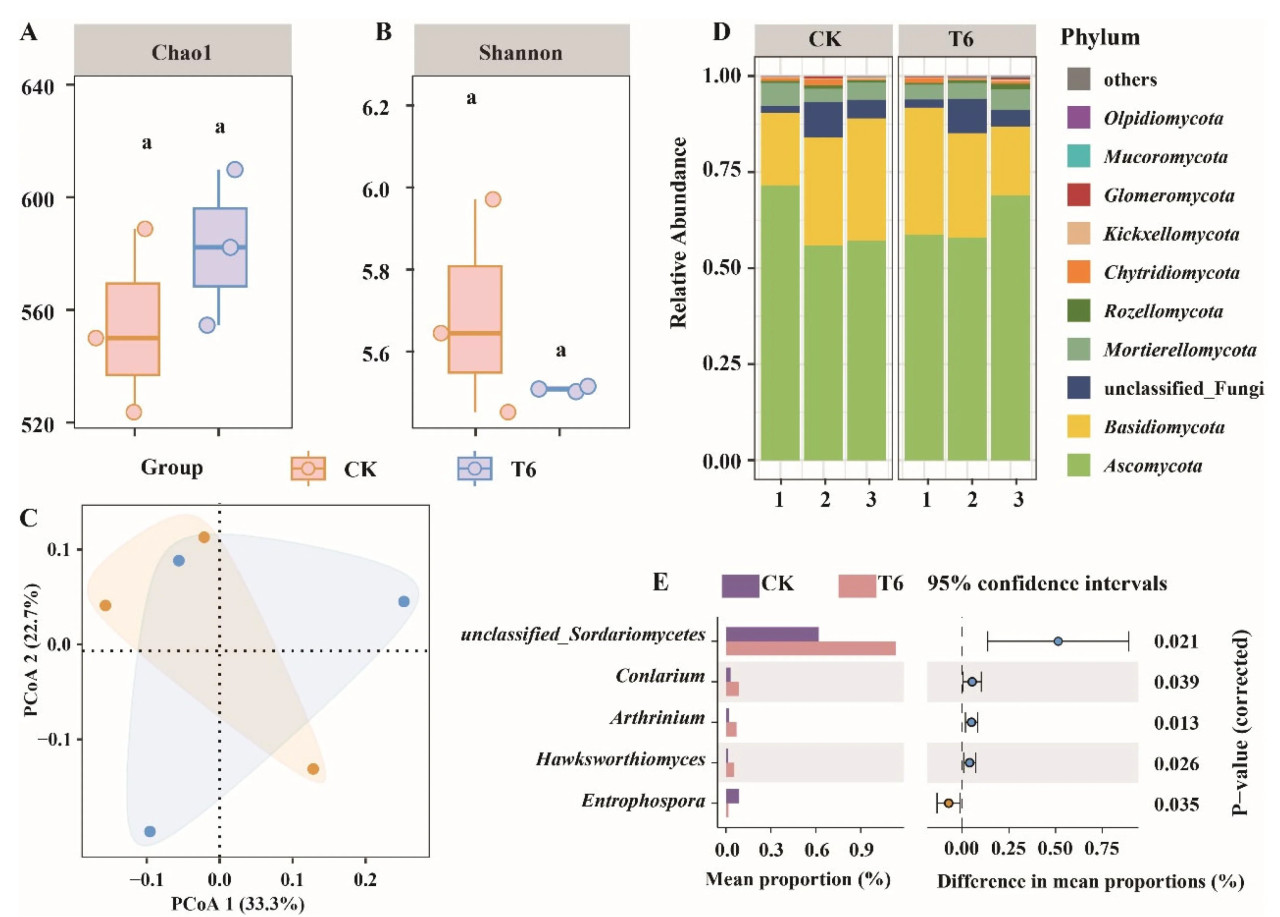
Чтобы охарактеризовать влияние TCS001 на изменения в ризосферных микробных сообществах гикори после инокуляции патогенным грибом P. cinnamomi, альфа-разнообразие грибных и бактериальных сообществ анализировали с использованием индекса Chao1 и индекса Шеннона. Наши анализы показали, что после инокуляции P. cinnamomi TCS001 оказал значительное влияние на разнообразие и богатство бактериального сообщества гикори (p < 0,05), но не оказал значительного эффекта на грибное сообщество (Рисунок 5 и Рисунок 6). Бактериальный индекс Chao1 и индекс Шеннона в CK были значительно выше, чем в ризосферной почве, обработанной TCS001 (p < 0,05) (Рисунок 5A,B). Для грибного сообщества индекс Chao ризосферной почвы группы CK был ниже, чем у группы T6, в то время как индекс Шеннона показал противоположную тенденцию, хотя это изменение не было значительным (p > 0,05) (Рисунок 6A). Кроме того, анализ главных координат (PCoA) на основе расстояния Брея-Кертиса дополнительно выявил значительные различия в ризосферных микробных сообществах гикори из-за TCS001 (Рисунок 6B). Мы обнаружили, что образцы, инокулированные патогенным грибом P. cinnamomi, можно было отличить от образцов, инокулированных патогенным грибом P. cinnamomi и TCS001, в бактериальных сообществах (Рисунок 5C), в то время как в грибных сообществах шесть образцов сгруппировались вместе и не могли быть различимы (Рисунок 6C). Этот результат в сочетании с результатами индексов Chao и Шеннона показал, что TCS001 значительно влияет на состав ризосферного бактериального сообщества.
Рисунок 5. Анализ альфа- и бета-разнообразия бактерий, а также состава микробиома. Альфа-разнообразие, оцененное с использованием индексов Chao (A) и Шеннона (B), продемонстрировало вариацию среди образцов ризосферной почвы при различных обработках, причем значительные различия обозначены разными строчными буквами (p < 0,05, критерий Тьюки). Анализ главных координат (PCoA) на основе матрицы расстояний Брея-Кертиса (C) выявил различные паттерны кластеризации. Относительная численность бактериальных типов в образцах ризосферной почвы из обработанных растений отображена на (D). Значительные различия на уровне бактериальных типов были выявлены с использованием критерия Крускала-Уоллиса H (E). (Строчные буквы обозначают значительные различия).
Рисунок 6. Грибное разнообразие и состав микробиома были проанализированы в различных образцах ризосферной почвы. Грибное альфа-разнообразие, оцененное с использованием индексов Chao (A) и Шеннона (B), показало значительную вариацию между обработками, причем разные строчные буквы обозначают значительные различия (p < 0,05, критерий Тьюки). Анализ главных координат (PCoA) на основе матрицы расстояний Брея-Кертиса (C) продемонстрировал различные паттерны кластеризации. Относительная численность грибных типов в образцах ризосферной почвы из различных обработок отображена на (D). Критерий Крускала-Уоллиса H (E) выявил значительные различия в грибных типах. (Строчные буквы обозначают значительные различия).
Мы дополнительно оценили относительную численность бактерий как на уровне типа, так и на уровне рода. Состав бактериального сообщества исследовали на этих таксономических уровнях. Proteobacteria, Actinobacteriota и Acidobacteriota были доминирующими типами как в контрольной (CK), так и в обработанной (T6) группах (Рисунок 5D; Таблица S2) с аналогичной относительной численностью: Proteobacteria (CK: 26,04%; T6: 25,36%), Actinobacteriota (CK: 23,28%; T6: 22,12%) и Acidobacteriota (CK: 15,81%; T6: 14,98%). На уровне рода t-критерий Уэлча выявил значительные различия (p < 0,05) в относительной численности десяти родов между CK и T6 (Рисунок 5E). Семь родов показали значительное увеличение численности в T6 по сравнению с CK (Рисунок 5E), включая Acidothermus, Sphingomonas и Bacillus (увеличение на 2,40%, 1,39% и 0,45% соответственно; Таблица S2).
В грибном сообществе Ascomycota и Basidiomycota были доминирующими типами как в контрольной (CK), так и в обработанной (T6) группах (Рисунок 6D; Таблица S2) с аналогичной относительной численностью: Ascomycota (CK: 61,52%; T6: 61,89%) и Basidiomycota (CK: 26,30%; T6: 26,10%). Хотя Ascomycota показала немного более высокую относительную численность в T6, это различие не было статистически значимым (p > 0,05). На уровне рода пять грибных родов показали значительные различия между CK и T6 (Рисунок 6E). Unclassified Sordariomycetes, Conlarium, Arthrinium и Hawksworthiomyces продемонстрировали значительно более высокую относительную численность в CK (увеличение на 0,47%, 0,02%, 0,08% и 0,04% соответственно; Рисунок 6E; Таблица S2), в то время как Entrophospora показала значительно более высокую относительную численность в T6 (увеличение на 0,02%). Эти результаты указывают на то, что TCS001 изменяет структуру бактериальных и грибных сообществ в ризосфере гикори.
3.6. Специфические различия в микробиомах ризосферной почвы
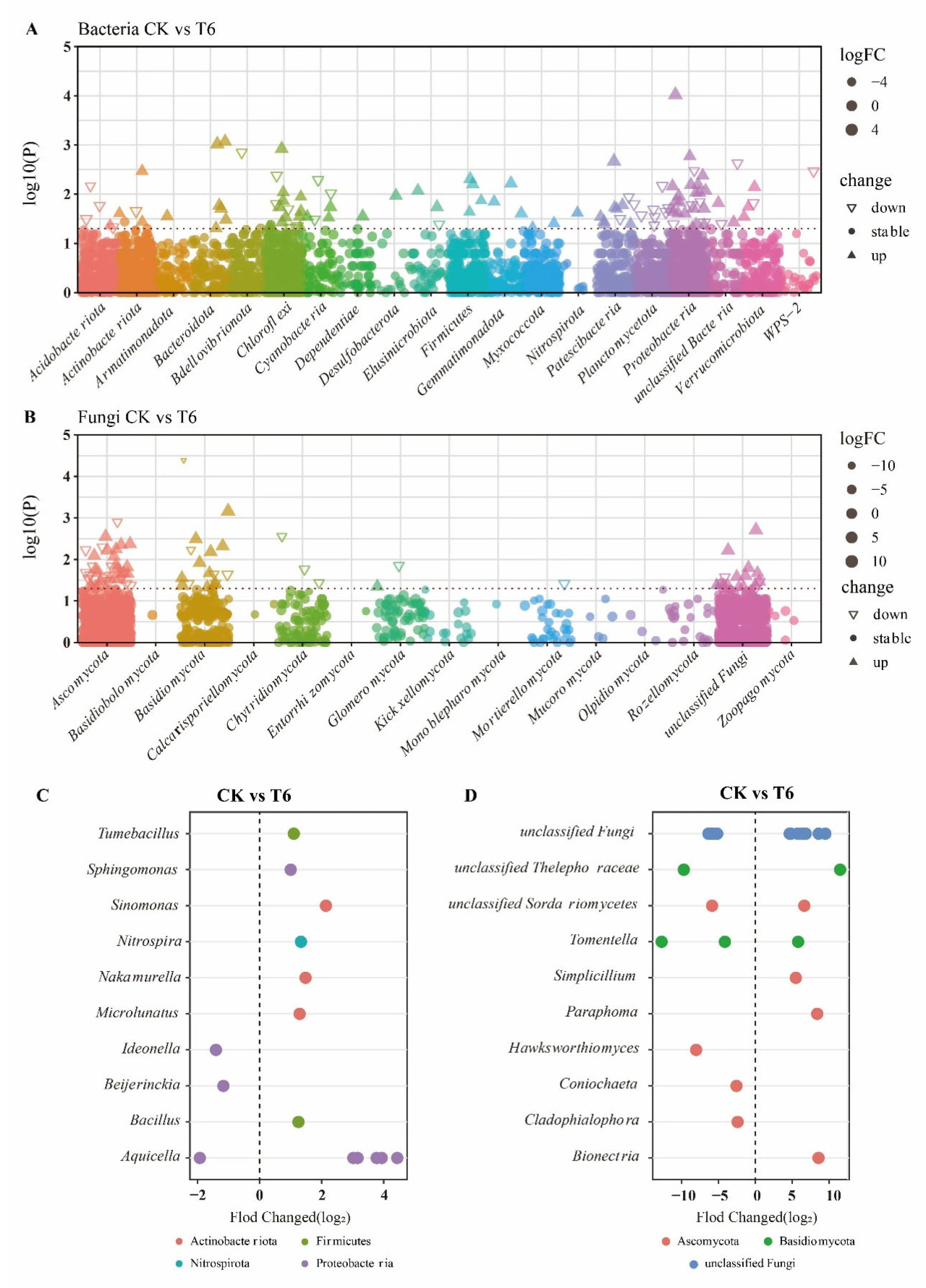
Чтобы дальнейшим образом охарактеризовать сдвиги в микробных сообществах, мы сравнили численность OTU между контрольными (CK; инокулированные патогеном) и обработанными (T6; инокулированные патогеном и TCS001) образцами ризосферы гикори с использованием диаграмм Манхэттена и точечных диаграмм. Были идентифицированы значительные различия в численности OTU (p < 0,05; Рисунок 7A,B; Таблицы S4 и S5): 144 бактериальных OTU и 81 грибная OTU. Они принадлежали к 20 бактериальным типам (Таблица S3), причем Proteobacteria (33 OTU), Chloroflexi (25 OTU) и Acidobacteriota (15 OTU) показали наиболее значительные различия. 81 дифференциально обильная грибная OTU представляла шесть известных типов и один неклассифицированный тип (Таблица S3), причем Ascomycota (39 OTU), Basidiomycota (17 OTU) и неклассифицированные грибы (16 OTU) продемонстрировали наибольшие различия (Таблица S4). T6 показал обогащение 72 бактериальными и 48 грибными OTU по сравнению с CK (Таблицы S3 и S4), в то время как топ-10 дифференциально обильных OTU включало больше грибов, чем бактерий (32 против 16; Рисунок 7C,D), что предполагает, что бактерии могут содержать больше низкообильных дифференциально обильных OTU. Примечательно, что потенциальный патоген Aspergillus (OTU1300) был значительно менее обильным в T6 (Таблица S4), в то время как полезные бактерии, такие как Sphingomonas (OTU3182) и Bacillus (OTU2812), были обогащены в T6 (Таблица S3). Эти результаты указывают на то, что совместная инокуляция с TCS001 способствует рекрутированию полезных микробов в ризосфере в ответ на патогенную угрозу.
Рисунок 7. Обогащение (вверх) и обеднение (вниз) бактериальных (A) и грибных (B) OTU между группами CK и T6. Сравнительный анализ 10 лучших бактериальных (C) и грибных (D) родов, показывающих значительное обогащение или обеднение OTU между группами обработок CK и T6. Цвета точек указывают на классификацию типа.
3.7. Характеристика сетей ко-встречаемости ризосферного микробиома
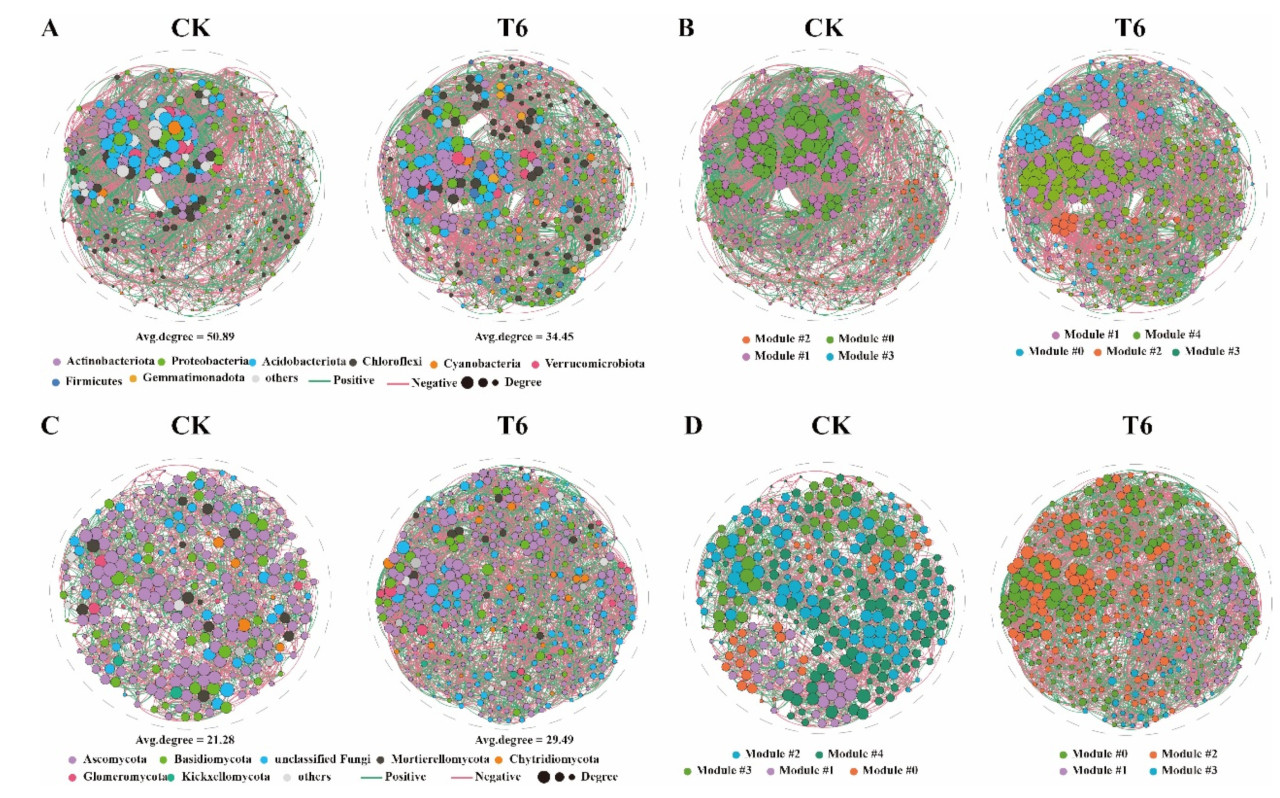
Чтобы исследовать влияние TCS001 на сети ко-встречаемости микробов ризосферы гикори, были построены бактериальные и грибные сети (Рисунок 8). Мы проанализировали долю графиков грибной и бактериальной сети на уровне типа. В бактериальной сети ко-встречаемости по сравнению с CK доля Actinobacteriota, Chloroflexi, Gemmatimonadota, Firmicutes, Myxococcota и WPS-2 увеличилась в группе T6. Аналогично, в грибной сети после инокуляции TCS001 доля пяти грибных типов (неклассифицированные грибы, Chytridiomycota, Kickxellomycota, Rozellomycota и Glomeromycota) увеличилась, и ранжирование восьми бактериальных типов и семи грибных типов также претерпело изменения (Рисунок 8A,C, Таблица S5).
Рисунок 8. Бактериальные и грибные сети ко-встречаемости. Бактериальные (A,B) и грибные (C,D) сети ко-встречаемости были построены с использованием коэффициента корреляции Спирмена на уровне OTU, отображая связи, которые представляют статистически значимые корреляции (p < 0,05) с величинами > 0,6 (положительная корреляция — синие ребра) или <−0,6 (отрицательная корреляция — фиолетовые ребра). Разные цвета соответствуют разным типам (A,C) и классам модулей (B,D).
Мы также проанализировали топологию сетей ко-встречаемости. Контрольная (CK) бактериальная сеть состояла из 377 узлов и 9440 ребер, в то время как грибная сеть состояла из 299 узлов и 3182 ребер (Таблица S6). После инокуляции TCS001 (T6) бактериальная сеть имела 367 узлов и 6371 ребро, а грибная сеть — 437 узлов и 6444 ребра (Таблица S6). Эти изменения предполагают более сильную связность внутри CK бактериальной сети по сравнению с T6 бактериальной сетью, и противоположную тенденцию для грибных сетей. Бактериальная сеть T6 показала более высокую долю положительных ребер (CK: 52,87%; T6: 56,34%), а грибная сеть T6 продемонстрировала более высокую среднюю степень (CK: 21,28; T6: 29,49; Рисунок 8C, Таблица S6). И бактериальные, и грибные сети показали снижение плотности после инокуляции TCS001 (бактериальные: CK 0,138, T6 0,094; грибные: CK 0,071, T6 0,068; Таблица S6). Мы также обнаружили, что после инокуляции антагонистической бактерией TCS001 модульность бактериальной сети стала более концентрированной. В бактериальной сети группа CK имела пять модулей, группа T6 имела четыре модуля, а грибная сеть была прямо противоположна бактериальной (Рисунок 8B,D). Эти данные демонстрируют, что применение TCS001 изменяет структуру микробной сети ризосферы.
4. Обсуждение
Представители рода Bacillus широко признаны за их потенциал в качестве агентов биоконтроля благодаря их высокому росту, стабильным физико-химическим свойствам и широкому антимикробному спектру [23]. Многочисленные исследования продемонстрировали способность различных штаммов Bacillus способствовать росту растений и подавлять болезни, подчеркивая их универсальность в различных сельскохозяйственных системах. Например, Bacillus amyloliquefaciens FZB42 и FZB24 были тщательно изучены на предмет их способности подавлять болезни у картофеля [24], хлопка [25], клубники [26], пшеницы [27], салата [28] и томатов [29]. Эти штаммы продуцируют различные вторичные метаболиты, включая липопептиды и поликетиды, которые проявляют сильную противогрибковую и антибактериальную активность, тем самым защищая растения от почвенных патогенов [16,17]. Аналогично, было показано, что Bacillus velezensis AP-3 повышает устойчивость томатов к солевому стрессу, модулируя антиоксидантную защитную систему растений и улучшая поглощение питательных веществ [30]. В другом исследовании Bacillus velezensis K-9 продемонстрировал значительную эффективность против парши картофеля, болезни, вызываемой Streptomyces scabies, путем продуцирования антимикробных соединений, ингибирующих рост патогена [31]. Более того, было сообщено, что Bacillus velezensis VJH504 контролирует увядание огурца, вызванное Fusarium, путем индуцирования системной устойчивости у растения и изменения ризосферного микробиома в пользу полезных микробов [23]. Данное исследование дальнейшим образом демонстрирует эффективность Bacillus velezensis TCS001 против Phytophthora cinnamomi, возбудителя корневой гнили гикори, с помощью как конфронтации на чашках, так и горшечных экспериментов. Полученные данные согласуются с предыдущими исследованиями видов Bacillus, подтверждая их потенциал в качестве агентов биоконтроля в различных сельскохозяйственных системах.
Заражение Phytophthora cinnamomi происходит в основном через активное движение двужгутиковых зооспор, хотя рост мицелия также способствует распространению болезни [32]. Зооспоры хемотаксически привлекаются к зоне удлинения корня, предпочтительно оседая в бороздках над антиклинальными клеточными стенками эпидермиса. При высокой плотности зооспоры демонстрируют самоагрегацию и кластеризацию, на которые влияют хемотаксис и биоконвекция. Прикрепление к поверхности корня опосредовано адгезивным белком PcVsv1 с молекулярной массой 250 кДа [33], секретируемым из вентральных везикул. Последующее проникновение и колонизация включают действие различных ферментов, разрушающих клеточную стенку растений [34]. После прикрепления рост мицелия продолжается через кору корня, как межклеточно, так и внутриклеточно, в конечном итоге достигая сосудистого пучка. Закупорка ксилемы мицелием нарушает транспорт воды, приводя к водному стрессу и гибели растения.
Виды Bacillus предлагают перспективную стратегию биоконтроля, вмешиваясь на различные стадии заражения Phytophthora [10,11]. Исследования показали, что Bacillus amyloliquefaciens может ингибировать прорастание и подвижность зооспор Phytophthora, ограничивая их способность заражать растения-хозяева [12]. В данном исследовании Bacillus velezensis TCS001, вероятно, нарушает заражение P. cinnamomi, продуцируя противогрибковые метаболиты, которые ингибируют прорастание зооспор и рост гиф, что подтверждается наблюдениями СЭМ деформированных гиф. TCS001 также может усиливать врожденный иммунный ответ растения, снижая восприимчивость к заражению P. cinnamomi, что согласуется с предыдущими исследованиями способности видов Bacillus бороться с почвенными патогенами.
Микробные инокулянты, включая виды Bacillus, могут усиливать полезные ризосферные микробные сообщества, такие как Flavobacterium, Pseudomonas, Agrobacterium и Lysobacter, одновременно подавляя почвенные патогены [13,35]. Виды Bacillus способствуют колонизации ризобий, увеличивают численность Flavobacterium johnsoniae и стимулируют местные популяции Pseudomonas, способствуя подавлению болезней [36,37,38]. Например, B. velezensis SQR9 увеличивает относительную численность Pseudomonas, Bacillus и Lysobacter, синергетически способствуя росту растений с нативной Pseudomonas stutzeri [39]. Данное исследование показывает, что применение TCS001 увеличивает относительную численность Sphingomonas, рода, стабильно обогащенного у здоровых растений, включая имбирь [40] и устойчивые к болезням томаты [41]. У риса Sphingomonas melonis повышает устойчивость к Burkholderia plantarii [42]. Эти данные предполагают, что TCS001 может подавлять заражение P. cinnamomi, способствуя обогащению ризосферы Sphingomonas. Кроме того, недавние исследования показали, что опосредованные Bacillus изменения в ризосферном микробиоме могут приводить к длительному подавлению болезней за счет увеличения численности антагонистических микробов и улучшения иммунных ответов растений [39,43].
Грибное сообщество в ризосфере гикори показало минимальные изменения в ответ на обработку Bacillus velezensis TCS001. Некоторые грибные таксоны, такие как Aspergillus, проявляли сниженную численность в ризосфере, обработанной TCS001, что указывает на то, что TCS001 может косвенно подавлять некоторые грибные патогены, способствуя развитию полезных бактерий, которые конкурируют с этими грибами или антагонизируют их [40,41]. Дополнительно, повышенная связность и модульность бактериальной сети ко-встречаемости в почвах, обработанных TCS001, может создавать более устойчивую микробную среду, ограничивающую укоренение грибных патогенов [44,45]. Хотя общее грибное сообщество оставалось относительно стабильным, специфическое снижение потенциальных грибных патогенов предполагает, что TCS001 все еще может играть роль в управлении грибными болезнями, хотя и косвенно, через свое воздействие на ризосферный микробиом. Недавние исследования подчеркивают критическую роль микробных модуляций в ответах растений на стресс, особенно в условиях таких стрессов, как засуха, засоление и патогенная атака [46,47,48].
Данное исследование изучало влияние B. velezensis TCS001 на структуру бактериальных и грибных сетей ко-встречаемости в ризосфере гикори, чтобы выяснить механизм его действия против нарушений окружающей среды. Между тем, ингибирующий эффект продуктов ферментации TCS001 на P. cinnamomi предполагает их потенциал в качестве агента биоконтроля корневой гнили гикори. Более высокая средняя степень и плотность сети указывают на повышенную чувствительность к изменению окружающей среды, в то время как высокая связность обеспечивает функциональную избыточность [49]. Эффективность сети оценивали с использованием средней степени и плотности сети [43], сравнивая сети ко-встречаемости с инокуляцией TCS001 и без нее. Бактериальное сообщество проявляло более высокую среднюю степень и плотность сети, чем грибное сообщество, особенно до инокуляции, указывая на более надежную структуру сети. Это согласуется с предыдущими данными, такими как эффекты подавления болезни B. velezensis ZN-S10 против бактериального некроза томата [50], подчеркивая важность антагонистических бактерий в формировании структуры микробного сообщества почвы. Увеличенная бактериальная связность обеспечивает функциональную избыточность, повышая устойчивость к патогенной инфекции. Таким образом, инокуляция TCS001 изменила структуру микробной сети ризосферы и повысила устойчивость к болезням, предлагая ценные данные для будущих исследований.
5. Выводы
В заключение, наше исследование предполагает, что TCS001 усиливает рекрутирование полезных микробов, ассоциированных с устойчивостью к болезням, тем самым ингибируя развитие болезни. Эти результаты подчеркивают, что TCS001 изменяет структуру ризосферного микробного сообщества в присутствии P. cinnamomi, предлагая ключевые данные для будущих исследований и развития эффективных подходов биоконтроля для борьбы с корневой гнилью гикори.
Дополнительные материалы
Следующая вспомогательная информация может быть загружена по адресу https://www.mdpi.com/article/10.3390/agriculture15020193/s1. Рисунок S1 Заболеваемость гикори, инокулированного выделенным патогеном. Гикори. (A) без обработки.; (B) инокулированный Phytophthora cinnamomi. Рисунок S2 Кривые разрежения 6 образцов для секвенирования гена 16S рДНК (A) и секвенирования гена ITS (B). Таблица S1. Сводка по обработке необработанных данных. Таблица S2. Относительная численность бактериальных сообществ на уровне типа. Таблица S3. Дифференциальная численность бактериальных OTU между CK и T6. Таблица S4. Дифференциальная численность грибных OTU между CK и T6. Таблица S5. Доли Типа в грибной сети. Таблица S6. Топологические свойства сетей.
Ссылки
1. Yang, J.; Zhou, F.; Xiong, L.; Mao, S.; Hu, Y.; Lu, B. Comparison of phenolic compounds, tocopherols, phytosterols and antioxidant potential in Zhejiang pecan [Carya cathayensis] at different stir-frying steps. LWT—Food Sci. Technol. 2015, 62, 541–548. [Google Scholar] [CrossRef]
2. Morales-Rodríguez, C.; Wang, Y.; Martignoni, D.; Vannini, A. Phytophthora cathayensis sp. nov., a new species pathogenic to Chinese Hickory (Carya cathayensis) in southeast China. Fungal Syst. Evol. 2020, 7, 99–111. [Google Scholar] [CrossRef]
3. Davison, E.M. Phytophthora Diseases Worldwide. Plant Pathology 1998, 47, 224–225. [Google Scholar] [CrossRef]
4. Tong, X.; Wu, J.; Mei, L.; Wang, Y. Detecting Phytophthora cinnamomi associated with dieback disease on Carya cathayensis using loop-mediated isothermal amplification. PLoS ONE 2021, 16, e0257785. [Google Scholar] [CrossRef]
5. Mendes, R.; Kruijt, M.; de Bruijn, I.; Dekkers, E.; van der Voort, M.; Schneider, J.H.M.; Piceno, Y.M.; DeSantis, T.Z.; Andersen, G.L.; Bakker, P.A.H.M.; et al. Deciphering the Rhizosphere Microbiome for Disease-Suppressive Bacteria. Science 2011, 332, 1097–1100. [Google Scholar] [CrossRef]
6. Liu, H.; Brettell, L.E.; Qiu, Z.; Singh, B.K. Microbiome-Mediated Stress Resistance in Plants. Trends Plant Sci. 2020, 25, 733–743. [Google Scholar] [CrossRef]
7. Kwak, M.-J.; Kong, H.G.; Choi, K.; Kwon, S.-K.; Song, J.Y.; Lee, J.; Lee, P.A.; Choi, S.Y.; Seo, M.; Lee, H.J.; et al. Rhizosphere microbiome structure alters to enable wilt resistance in tomato. Nat. Biotechnol. 2018, 36, 1100–1109. [Google Scholar] [CrossRef]
8. Castrillo, G.; Teixeira, P.J.P.L.; Paredes, S.H.; Law, T.F.; de Lorenzo, L.; Feltcher, M.E.; Finkel, O.M.; Breakfield, N.W.; Mieczkowski, P.; Jones, C.D.; et al. Root microbiota drive direct integration of phosphate stress and immunity. Nature 2017, 543, 513–518. [Google Scholar] [CrossRef]
9. Berendsen, R.L.; Vismans, G.; Yu, K.; Song, Y.; de Jonge, R.; Burgman, W.P.; Burmølle, M.; Herschend, J.; Bakker, P.A.H.M.; Pieterse, C.M.J. Disease-induced assemblage of a plant-beneficial bacterial consortium. ISME J. 2018, 12, 1496–1507. [Google Scholar] [CrossRef]
10. Yin, X.-T.; Xu, L.; Fan, S.-S.; Xu, L.-N.; Li, D.-C.; Liu, Z.-Y. Isolation and characterization of an AHL lactonase gene from Bacillus amyloliquefaciens. World J. Microbiol. Biotechnol. 2010, 26, 1361–1367. [Google Scholar] [CrossRef]
11. Pane, C.; Zaccardelli, M. Evaluation of Bacillus strains isolated from solanaceous phylloplane for biocontrol of Alternaria early blight of tomato. Biol. Control 2015, 84, 11–18. [Google Scholar] [CrossRef]
12. Dimopoulou, A.; Theologidis, I.; Liebmann, B.; Kalantidis, K.; Vassilakos, N.; Skandalis, N. Bacillus amyloliquefaciens MBI600 differentially induces tomato defense signaling pathways depending on plant part and dose of application. Sci. Rep. 2019, 9, 19120. [Google Scholar] [CrossRef] [PubMed]
13. Qin, Y.; Shang, Q.; Zhang, Y.; Li, P.; Chai, Y. Bacillus amyloliquefaciens L-S60 Reforms the Rhizosphere Bacterial Community and Improves Growth Conditions in Cucumber Plug Seedling. Front. Microbiol. 2017, 8, 2620. [Google Scholar] [CrossRef]
14. Choudhary, D.K.; Johri, B.N. Interactions of Bacillus spp. and plants—With special reference to induced systemic resistance (ISR). Microbiol. Res. 2009, 164, 493–513. [Google Scholar] [CrossRef] [PubMed]
15. Chen, Y.; Li, Y.; Fu, Y.; Jia, L.; Li, L.; Xu, Z.; Zhang, N.; Liu, Y.; Fan, X.; Xuan, W.; et al. The beneficial rhizobacterium Bacillus velezensis SQR9 regulates plant nitrogen uptake via an endogenous signaling pathway. J. Exp. Bot. 2024, 75, 3388–3400. [Google Scholar] [CrossRef] [PubMed]
16. Chen, X.H.; Koumoutsi, A.; Scholz, R.; Eisenreich, A.; Schneider, K.; Heinemeyer, I.; Morgenstern, B.; Voss, B.; Hess, W.R.; Reva, O.; et al. Comparative analysis of the complete genome sequence of the plant growth–promoting bacterium Bacillus amyloliquefaciens FZB42. Nat. Biotechnol. 2007, 25, 1007–1014. [Google Scholar] [CrossRef] [PubMed]
17. Ongena, M.; Jacques, P. Bacillus lipopeptides: Versatile weapons for plant disease biocontrol. Trends Microbiol. 2008, 16, 115–125. [Google Scholar] [CrossRef]
18. Edgar, R.C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads. Nat. Methods 2013, 10, 996–998. [Google Scholar] [CrossRef]
19. Stackebrandt, E.; Goebel, B.M. Taxonomic Note: A Place for DNA-DNA Reassociation and 16S rRNA Sequence Analysis in the Present Species Definition in Bacteriology. Int. J. Syst. Evol. Microbiol. 1994, 44, 846–849. [Google Scholar] [CrossRef]
20. Douglas, G.M.; Maffei, V.J.; Zaneveld, J.R.; Yurgel, S.N.; Brown, J.R.; Taylor, C.M.; Huttenhower, C.; Langille, M.G.I. PICRUSt2 for prediction of metagenome functions. Nat. Biotechnol. 2020, 38, 685–688. [Google Scholar] [CrossRef]
21. Schloss Patrick, D.; Westcott Sarah, L.; Ryabin, T.; Hall Justine, R.; Hartmann, M.; Hollister Emily, B.; Lesniewski Ryan, A.; Oakley Brian, B.; Parks Donovan, H.; Robinson Courtney, J.; et al. Introducing mothur: Open-Source, Platform-Independent, Community-Supported Software for Describing and Comparing Microbial Communities. Appl. Environ. Microbiol. 2009, 75, 7537–7541. [Google Scholar] [CrossRef] [PubMed]
22. Barberán, A.; Bates, S.T.; Casamayor, E.O.; Fierer, N. Using network analysis to explore co-occurrence patterns in soil microbial communities. ISME J. 2012, 6, 343–351. [Google Scholar] [CrossRef] [PubMed]
23. Yang, F.; Jiang, H.; Ma, K.; Wang, X.; Liang, S.; Cai, Y.; Jing, Y.; Tian, B.; Shi, X. Genome sequencing and analysis of Bacillus velezensis VJH504 reveal biocontrol mechanism against cucumber Fusarium wilt. Front. Microbiol. 2023, 14, 1279695. [Google Scholar] [CrossRef] [PubMed]
24. Schmiedeknecht, G.; Bochow, H.; Junge, H. Use of Bacillus subtilis as biocontrol agent. II. Biological control of potato diseases. J. Plant Dis. Prot. 1998, 105, 376–386. [Google Scholar]
25. Yao, A.V.; Bochow, H.; Karimov, S.; Boturov, U.; Sanginboy, S.; Sharipov, A.K. Effect of FZB 24 Bacillus subtilis as biofertilizer on cotton yields in field tests. Arch. Phytopathol. Plant Prot. 2006, 39, 323–328. [Google Scholar] [CrossRef]
26. Sylla, J.; Alsanius, B.W.; Krüger, E.; Reineke, A.; Strohmeier, S.; Wohanka, W. Leaf Microbiota of Strawberries as Affected by Biological Control Agents. Phytopathology® 2013, 103, 1001–1011. [Google Scholar] [CrossRef]
27. Talboys, P.J.; Owen, D.W.; Healey, J.R.; Withers, P.J.A.; Jones, D.L. Auxin secretion by Bacillus amyloliquefaciens FZB42 both stimulates root exudation and limits phosphorus uptake in Triticum aestivum. BMC Plant Biol. 2014, 14, 51. [Google Scholar] [CrossRef]
28. Chowdhury, S.P.; Dietel, K.; Rändler, M.; Schmid, M.; Junge, H.; Borriss, R.; Hartmann, A.; Grosch, R. Effects of Bacillus amyloliquefaciens FZB42 on Lettuce Growth and Health under Pathogen Pressure and Its Impact on the Rhizosphere Bacterial Community. PLoS ONE 2013, 8, e68818. [Google Scholar] [CrossRef]
29. Elanchezhiyan, K.; Keerthana, U.; Nagendran, K.; Prabhukarthikeyan, S.R.; Prabakar, K.; Raguchander, T.; Karthikeyan, G. Multifaceted benefits of Bacillus amyloliquefaciens strain FBZ24 in the management of wilt disease in tomato caused by Fusarium oxysporum f. sp. lycopersici. Physiol. Mol. Plant Pathol. 2018, 103, 92–101. [Google Scholar] [CrossRef]
30. Medeiros, C.A.; Bettiol, W. Multifaceted intervention of Bacillus spp. against salinity stress and Fusarium wilt in tomato. J. Appl. Microbiol. 2021, 131, 15095. [Google Scholar] [CrossRef]
31. Ma, S.; Wang, T.; Wang, Y. Bacillus velezensis K-9 as a Potential Biocontrol Agent for Managing Potato Scab. Plant Dis. 2023, 107, 3943–3951. [Google Scholar] [CrossRef] [PubMed]
32. Hardham, A.R.; Blackman, L.M. Phytophthora cinnamomi. Mol. Plant Pathol. 2018, 19, 260–285. [Google Scholar] [CrossRef] [PubMed]
33. Robold, A.V.; Hardham, A.R. During attachment Phytophthora spores secrete proteins containing thrombospondin type 1 repeats. Curr. Genet. 2005, 47, 307–315. [Google Scholar] [CrossRef] [PubMed]
34. Chang, H.-X.; Yendrek, C.R.; Caetano-Anolles, G.; Hartman, G.L. Genomic characterization of plant cell wall degrading enzymes and in silico analysis of xylanses and polygalacturonases of Fusarium virguliforme. BMC Microbiol. 2016, 16, 147. [Google Scholar] [CrossRef]
35. Xiong, W.; Guo, S.; Jousset, A.; Zhao, Q.; Wu, H.; Li, R.; Kowalchuk, G.A.; Shen, Q. Bio-fertilizer application induces soil suppressiveness against Fusarium wilt disease by reshaping the soil microbiome. Soil Biol. Biochem. 2017, 114, 238–247. [Google Scholar] [CrossRef]
36. Han, Q.; Ma, Q.; Chen, Y.; Tian, B.; Xu, L.; Bai, Y.; Chen, W.; Li, X. Variation in rhizosphere microbial communities and its association with the symbiotic efficiency of rhizobia in soybean. ISME J. 2020, 14, 1915–1928. [Google Scholar] [CrossRef]
37. Peterson, S.B.; Dunn, A.K.; Klimowicz, A.K.; Handelsman, J. Peptidoglycan from Bacillus cereus Mediates Commensalism with Rhizosphere Bacteria from the Cytophaga-Flavobacterium Group. Appl. Environ. Microbiol. 2006, 72, 5421–5427. [Google Scholar] [CrossRef]
38. Tao, C.; Li, R.; Xiong, W.; Shen, Z.; Liu, S.; Wang, B.; Ruan, Y.; Geisen, S.; Shen, Q.; Kowalchuk, G.A. Bio-organic fertilizers stimulate indigenous soil Pseudomonas populations to enhance plant disease suppression. Microbiome 2020, 8, 137. [Google Scholar] [CrossRef]
39. Sun, X.; Xu, Z.; Xie, J.; Hesselberg-Thomsen, V.; Tan, T.; Zheng, D.; Strube, M.L.; Dragoš, A.; Shen, Q.; Zhang, R.; et al. Bacillus velezensis stimulates resident rhizosphere Pseudomonas stutzeri for plant health through metabolic interactions. ISME J. 2022, 16, 774–787. [Google Scholar] [CrossRef]
40. Wang, W.; Portal-Gonzalez, N.; Wang, X.; Li, J.; Li, H.; Portieles, R.; Borras-Hidalgo, O.; He, W.; Santos-Bermudez, R. Metabolome-driven microbiome assembly determining the health of ginger crop (Zingiber officinale L. Roscoe) against rhizome rot. Microbiome 2024, 12, 167. [Google Scholar] [CrossRef]
41. Jin, X.; Jia, H.; Ran, L.; Wu, F.; Liu, J.; Schlaeppi, K.; Dini-Andreote, F.; Wei, Z.; Zhou, X. Fusaric acid mediates the assembly of disease-suppressive rhizosphere microbiota via induced shifts in plant root exudates. Nat. Commun. 2024, 15, 5125. [Google Scholar] [CrossRef] [PubMed]
42. Matsumoto, H.; Fan, X.; Wang, Y.; Kusstatscher, P.; Duan, J.; Wu, S.; Chen, S.; Qiao, K.; Wang, Y.; Ma, B.; et al. Bacterial seed endophyte shapes disease resistance in rice. Nat. Plants 2021, 7, 60–72. [Google Scholar] [CrossRef] [PubMed]
43. Ping, X.; Khan, R.A.A.; Chen, S.; Jiao, Y.; Zhuang, X.; Jiang, L.; Song, L.; Yang, Y.; Zhao, J.; Li, Y.; et al. Deciphering the role of rhizosphere microbiota in modulating disease resistance in cabbage varieties. Microbiome 2024, 12, 160. [Google Scholar] [CrossRef]
44. Shaw, G.T.-W.; Liu, A.-C.; Weng, C.-Y.; Chen, Y.-C.; Chen, C.-Y.; Weng, F.C.-H.; Wang, D.; Chou, C.-Y. A network-based approach to deciphering a dynamic microbiome’s response to a subtle perturbation. Sci. Rep. 2020, 10, 19530. [Google Scholar] [CrossRef]
45. Mougi, A.; Kondoh, M. Diversity of Interaction Types and Ecological Community Stability. Science 2012, 337, 349–351. [Google Scholar] [CrossRef]
46. Ma, Y.; Rajkumar, M.; Zhang, C.; Freitas, H. Beneficial role of bacterial endophytes in heavy metal phytoremediation. J. Environ. Manag. 2016, 174, 14–25. [Google Scholar] [CrossRef]
47. Santos-Medellín, C.; Edwards, J.; Liechty, Z.; Nguyen, B.; Sundaresan, V. Drought Stress Results in a Compartment-Specific Restructuring of the Rice Root-Associated Microbiomes. mBio 2017, 8, e00764-17. [Google Scholar] [CrossRef]
48. Yuan, Z.; Druzhinina, I.S.; Labbé, J.; Redman, R.; Qin, Y.; Rodriguez, R.; Zhang, C.; Tuskan, G.A.; Lin, F. Specialized Microbiome of a Halophyte and its Role in Helping Non-Host Plants to Withstand Salinity. Sci. Rep. 2016, 6, 32467. [Google Scholar] [CrossRef]
49. Hernandez, D.J.; David, A.S.; Menges, E.S.; Searcy, C.A.; Afkhami, M.E. Environmental stress destabilizes microbial networks. ISME J. 2021, 15, 1722–1734. [Google Scholar] [CrossRef]
50. Chen, E.; Chao, S.; Shi, B.; Liu, L.; Chen, M.; Zheng, Y.; Feng, X.; Wu, H. Bacillus velezensis ZN-S10 Reforms the Rhizosphere Microbial Community and Enhances Tomato Resistance to TPN. Plants 2023, 12, 3636. [Google Scholar] [CrossRef]
Xie C, Wu Y, Wu Z, Cao H, Huang X, Cui F, Meng S, Chen J. Bacillus velezensis TCS001 Enhances the Resistance of Hickory to Phytophthora cinnamomi and Reshapes the Rhizosphere Microbial Community. Agriculture. 2025; 15(2):193. https://doi.org/10.3390/agriculture15020193
Перевод статьи «Bacillus velezensisTCS001 Enhances the Resistance of Hickory toPhytophthora cinnamomiand Reshapes the Rhizosphere Microbial Community» авторов Xie C, Wu Y, Wu Z, Cao H, Huang X, Cui F, Meng S, Chen J., оригинал доступен по ссылке. Лицензия: CC BY. Изменения: переведено на русский язык
































































































Комментарии (0)