3D-сегментация сои: быстрый путь к точным данным для селекционеров
Фенотипический анализ созревших растений сои является критически важным аспектом селекции сои. Однако ручное получение фенотипических параметров не только требует много времени и трудозатрат, но также страдает от недостатка объективности. Следовательно, существует настоятельная потребность в быстром, точном и эффективном методе сбора фенотипических параметров сои.
Аннотация
В данном исследовании разработан новый подход (пайплайн) для получения фенотипических признаков созревших растений сои на основе трёхмерных (3D) облаков точек. Во-первых, облака точек сои получают с использованием метода многоракурсной стерео 3D-реконструкции, после чего проводят предобработку для построения набора данных. Во-вторых, специально для сегментации стручков и стеблей сои предложена сеть на основе глубокого обучения — PVSegNet (Point Voxel Segmentation Network). Эта сеть повышает способность извлечения признаков за счёт интеграции свёртки по точкам и вокселям, а также благодаря модулю кодирования ориентации (orientation-encoding, OE). Наконец, фенотипические параметры, такие как диаметр стебля, длина и ширина стручка, извлекаются и проверяются путём сравнения с ручными измерениями.
Экспериментальные результаты показывают, что средний показатель пересечения по отношению к объединению (IoU) для семантической сегментации составляет 92,10%, точность — 96,38%, полнота — 95,41%, а F1-мера — 95,87%. Для сегментации экземпляров сеть достигает средней точности (AP@50) 83,47% и средней полноты (AR@50) 87,07%. Эти результаты указывают на пригодность сети для сегментации экземпляров стручков и стеблей. При извлечении параметров растений прогнозируемые значения ширины стручка, длины стручка и диаметра стебля, полученные с помощью метода извлечения фенотипических параметров, демонстрируют коэффициенты детерминации (Р2Значения составили 0,9489, 0,9182 и 0,9209 соответственно при ручных измерениях. Это демонстрирует, что наш метод может значительно повысить эффективность и точность, способствуя применению автоматизированной технологии анализа 3D-облаков точек в селекции сои.
1. Введение
Соя является одной из основных мировых продовольственных культур, играющей жизненно важную роль в питании и здоровье человека [1,2]. Хотя достижения в технологии селекции значительно увеличили урожайность сои, остается крайне важным выведение новых сортов, которые были бы высокоурожайными, высококачественными и обладали бы множественными признаками устойчивости для удовлетворения растущего спроса [3]. Фенотипический анализ особенно важен в селекции сои, так как включает детальное наблюдение и измерение ключевых признаков растений сои [4]. Среди фенотипических параметров в селекции сои характеристики стручков и главного стебля являются критическими показателями для повышения урожайности и отбора лучших сортов. Такие признаки, как цвет, размер и количество стручков, а также высота главного стебля и количество узлов, оказывают существенное влияние на урожайность и качество сои [5]. Следовательно, применение эффективных методов сбора и анализа фенотипических параметров необходимо для продвижения научного и точного развития селекции сои [6]. Традиционные методы ручного извлечения, такие как визуальный подсчет и ручное измерение, хотя и интуитивно понятны, имеют ряд ограничений: неточный подсчет, высокие трудозатраты и низкая точность измерений [5]. Эти ручные методы часто не соответствуют требованиям крупномасштабного и высокоточного анализа, особенно при обработке больших объемов образцов, когда трудно сбалансировать эффективность и точность. В последнее время методы на основе компьютерного зрения получают все большее внимание в исследованиях растений, поскольку эти технологии могут эффективно визуализировать структуры растений, точно измерять фенотипические признаки и уменьшать человеческие ошибки, открывая новые возможности для высокопроизводительного фенотипирования растений [7].
Методы анализа двумерных (2D) изображений широко применяются в высокопроизводительном фенотипировании растений. Например, исследования по извлечению фенотипических признаков из 2D-изображений различных культур, таких как томаты [8], кукуруза [9], сорго [10] и пшеница [11], продемонстрировали эффективность этих методов. Однако эти 2D-методы визуализации имеют существенные ограничения, включая (1) трудности в решении проблемы окклюзии из-за отсутствия информации о глубине [12] и (2) сложности в определении структурной информации объектов [10].
Для решения ограничений методов на основе 2D-изображений были приложены значительные усилия по разработке 3D-систем визуализации для фенотипирования растений [13,14]. По сравнению с 2D-данными, 3D-данные не только преодолевают эти ограничения, но и повышают точность оценки, переходя от уровня пикселей в 2D к уровню точек в 3D [15]. В настоящее время быстрый сбор 3D-данных растений облегчается благодаря достижениям в технологии сенсоров и улучшению вычислительной производительности [16]. При получении 3D-данных растений такие технологии, как лидар (LiDAR) [17], времяпролетные камеры (ToF) [18], камеры глубины [19] и многоракурсные стереокамеры (MVS) [20], широко используются для 3D-реконструкции и фенотипического анализа растений. Sun и др. [21] применили технологию MVS для 3D-реконструкции сои, охватив весь ее цикл роста. Дополнительно, Liyi Luo и др. [22] разработали недорогую платформу для 3D-реконструкции и сканирования для получения данных облака точек сои, из которых были извлечены фенотипические параметры, такие как длина листа, ширина листа и диаметр стебля.
Сегментация облаков точек сои является чрезвычайно сложной задачей из-за нерегулярного и дисперсного характера морфологии. Для эффективного и точного получения семантических меток посредством сегментации необходим эффективный процесс сегментации для извлечения признаков из облака точек растений сои. Многочисленные существующие исследования разработали традиционные методы компьютерного зрения для сегментации органов растений, такие как методы на основе порогов [23], методы на основе геометрии [24], методы на основе октодеревьев [25] и методы на основе 3D-скелетов [26]. Однако эти методы часто сильно зависят от предопределенных правил и априорных знаний о целях сегментации [27]. Ограниченные априорные знания о морфологии растений сужают применение традиционных методов при обработке простых структур и признаков 3D-фенотипов растений [28].
В последние годы методы на основе глубокого обучения быстро развиваются, значительно улучшая универсальность и точность сегментации растений [29]. Методы на основе глубокого обучения для сегментации облаков точек растений в основном включают воксельные и точечные подходы. Точечные методы используют общие многослойные перцептроны (MLP) для непосредственного изучения признаков каждой точки [30]. Li и др. [31] разработали сеть для сегментации облаков точек растений под названием DeepSeg3DMaize, основанную на сети PointNet, которая интегрирует высокопроизводительный сбор данных и глубокое обучение. PlantNet [32] — это 3D-сеть глубокого обучения на основе точек с двойной функцией, способная выполнять как семантическую, так и инстанс-сегментацию стеблей и листьев для трех различных культур. Однако большинство точечных методов в основном предназначены для входных облаков точек небольшого масштаба из-за аппаратных ограничений, влияющих на архитектуру сети и выбор гиперпараметров. Следовательно, вычислительная стоимость точечных методов сильно зависит от количества входных точек, и полномасштабный ввод может снизить скорость обучения без обязательного улучшения производительности. С другой стороны, воксельные методы преобразуют облака точек в сетки и используют 3D-воксельные свертки для обработки облаков точек, что полезно для моделирования локального контекста. Однако эти методы требуют высокого разрешения вокселизации, чтобы избежать потери информации [33]. Jin и др. [34] разработали воксельную сеть глубокого обучения специально для семантической классификации и инстанс-сегментации листьев кукурузы. PSegNet [35] — это еще одна сеть, которая применяет воксельные методы для выполнения задач как инстанс, так и семантической сегментации для нескольких культур. Дополнительно, часто бывает сложно найти баланс между производительностью и вычислительной стоимостью при использовании методов свертки на воксельных сетках [36].
Основной проблемой в этой области является отсутствие открытых наборов данных для обучения и оценки методов инстанс-сегментации облаков точек растений. Большие, разнообразные и хорошо аннотированные наборы данных крайне важны для обучения сетей глубокого обучения и оценки их производительности [37]. Pheno4D [38] предоставляет пространственно-временной набор данных облаков точек, но он включает только семь растений томата и семь растений кукурузы. ROSE-X [27] предлагает аннотированный 3D-набор данных растений розы, специально разработанный для обучения и оценки методов сегментации органов. Однако этот набор данных содержит только 11 аннотированных 3D-моделей растений, каждая с воксельными метками органов, соответствующими ветвям растения. Высококачественные наборы данных облаков точек обычно повышают производительность сегментации глубоких сетей, что делает построение таких наборов данных центральным направлением исследований в области сегментации облаков точек растений [39].
Для решения этих проблем в данном исследовании предлагается пайплайн для извлечения фенотипических параметров из облаков точек сои посредством инстанс-сегментации стручков и стеблей. Основные вклады заключаются в следующем: (1) Мы разработали 3D-платформу для сканирования, которая захватывает изображения сои под разными углами и генерирует высококачественные облака точек отдельных растений с использованием алгоритма многоракурсной 3D-реконструкции. Был построен набор данных, состоящий из облаков точек 60 растений сои, с инстанс-аннотациями для стручков и стеблей. (2) Мы разработали сеть PVSegNet специально для инстанс-сегментации стручков и стеблей. Путем интеграции воксельных и точечных признаков и агрегации локальных признаков, способности сети к извлечению признаков улучшены. Дополнительно, для достижения сквозной инстанс-сегментации стручков и стеблей был применен алгоритм PointGroup, что демонстрирует осуществимость 3D инстанс-сегментации облаков точек сои. (3) На основе результатов сегментации были извлечены фенотипические параметры, такие как длина стручка, ширина стручка и диаметр стебля, обеспечивая техническую поддержку для высокопроизводительного фенотипирования растений.
2. Материалы и методы
2.1. Экспериментальные образцы сои
Экспериментальные материалы были высажены на экспериментальной базе Северо-Восточного сельскохозяйственного университета в Харбине, провинция Хэйлунцзян, Китай (45°36′ с.ш., 126°18′ в.д.). Экспериментальное поле для выращивания сои показано на Рисунке 1c. Созревшая соя, собранная в 2023 году, послужила экспериментальным материалом для фенотипического сбора в данном исследовании. Метод горшечного выращивания был следующим: два растения выращивались на один горшок, с расстоянием 30 сантиметров между горшками. Посев проводился в середине августа, а сбор урожая был запланирован на середину октября. На стадии созревания сои было собрано 60 растений в качестве экспериментальных образцов.
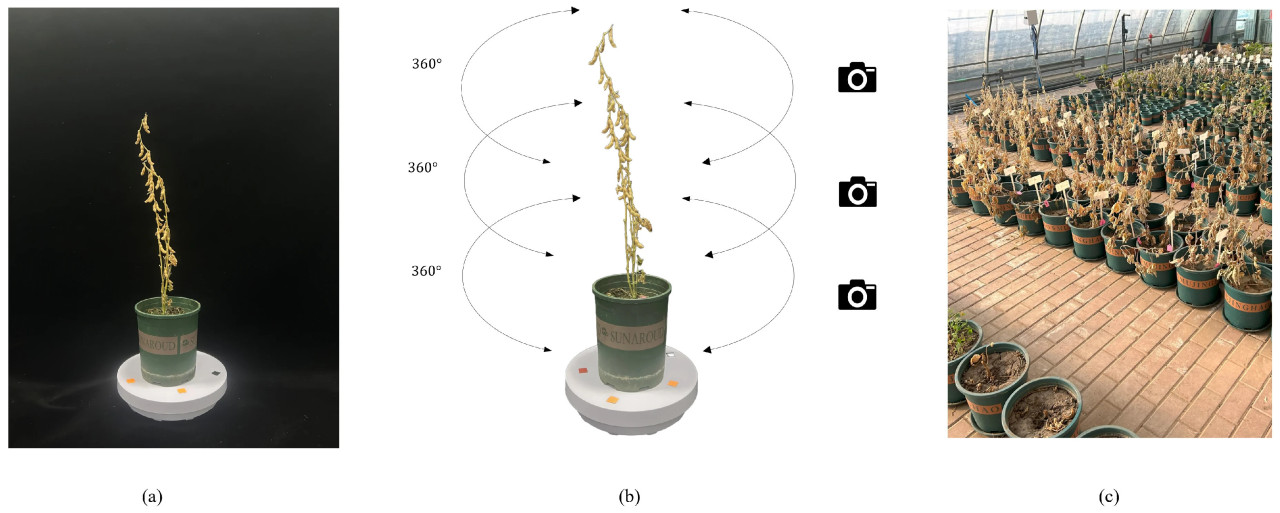
Рисунок 1. Сбор образцов сои и получение данных. (a) Место съемки, (b) метод съемки и (c) место посадки.
2.2. Генерация данных облака точек
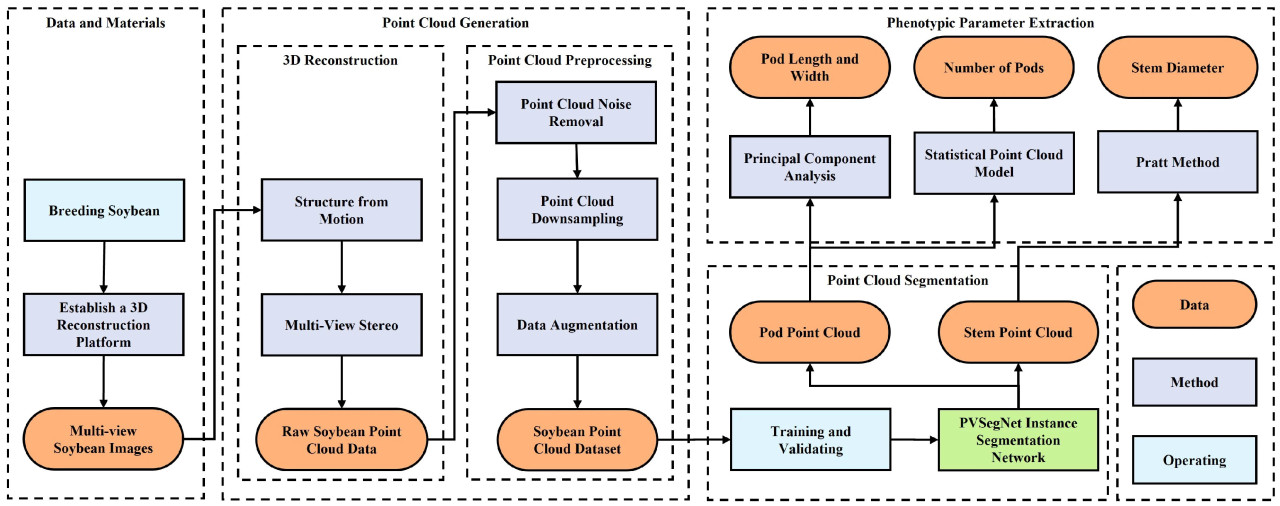
Метод, использованный в этом исследовании, состоит из четырех ключевых этапов: (1) сбор материалов и данных, (2) генерация 3D облаков точек сои, (3) сегментация областей стручков и стеблей с помощью нейронной сети и (4) извлечение фенотипических параметров сои. Рисунок 2 предоставляет детальный обзор методологической последовательности, включая конкретные шаги для извлечения фенотипических параметров сои с использованием данных облака точек.
Рисунок 2. Блок-схема фенотипирования растения сои, включающая сбор данных, генерацию облака точек, сегментацию облака точек и извлечение фенотипических параметров.
2.2.1. Получение изображений
В этом исследовании для 3D-реконструкции были собраны многоракурсные изображения сои на разных высотах (как показано на Рисунке 1b). Этот подход по сравнению с традиционной съемкой с фиксированной высоты позволяет более полно захватить структурную информацию сои [40]. Для достижения 3D-реконструкции растений сои мы разработали платформу для 3D-сканирования, которая включает камеру Sony A7, черную светопоглощающую ткань, штатив, моторизованный поворотный стол и освещение. Процент перекрытия захваченных изображений напрямую влияет на точность реконструкции; низкий процент перекрытия может привести к несоответствию изображений и потере реконструируемых областей [41]. Мы регулировали высоту штатива камеры и углы съемки, а также контролировали скорость поворотного стола, чтобы определить количество сделанных снимков. Из-за ограниченной чувствительности сенсора изображения камеры освещение играло ключевую роль в эксперименте, помогая более эффективно захватывать детали изображения, при этом дополнительные источники света использовались для подсветки. Чрезмерный фон, перекрытие растений и темные области обычно трудно захватить и они увеличивают риск неудачи 3D-реконструкции [42]. Мы использовали черную светонепроницаемую ткань для устранения теней и фоновых помех, как показано на Рисунке 1a.
Растения сои размещались на поворотном столе, и угол поворота контролировался путем регулировки скорости двигателя. Тем временем мы вручную регулировали угол штатива камеры для захвата изображений растений сои с нескольких перспектив. Для поддержания стабильности в процессе захвата изображений поворотный стол останавливался ритмично, а не вращался непрерывно. Фотография делалась примерно каждые 6 градусов поворота, что давало 60 изображений за один полный оборот. Затем высота камеры регулировалась три раза, от низкой к высокой, каждый раз на одну треть высоты растения. Этот процесс повторялся три раза, в результате чего получалось три набора изображений с вращения под разными углами, всего 180 изображений. Съемка изображений проводилась в контролируемых лабораторных условиях, чтобы избежать помех от внешних факторов, таких как освещение и ветер. На каждое растение сои уходило примерно 5 минут для фотографирования. После завершения съемки мы измерили фенотипические параметры растений сои для проверки метода, предложенного в данном исследовании. Используя линейку с единицей измерения 1 мм, измеряли длину и ширину стручков сои. Для диаметра стебля использовался штангенциркуль с единицей измерения 0,01 мм.
2.2.2. Трехмерная реконструкция
В этом исследовании 3D-реконструкция растений сои была выполнена с использованием многоракурсных изображений сои и алгоритма Multi-view Stereo (MVS) [43], генерируя соответствующие данные облака точек. Из полученных последовательностей изображений с помощью метода Structure-from-Motion (SFM) [44] были восстановлены разреженные данные облака точек. Впоследствии мы использовали алгоритм MVS для дальнейшей реконструкции этих разреженных облаков точек в плотные облака точек. Комбинируя алгоритмы SFM и MVS, мы эффективно реконструировали 3D-структуру растений сои из изображений, сделанных под разными углами, предоставляя высококачественные данные облака точек для поддержки последующих анализов и приложений. Этот метод не только позволил получить 3D-данные облака точек, но и предоставил надежные исходные данные для обучения сетей глубокого обучения.
Алгоритм SFM — это фотограмметрическая техника, которая оценивает 3D-структуры из последовательностей изображений. Посредством этих технических шагов мы смогли точно вычислить внутренние и внешние параметры каждого изображения и сгенерировать разреженные облака точек растений сои. Алгоритм MVS затем использовал откалиброванные изображения и эпиполярную геометрию исходных данных для генерации высокоплотных облаков точек, которые были раскрашены для получения окончательных плотных данных облака точек.
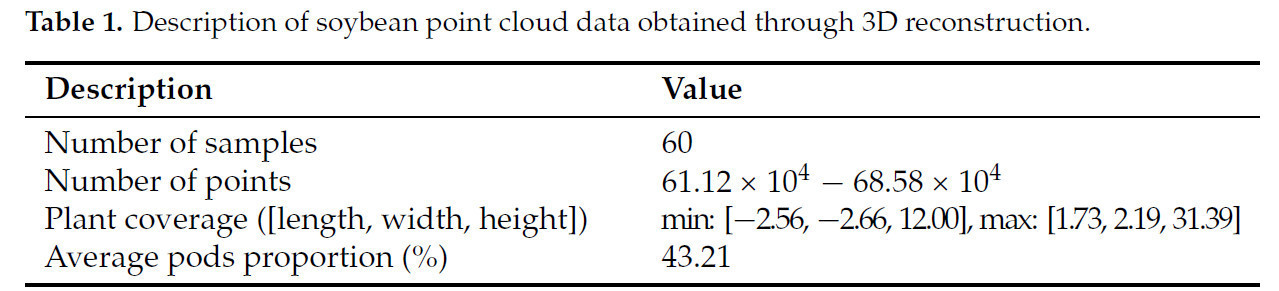
Мы использовали Agisoft Metashape (Agisoft LLC, Санкт-Петербург, Россия) для генерации требуемых данных облака точек сои. Agisoft Metashape был применен для 3D-реконструкции [45], поскольку он способен обрабатывать высококачественные последовательности изображений и предоставлять высокоточные и высококачественные результаты реконструкции. Программное обеспечение предлагает комплексный рабочий процесс, включая выравнивание фотографий, извлечение признаков, сопоставление признаков, расчет точек обзора камеры и 3D-реконструкцию облака точек. Чтобы обеспечить точность реконструкции, мы отрегулировали параметры программного обеспечения: максимальное количество признаков на изображение было установлено на 40 000, перекрытие изображений установлено на высокое, а точность реконструкции установлена на самый высокий уровень. Поскольку Agisoft Metashape поддерживает как GPU, так и CPU-ускорение, он предлагает высокую скорость и низкие временные затраты во время 3D-реконструкции. Процесс реконструкции занимает приблизительно 30 минут на объект. По завершении данные облака точек, экспортированные из Agisoft Metashape, были в формате PLY (Polygon File Format), как показано в Таблице 1. Файл данных облака точек содержит массив M × 6, где M представляет количество точек, а 6 включает координаты X, Y, Z и каналы цвета: красный, зеленый и синий.
Таблица 1. Описание данных облака точек сои, полученных посредством 3D-реконструкции.
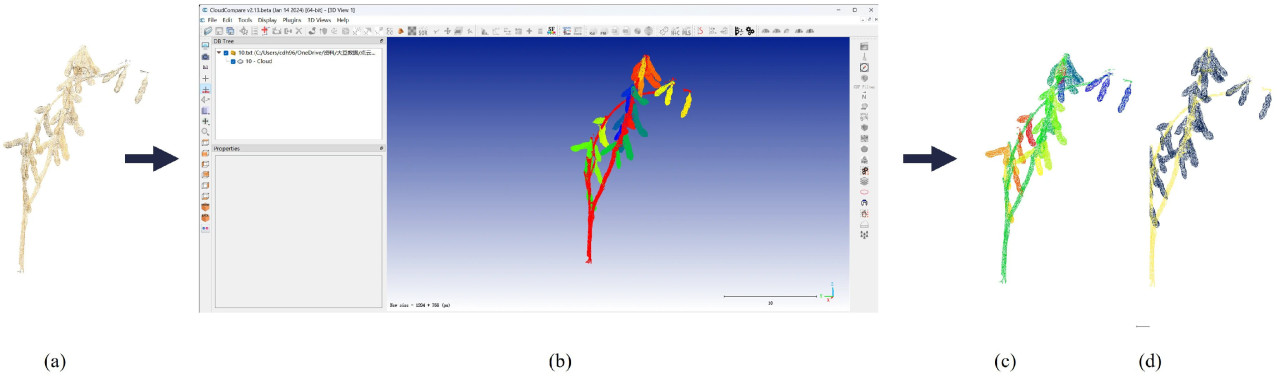
Аннотирование данных — трудоемкая задача, но она крайне важна для обучения сетей глубокого обучения [46]. Мы использовали программное обеспечение CloudCompare (версия 2.12.4) для ручного выбора и удаления несоевых частей, чтобы получить чистые данные облака точек растения сои. Мы приняли стандартный формат открытого набора данных S3DIS [47] для аннотирования набора данных облака точек сои. Во время ручной аннотации мы присваивали семантические и инстанс-метки каждой точке в облаке точек. Семантическая аннотация классифицировала облако точек на две категории: стручки и стебли. На этапе инстанс-сегментации каждый стручок был индивидуально помечен. Файл аннотации содержит исходные данные облака точек вместе с семантическими и инстанс-категориями каждой точки для стручков и стеблей. Рисунок 3 показывает процесс аннотирования данных облака точек сои с использованием CloudCompare. В конечном итоге набор данных содержит 60 полных облаков точек сои и 1782 соответствующих файла аннотаций.
Рисунок 3. Процесс аннотирования данных. (a) Исходное облако точек сои, (b) интерфейс программного обеспечения для аннотирования, (c) инстанс-аннотация, где разные цвета представляют разные стручки, и (d) семантическая аннотация, где разные цвета представляют категории стручков и стеблей.
2.2.3. Предварительная обработка облака точек
После обработки многоракурсных изображений сои с помощью Agisoft Metashape результирующие модели облаков точек растений сои были очень плотными, с приблизительно 600 000 точек на модель. Чтобы гарантировать, что данные подходят для последующего обучения сети сегментации, мы реализовали следующие шаги предварительной обработки облака точек: (1) шумоподавление облака точек для удаления выбросов из облака точек сои; (2) понижающая дискретизация облака точек для сохранения исходного распределения данных при одновременном уменьшении плотности облака точек, тем самым повышая эффективность обработки; и (3) аугментация данных для расширения исходного набора данных и улучшения способности сети к обобщению и ее устойчивости.
Шумоподавление облака точек
Мы использовали радиальный фильтр и статистический фильтр для устранения шума из облаков точек сои [48]. Ручное удаление выбросов может быть сложным, но эти фильтры эффективно отфильтровывают шум облака точек. Радиальный фильтр демонстрирует превосходную производительность в сохранении формы и краев, предлагая при этом высокую скорость обработки, что делает его эффективным для обработки больших объемов шума. С другой стороны, статистический фильтр использует глобальные статистические характеристики и эффективен для удаления незначительного шума.
Понижающая дискретизация облака точек
Понижающая дискретизация облака точек в основном использовалась для уменьшения объема данных и повышения вычислительной эффективности [49]. Метод равномерной понижающей дискретизации [50] сохраняет распределение данных и основную информацию о признаках, одновременно уменьшая плотность облака точек. Впоследствии мы применили случайную понижающую дискретизацию [51] для стандартизации количества точек в облаках точек сои. Изначально данные облака точек содержали около 600 000 точек, которые мы в конечном итоге сократили до 10 240 точек на растение сои.
Аугментация данных
Теоретически методы аугментации данных могут значительно повысить устойчивость и способность к обобщению сети [52]. В процессе 3D-реконструкции облака точек могут сталкиваться с такими проблемами, как частичная потеря, смещение системы координат и вариации масштаба [53]. Для решения этих проблем мы применили глобальные методы аугментации данных, такие как масштабирование, кадрирование, поворот и перемещение (Рисунок 4). Эти техники аугментации увеличили разнообразие и богатство данных, позволяя сети лучше адаптироваться к различным изменениям окружающей среды [54]. Используя эти методы аугментации данных, мы расширили исходный набор данных облака точек, создав набор из 300 образцов для обучения и тестирования сети сегментации органов сои. Предварительно обработанные облака точек сои прошли последовательно четыре техники аугментации данных, что привело к расширенному набору данных облака точек сои.
Рисунок 4. Изменения в данных облака точек сои до и после аугментации. Серое облако точек представляет исходные данные, а цветное облако точек показывает результаты аугментации. (a) Перемещение, (b) масштабирование, (c) кадрирование и (d) поворот.
2.3. Сегментация облака точек
2.3.1. Архитектура сети
В исследовании сегментации облака точек растений сои наша цель — достичь как семантической, так и инстанс-сегментации стручков и стеблей. Это означает, что каждая точка не только получает семантическую метку, но и ей присваивается конкретный инстанс-ID для соответствующего стручка. Мы разработали сквозную сеть инстанс-сегментации для сегментации органов сои, с архитектурой сети PVSegNet, проиллюстрированной на Рисунке 5. Сеть интегрирует модули PVConv [55] и orientation-encoding (OE) [56] в структуру PointNet++ для улучшения извлечения признаков. Это помогает решить проблемы, вызванные нерегулярностью и дисперсностью стручков и стеблей, тем самым повышая точность семантической сегментации. Дизайн сети извлечения признаков показан на Рисунке 6. Мы включили метод PointGroup для достижения сквозной инстанс-сегментации растений сои.
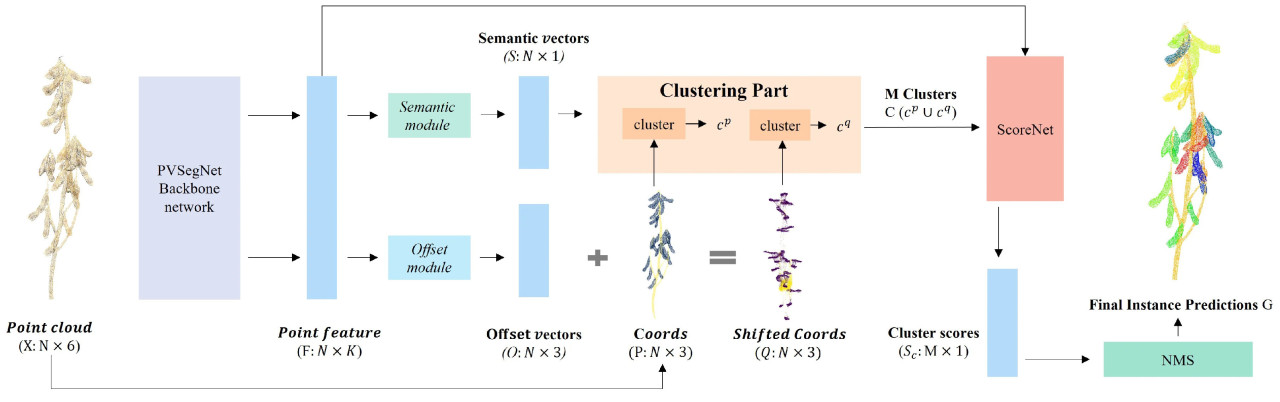
Рисунок 5. Архитектура сети PVSegNet для сегментации областей стручков и стеблей в данных облака точек сои. Синие прямоугольники представляют векторы, а другие цветные прямоугольники представляют соответствующие операции, указанные текстом внутри них. Сначала облако точек сои X пропускается через базовую сеть для извлечения признаков F. Затем семантический модуль и модуль смещения генерируют S и O соответственно. Координаты P извлекаются из X, и смещение O добавляется к P для получения смещенных координат Q. Затем используются методы кластеризации для получения результатов кластеризации экземпляров 𝐶𝑝 и 𝐶𝑞 для стручков или стеблей, а объединение 𝐶𝑝 и 𝐶𝑞 дает M кластеров C. Эти кластеры оцениваются с помощью ScoreNet для получения соответствующих оценок кластеров 𝑆𝑐. Наконец, для достижения окончательного результата инстанс-сегментации G используется метод NMS (Non-Maximum Suppression).
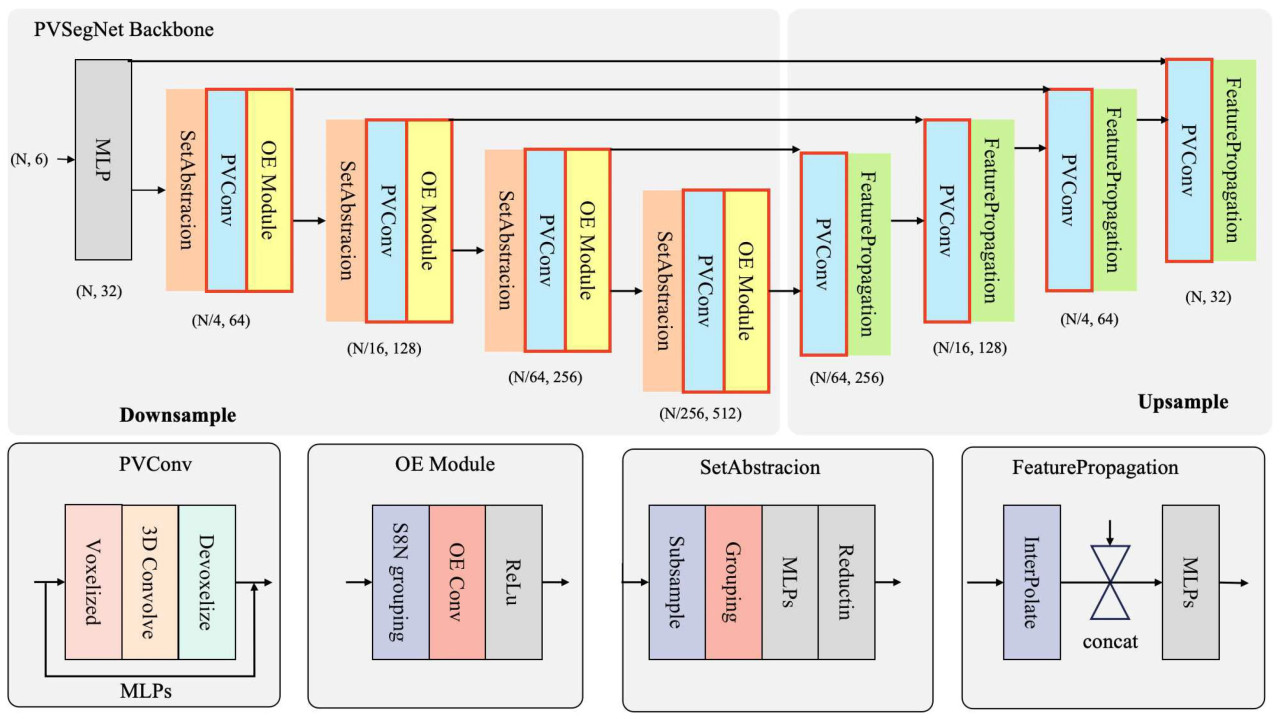
Рисунок 6. На рисунке показана архитектура PVSegNet, включающая как компоненты повышающей, так и понижающей дискретизации. Она включает модули PVConv, OE, SetAbstraction и FeaturePropagation.
PVConv [55] использует точечно-воксельную сверточную нейронную сеть, которая улучшает производительность сети за счет комбинации точечных и воксельных представлений. Модуль PVConv интегрирует преимущества точечных методов, которые имеют более низкое использование памяти, и воксельных методов, которые полезны для регулярности локальных признаков. Этот модуль улучшает эффективность и действенность каждой ветви за счет мелкозернистого преобразования признаков и крупнозернистой агрегации окрестностей. Как показано на Рисунке 6, модуль PVConv включает две ветви: одна генерирует воксельную карту признаков для входного облака точек, а другая производит точечную карту признаков. Эти признаки объединяются посредством операции сложения для получения выхода модуля PVConv. В ветви вокселизации облако точек сначала вокселизируется через модуль вокселизации, затем следует 3D-свертка для извлечения признаков, и де-вокселизация используется для слияния с признаками облака точек. Наконец, эти признаки объединяются с ветвью облака точек через слои многослойного перцептрона (MLP). В этом исследовании мы заменили слои MLP в исходном PointNet++ на модуль PVConv, чтобы одновременно извлекать признаки как из воксельных, так и из точечных данных, улучшая способность модели к извлечению признаков.
Модуль ориентационного кодирования (OE) [56] в сети PointSIFT играет ключевую роль в улучшении извлечения информации об окрестностях, служа основным компонентом сети. Проще говоря, модуль OE кодирует информацию из восьми различных направлений, помогая сети лучше понимать локальные признаки. Внутренняя структура модуля OE показана на Рисунке 6. Этот модуль кодирует информацию из восьми направлений с помощью точечных дескрипторов для улучшения выражения локальных признаков. Конкретно, модуль OE использует поиск по стеку из 8 окрестностей (S8N), идентифицируя ближайших соседей в пределах октанта для генерации куба 2 × 2 × 2, описывающего локальные признаки. Впоследствии куб проходит три уровня OE-свертки вдоль осей X, Y и Z, интегрируя информацию из восьми пространственных направлений для формирования представления, кодирующего информацию о направлении. В этом исследовании мы добавили модуль OE в часть понижающей дискретизации сети, чтобы улучшить способность сети извлекать локальные признаки.
В текущих исследованиях методы сегментации органов растений включают прямую кластеризацию для сегментации или использование методов кластеризации после семантической сегментации. Эти методы часто требуют ручной настройки параметров и корректировок для различных сред [57,58]. Между тем, PointGroup [59] — это двухэтапная сеть, которая может быть интегрирована с PVSegNet для достижения инстанс-сегментации органов сои. Чтобы упростить процесс сегментации, мы использовали PointGroup, сквозной метод сегментации, в рамках которого строится сеть PVSegNet, при этом сеть сначала получает данные облака точек X и извлекает признаки F через сеть PVsegNet. Затем признаки подаются в две ветви: семантическую и модуль смещения. В семантической ветви признаки F проходят через слои MLP и преобразуются в семантические метки S с помощью операции Argmax. В ветви смещения цель модуля смещения — сдвинуть каждую точку к центру ее экземпляра, тем самым позволяя идентифицировать отдельные экземпляры. Модуль смещения [59] вычисляет вектор смещения O для каждой точки, который добавляется к исходным координатам P из X для получения смещенных координат Q. Это направлено на выравнивание каждой точки с центроидом ее экземпляра. Скорректированные координаты P и Q группируются в кандидатные кластеры 𝐶𝑃 и 𝐶𝑄 с использованием метода кластеризации [59]. Этот процесс кластеризации создает сферические области, где соседние точки кластеризуются вместе. Когда точки внутри сферы имеют метки стручков или стеблей, они группируются как соответствующий экземпляр. Объединение 𝐶𝑃 и 𝐶𝑄 формирует окончательный результат кластеризации C. Далее, F и C обрабатываются через ScoreNet [59] для расчета оценки 𝑆𝑐 для каждого кандидатного экземпляра.
Во время фазы обучения функция потерь состоит из четырех компонентов: потери семантической сегментации, потери регрессии ветви смещения, потери направления ветви смещения и потери ScoreNet. Потери семантической сегментации и потери ScoreNet рассчитываются с использованием функции потерь перекрестной энтропии. Семантическая ветвь классифицирует каждую точку для вычисления ее потерь, с конкретным выражением функции потерь следующим образом, где N — общее количество точек, C — количество семантических категорий, 𝑦𝑖,𝑐 — истинная метка класса i-й точки, и 𝑝𝑖,𝑐 — предсказанная вероятность того, что i-я точка принадлежит классу c:
Ветвь смещения изучает вектор смещения от каждой точки к центроиду соответствующего экземпляра, с двумя функциями потерь следующим образом, где 𝑜𝑖 — предсказанный вектор смещения для i-й точки, и 𝑜𝑖̂ — истинный вектор смещения для i-й точки:
ScoreNet отвечает за предсказание оценки кандидатных кластеров, с функцией потерь следующим образом, где M — количество кандидатных кластеров, 𝑦𝑗 — истинная метка j-го кластера (1 для действительных кластеров, 0 для недействительных), и 𝑝𝑗 — предсказанная вероятность того, что j-й кластер действителен:
Комбинируя вышеуказанные функции потерь, общая функция потерь сети равна
На этапе вывода к C и полученным оценкам 𝑆𝑐 применяется Non-Maximum Suppression (NMS) для фильтрации низкокачественных кластеров, что дает окончательный результат инстанс-сегментации G.
2.3.2. Метрики оценки
Производительность сети в основном оценивается с точки зрения инстанс-сегментации и семантической сегментации. Для анализа семантической сегментации используются количественные метрики, такие как точность (Prec), полнота (Rec), F1-мера и пересечение по объединению (IoU). Здесь 𝑇𝑃, 𝐹𝑃 и 𝐹𝑁 представляют истинно положительные, ложноположительные и ложноотрицательные результаты соответственно, а n представляет количество экземпляров:
В инстанс-сегментации производительность инстанс-сегментации органов сои оценивается с использованием средней точности (AP) и средней полноты (AR). Метрики AP@25, AP@50, AR@25 и AR@50 [59] указывают оценки, когда порог IoU установлен на 25% и 50% соответственно.
2.4. Извлечение морфологических параметров
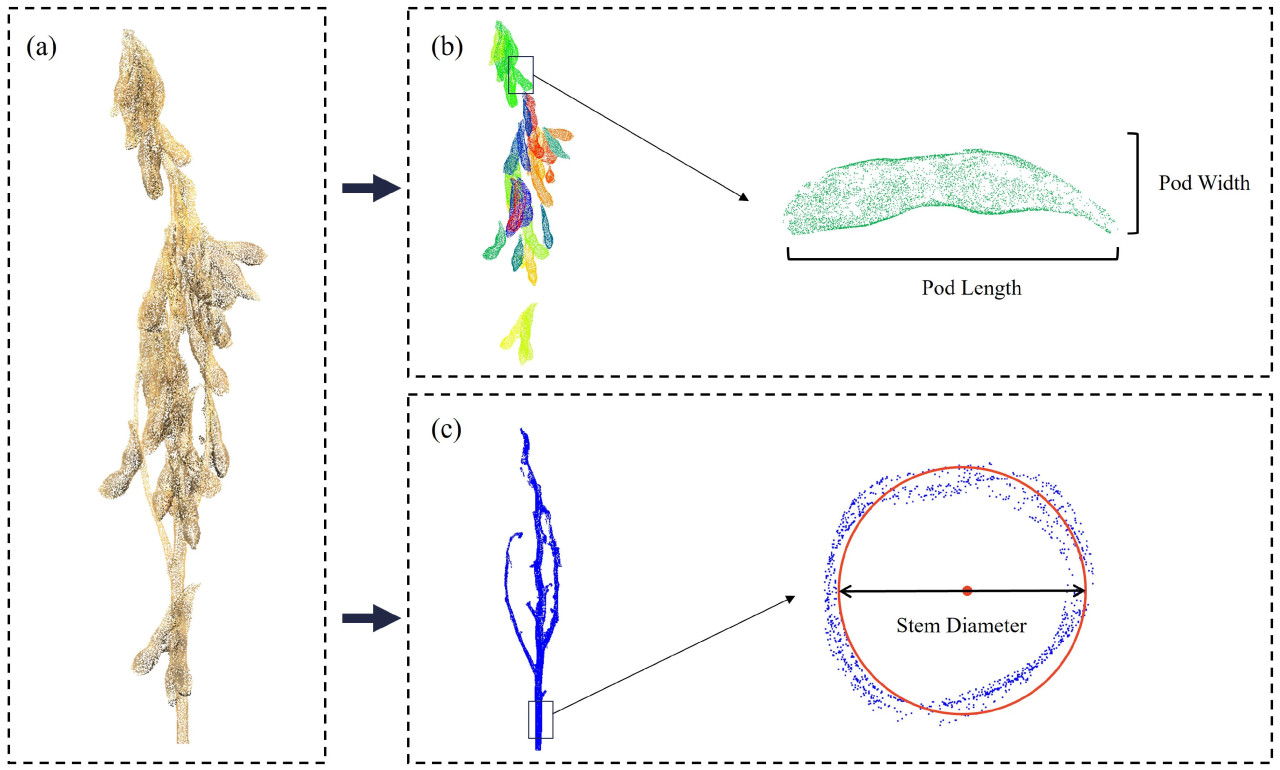
На основе данных облака точек сои мы использовали сеть для извлечения областей облака точек, соответствующих стеблю и стручкам сои, а затем определили фенотипические параметры, такие как длина стручка, ширина стручка и диаметр стебля, с использованием конкретных методов. Последовательность обработки проиллюстрирована на Рисунке 7. Для извлечения диаметра стебля мы выбрали точки в пределах самого нижнего 1 см участка главного стебля. Эти точки проецировались на плоскость 𝑥𝑦, и с помощью метода Пратта [60] подбирался круг для расчета диаметра, таким образом получая диаметр стебля. Для извлечения длины и ширины стручка мы сначала выполнили анализ главных компонент (PCA), чтобы идентифицировать три главные оси облака точек каждого стручка [61]. Мы отметили конечные точки вдоль первой главной оси и рассчитали длину стручка, определив кратчайший путь между этими конечными точками. Далее мы разделили облако точек стручка на пять секций вдоль вектора первой главной компоненты. Внутри каждой секции мы определили конечные точки вдоль второй и третьей главных компонентных векторов, причем самый длинный кратчайший путь был определен как ширина стручка. Наконец, мы оценили точность извлечения фенотипических признаков с использованием коэффициента детерминации (𝑅2) и среднеквадратичной ошибки (RMSE), рассчитанных по следующим формулам:
где 𝑒𝑖 и 𝑒̂𝑖 представляют истинные и предсказанные значения, 𝑒̲𝑖 — среднее истинных значений, а m представляет количество сравниваемых образцов.
Рисунок 7. Иллюстрация извлечения фенотипических параметров: (a) облако точек сои, (b) инстанс-сегментация стручков сои с разными цветами, представляющими разные стручки, затем извлечение фенотипических параметров длины и ширины стручка для каждого стручка, (c) сегментированная область стебля с фенотипическими параметрами диаметра стебля, извлеченными с помощью геометрического подбора.
3. Результаты
3.1. Экспериментальная установка
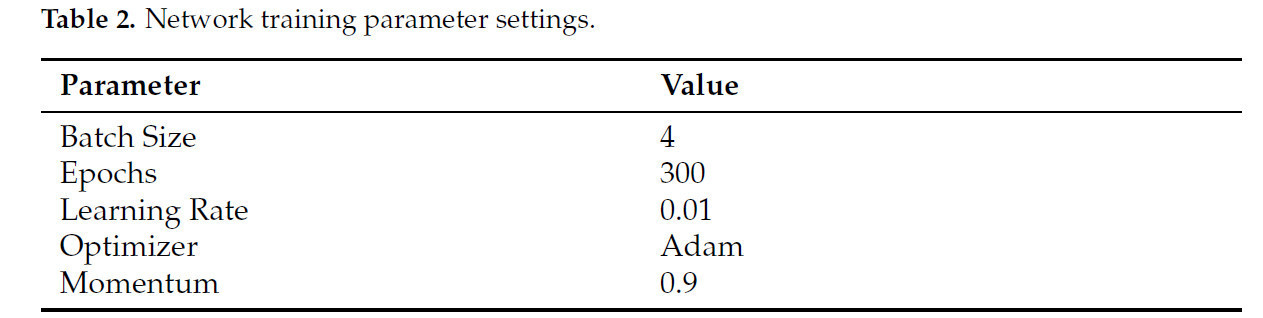
В наших экспериментах расширенные данные облака точек сои были разделены на обучающую, проверочную и тестовую выборки в соотношении 7:1:2. Все эксперименты проводились на графическом процессоре NVIDIA GeForce RTX 3090, и сеть была построена и обучена с использованием библиотеки PyTorch. Структура сети PVSegNet является улучшенной версией на основе PointNet++, с ReLU в качестве функции активации. Входные и выходные каналы показаны на Рисунке 6, в то время как другие параметры используют настройки по умолчанию модуля. Перед подачей в сеть каждый канал данных был нормализован. Как показано в Таблице 2, параметры обучения были установлены с оптимизатором Adam, и сеть обучалась в общей сложности 300 эпох с размером пакета 4. Скорость обучения изначально была установлена на 0,01 и оптимизирована с использованием косинусного затухания. Весовой коэффициент затухания установлен на 0,0001. Мы сохранили параметры сети, соответствующие эпохе с наивысшим IoU на проверочной выборке, для последующего тестирования производительности. Кроме того, случайное начальное число было зафиксировано для обеспечения воспроизводимости экспериментов.
Таблица 2. Настройки параметров обучения сети.
3.2. Абляционное исследование эффективности метода
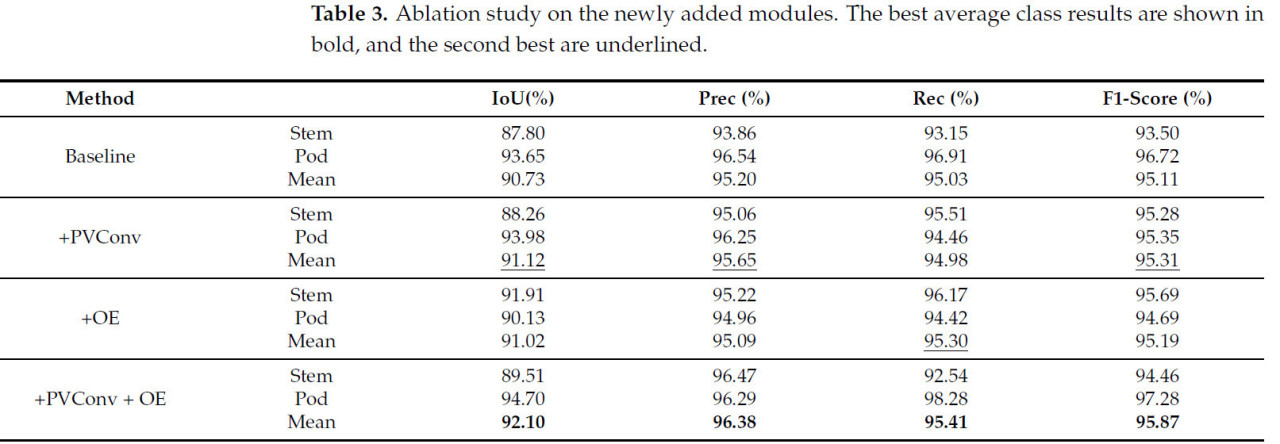
Чтобы проверить эффективность каждого модуля в методе, было проведено абляционное исследование, результаты которого представлены в Таблице 3. В таблице показано влияние добавления каждого модуля на производительность сети. С добавлением модулей производительность модели стабильно улучшалась. После добавления модуля PVConv mIoU увеличился на 0,39%, Prec увеличилась на 0,45%, Rec снизилась на 0,05%, а F1-мера увеличилась на 0,2%. После добавления модуля OE IoU увеличился на 0,29%, Prec снизилась на 0,11%, Rec увеличилась на 0,27%, а F1-мера увеличилась на 0,08%. Когда были добавлены и модуль PVConv, и модуль OE, окончательная сеть улучшилась по сравнению с сетью только с модулем PVConv, которая показала снижение Prec, и сетью только с модулем OE, которая показала снижение Rec. В конечном итоге, Prec увеличилась на 1,18%, а Rec увеличилась на 0,38%.
Таблица 3. Абляционное исследование по вновь добавленным модулям. Лучшие средние результаты по классам показаны жирным шрифтом, а вторые лучшие подчеркнуты.
3.3. Результаты сегментации и сравнение
3.3.1. Сравнение методов семантической сегментации
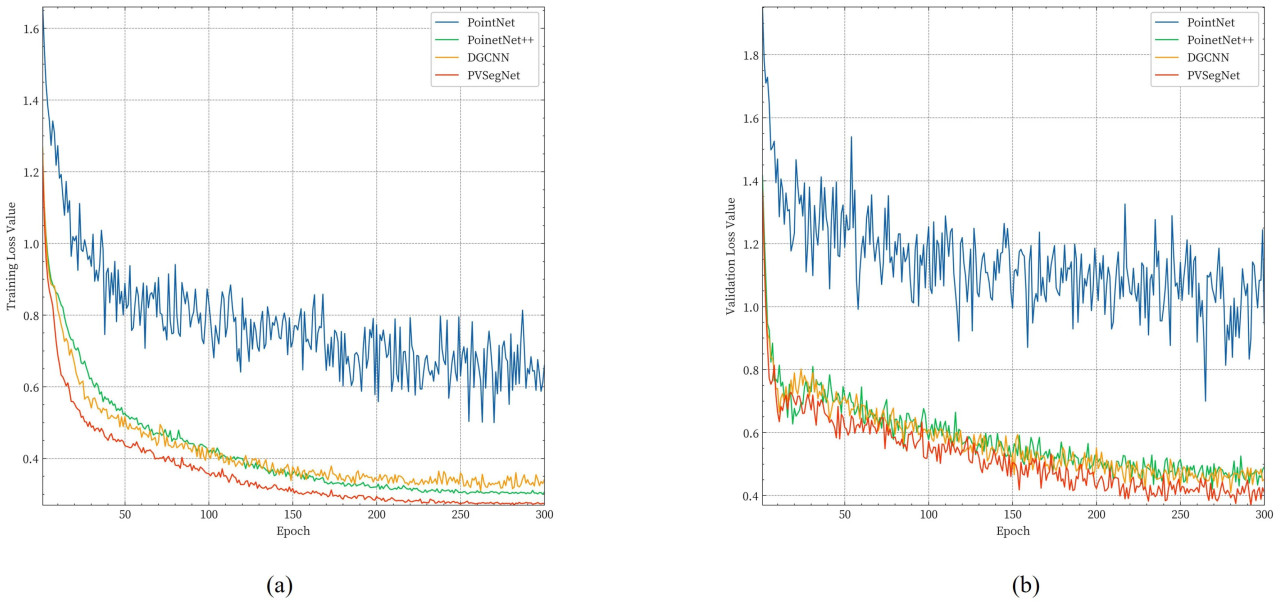
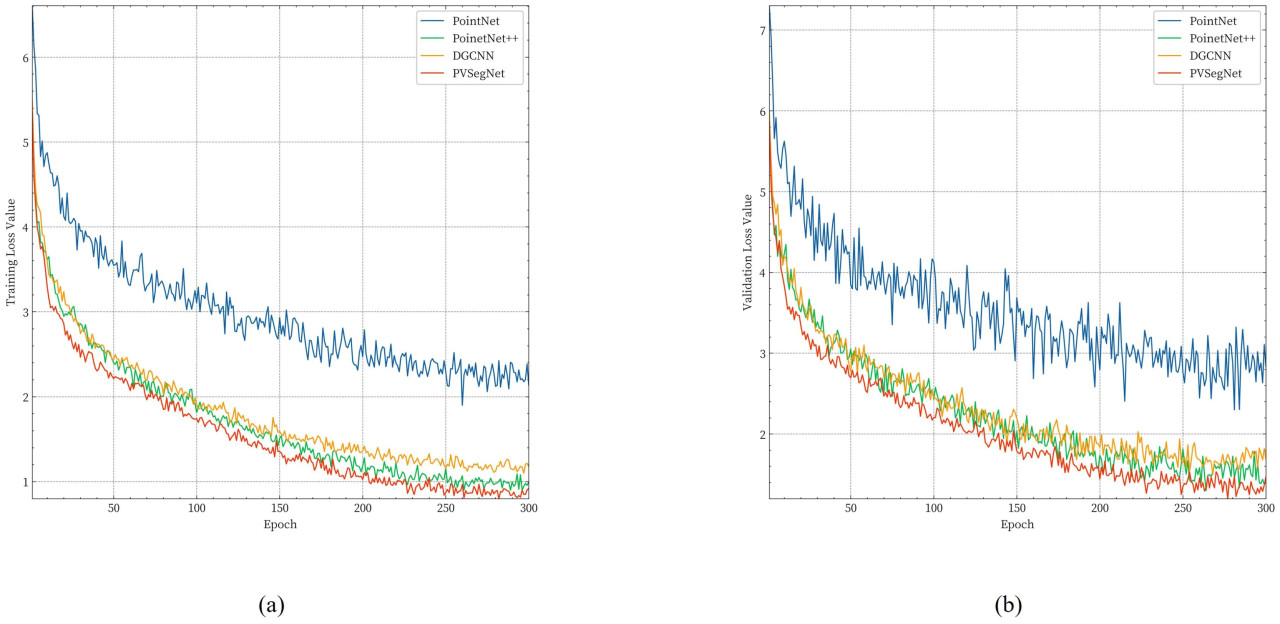
В этом разделе предложенная сеть PVSegNet сравнивается с широко используемыми сетями семантической сегментации облаков точек, включая PointNet [30], PointNet++ [62] и DGCNN [63]. Все сети обучались на одном и том же наборе данных с использованием идентичных техник аугментации данных и конфигураций гиперпараметров. Чтобы оценить производительность каждой сети, мы проанализировали изменения функции потерь в течение фаз обучения и проверки, как показано на Рисунке 8. Мы наблюдали, что значение функции потерь PointNet было очень нестабильным во время обучения. В отличие от этого, значения потерь для других сетей быстро уменьшались в течение первых 120 эпох, затем постепенно стабилизировались и выходили на плато после 200 эпох. Среди всех сетей PVSegNet продемонстрировала самые низкие значения потерь как на обучающей, так и на проверочной выборках, что указывает на лучшую обобщающую способность на проверочном наборе.
Рисунок 8. Изменение потерь при обучении семантической сегментации. (a) Изменение потерь на обучающем наборе. (b) Изменение потерь на проверочном наборе.
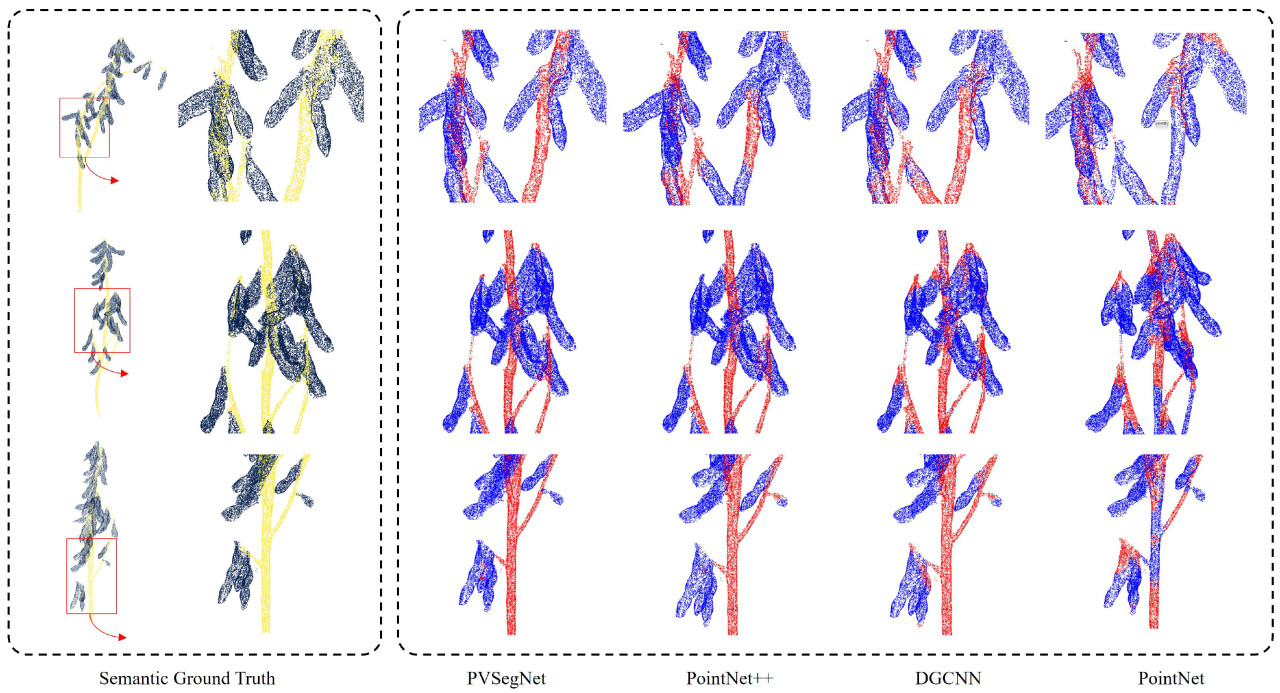
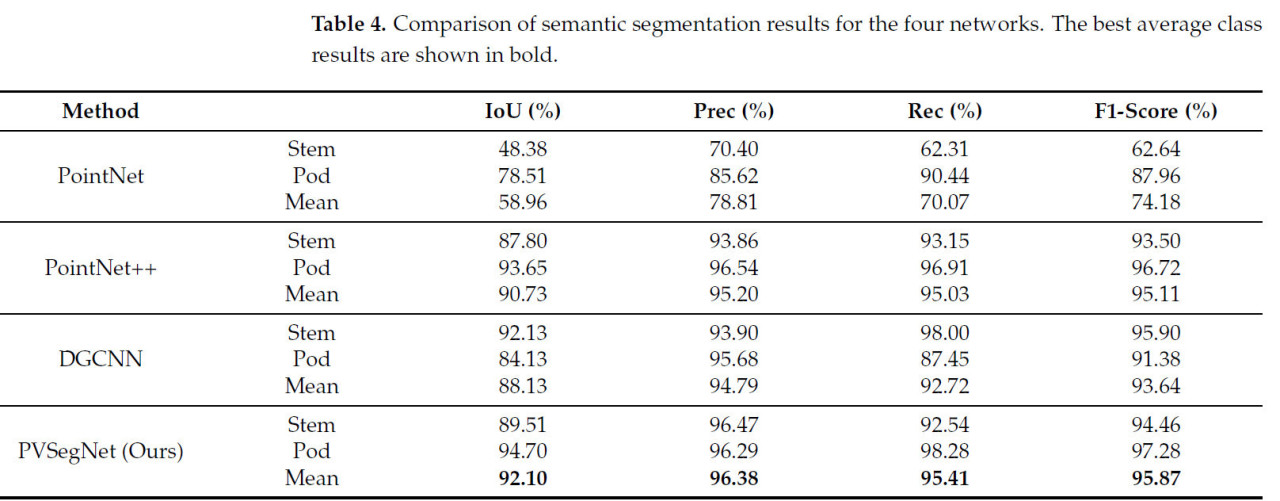
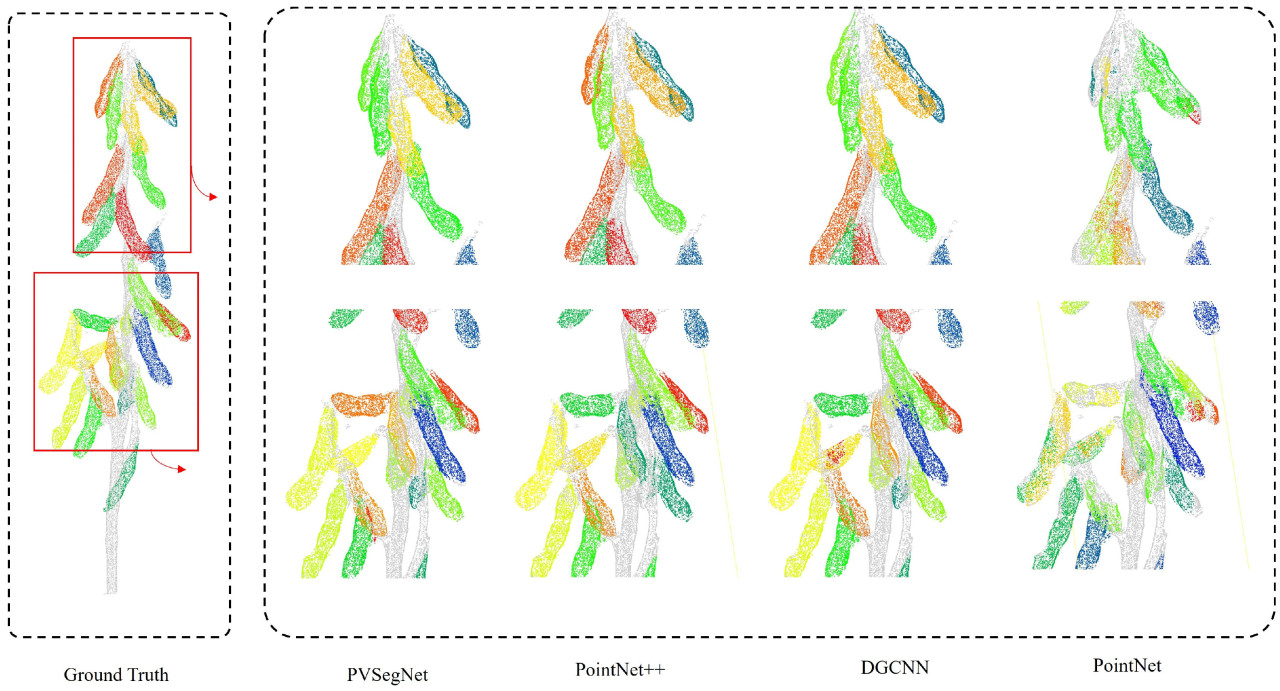
Согласно Таблице 4, PVSegNet продемонстрировала наилучшую производительность, достигнув наивысшего mIoU (92,10%), Prec (96,38%), Rec (95,41%) и F1-меры (95,87%). По сравнению с PointNet++, PVSegNet улучшила показатели на 1,47% по mIoU, на 1,18% по Prec, на 0,38% по Rec и на 0,76% по F1-мере. Как графовый метод, DGCNN достиг результатов mIoU 88,13%, Prec 92,47%, Rec 92,72% и F1-мера 93,64%. PointNet показал худшие результаты, с mIoU всего 58,96%, вероятно, из-за неспособности эффективно извлекать локальные признаки, что затрудняло точную идентификацию стеблей и стручков. На Рисунке 9 представлены предсказания семантической сегментации стручков и стеблей сои в тестовом наборе с использованием четырех методов. Визуально основные различия в результатах предсказания наблюдаются в местах соединений стручков и стеблей и в областях с плотным расположением стручков. Сеть PointNet неправильно классифицировала области стручков и стеблей, в то время как другие сети допустили ошибки классификации на границах. Предсказания PVSegNet были наиболее близки к размеченным данным, демонстрируя более острое восприятие и значительные преимущества, особенно в детальных и граничных областях.
Рисунок 9. Результаты предсказания семантической сегментации четырех сетей. Левая сторона показывает размеченные классы, где желтый и голубой представляют классы стручка и стебля соответственно. Правая сторона обеспечивает четкое отображение локализованных результатов предсказания внутри прямоугольных рамок. В предсказанной метке синий цвет представляет стручки, а красный — стебли.
Таблица 4. Сравнение результатов семантической сегментации для четырех сетей. Лучшие средние результаты по классам показаны жирным шрифтом.
3.3.2. Сравнение методов инстанс-сегментации
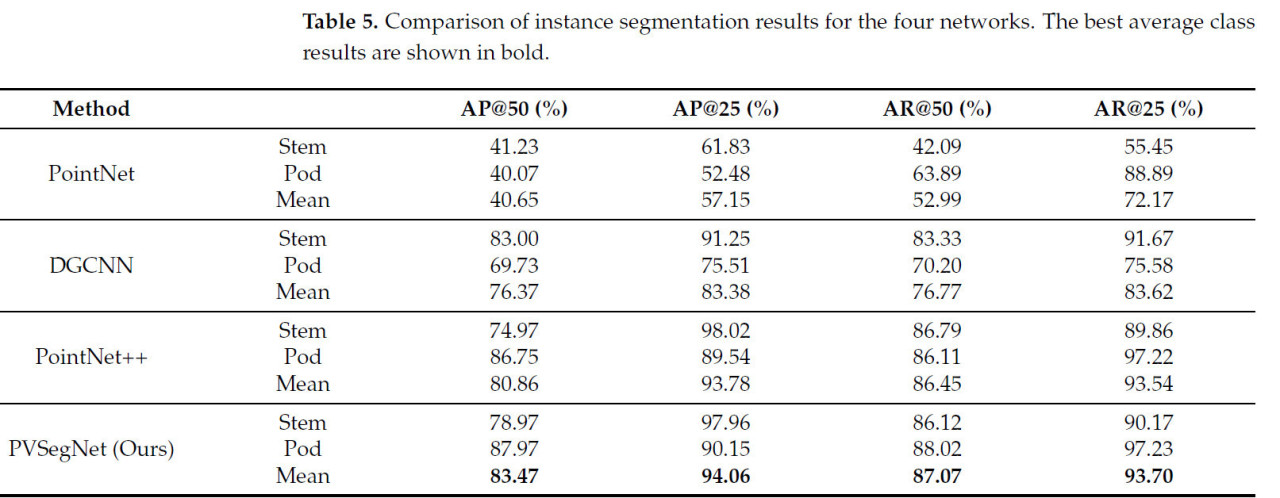
Этот раздел представляет производительность сетей PointNet, PointNet++, DGCNN и PVSegNet в задаче инстанс-сегментации. На Рисунке 10 показаны изменения значений потерь в течение фаз обучения и проверки. Четыре сети были протестированы на инстанс-сегментацию с использованием алгоритма PointGroup, и для обеспечения справедливости при обучении и тестировании использовались параметры по умолчанию. Согласно результатам в Таблице 5, PVSegNet показала наилучшие результаты, достигнув AP@50 83,47%, AP@25 94,06%, AR@50 87,07% и AR@25 93,70%. По сравнению с PointNet++, показатели AP@50, AP@25, AR@50 и AR@25 улучшились на 2,61%, 0,28%, 0,62% и 0,16% соответственно. PointNet показал худшие результаты с AP@50 40,65% и AR@50 52,99%, подчеркивая влияние результатов семантической сегментации на результаты инстанс-сегментации. Графовая сеть DGCNN достигла AP@50 76,37% и AR@50 76,77%. Результаты предсказания инстанс-сегментации PVSegNet визуализированы на Рисунках 11 и 12. Результаты предсказания сети PointNet показали значительные ошибки в инстанс-сегментации из-за влияния семантической сегментации. В сетях PointNet++ и DGCNN стручки сегментировались как экземпляры, но были области на перекрытиях и границах, которые были неправильно идентифицированы как стебли. Результаты PVSegNet наиболее точно соответствовали аннотациям, предоставляя надежный источник данных для последующего извлечения фенотипов.
Рисунок 10. Изменение потерь при обучении инстанс-сегментации. (a) Изменение потерь на обучающем наборе. (b) Изменение потерь на проверочном наборе.
Рисунок 11. На рисунке показаны результаты предсказания инстанс-сегментации четырех разных сетей по сравнению с ручными аннотациями. Красная рамка выделяет увеличенную область. Разные цвета представляют разные стручки, а серый представляет стебель.
Рисунок 12. На рисунке показаны результаты предсказания инстанс-сегментации PVSegNet для разных образцов по сравнению с ручными аннотациями. Разные цвета представляют разные стручки, а серый представляет стебель.
Таблица 5. Сравнение результатов инстанс-сегментации для четырех сетей. Лучшие средние результаты по классам показаны жирным шрифтом.
3.4. Результаты извлечения фенотипических параметров
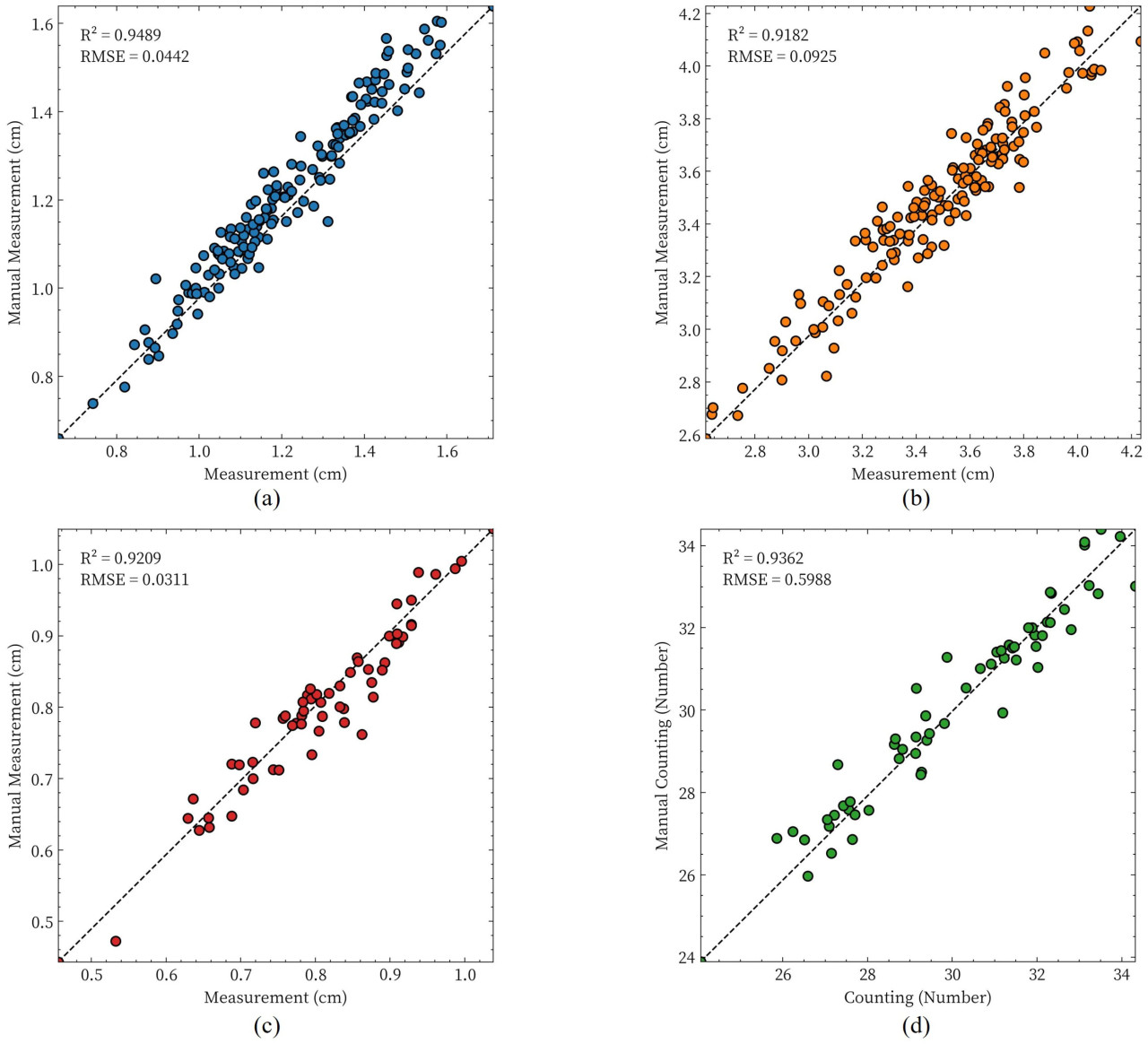
Мы оценили точность предложенного метода для извлечения фенотипических параметров стручков и стебля, сравнив результаты сегментации облака точек с ручными измерениями. Для экспериментальных материалов были измерены количество стручков и диаметр стебля у 60 растений сои. Дополнительно, 150 стручков были случайным образом отобраны для сравнения их длин и ширин. Как показано на Рисунке 13d, результаты проверки для подсчета стручков показывают 𝑅2, равный 0,9362, и RMSE, равный 0,5988. Рисунки 13a–c отображают результаты проверки для ширины стручка, длины стручка и толщины стебля, со значениями 𝑅2, равными 0,9489, 0,9182 и 0,9209 соответственно, и значениями RMSE, равными 0,0442 см, 0,0925 см и 0,0311 см соответственно. Оценочные результаты для длины стручка были ниже по сравнению с результатами для ширины стручка. Эти результаты указывают, что предложенный метод может соответствовать требованиям автоматизированного извлечения фенотипических параметров.
Рисунок 13. Сравнение между фенотипическими параметрами, извлеченными из сегментации облака точек растения сои, и измеренными значениями. (a) Ширина стручка, (b) длина стручка, (c) толщина стебля и (d) количество стручков.
4. Обсуждение
4.1. Генерация облака точек
Было показано, что генерация высококачественных облаков точек улучшает производительность нейронных сетей, смягчает проблемы сегментации облаков точек и повышает точность результатов извлечения фенотипов [64]. В этом исследовании мы разработали многоракурсную схему сбора и использовали специально построенную платформу для получения многоракурсных изображений. Посредством технологии многоракурсной 3D-реконструкции мы успешно построили высококачественный набор данных облака точек сои, который соответствует требованиям для получения фенотипических параметров. Однако факторы окружающей среды и степень перекрытия изображений в процессе реконструкции влияли на качество реконструкции, приводя к потенциальным ошибкам в последующей обработке. Чтобы улучшить качество реконструкции, мы контролировали условия освещения и фона, чтобы минимизировать помехи от внешних факторов окружающей среды. Вариации и недостаточность освещения могут вызывать трудности в сопоставлении признаков изображений во время реконструкции, что, в свою очередь, влияет на четкое отображение признаков растения. При построении платформы для многоракурсного сбора мы использовали черную светопоглощающую ткань в качестве фона для устранения теней растений и обеспечения того, чтобы только интересующий объект появлялся на изображениях. Основной целью этого подхода было минимизировать влияние внешних объектов на точность реконструкции. Путем регулировки высоты съемки и интервалов съемки поворотного стола мы увеличили перекрытие сбора изображений, тем самым обеспечивая качество реконструкции.
4.2. Понижающая дискретизация количества точек облака
Количество точек в облаке точек существенно влияет на производительность сетей глубокого обучения. Большее количество точек, как правило, улучшает точность и полноту сети, но также увеличивает вычислительную сложность и требования к хранению [65,66]. Чтобы выбрать подходящее количество точек, которое балансирует точность и скорость, мы использовали метод случайной понижающей дискретизации для сокращения облака точек до разного количества (4096, 6144, 10 240 и 12 288 точек). Эксперименты по семантической сегментации проводились с размером пакета 4, в то время как другие параметры оставались согласованными с Разделом 3.1. Результаты, показанные в Таблице 6, указывают, что увеличение количества точек после понижающей дискретизации положительно влияет на производительность сети. Это улучшение можно объяснить усиленной информацией и плотностью признаков [63], позволяя сети улавливать более сложные детали. Из-за вычислительных ограничений мы не тестировали ввод с большим количеством точек. Примечательно, что по мере увеличения количества точек скорость вывода сети имеет тенденцию снижаться, чтобы отдать приоритет более высокой точности, чтобы достичь баланса между точностью и скоростью. Следовательно, в практических приложениях крайне важно выбрать количество точек после понижающей дискретизации, которое оптимизирует баланс между точностью предсказания сети и эффективностью, принимая во внимание доступную вычислительную мощность, определяемую конфигурацией аппаратного обеспечения.
Таблица 6. Результаты семантической сегментации PVSegNet при разном количестве точек.
4.3. Будущая работа
Это исследование подтвердило осуществимость извлечения фенотипических параметров стручков и стеблей сои на основе 3D-алгоритмов сегментации. Предложенная сеть PVSegNet значительно улучшила точность сегментации, комбинируя признаки облака точек и вокселей. Учитывая аппаратные ограничения и потребность в сценариях оценки в реальном времени, будущие исследования будут изучать легковесные функциональные модули для повышения скорости вывода сети. Более того, разнообразие и масштаб набора данных крайне важны для обучения сети. В последующих исследованиях мы планируем расширить выборки данных, используя разные материалы, чтобы дополнительно повысить производительность и устойчивость сети, тем самым обеспечивая больший потенциальный потенциал для применения технологий точного земледелия.
5. Выводы
В заключение, в этом исследовании был разработан новый пайплайн для получения фенотипических признаков созревшей сои на основе 3D облаков точек и предложена сеть PVSegNet на основе глубокого обучения, интегрирующая свертку облака точек и вокселей для улучшения извлечения признаков для сегментации стручков и стеблей сои. Экспериментальные результаты продемонстрировали IoU 92,10%, точность 96,38%, полноту 95,41% и F1-меру 95,87% в семантической сегментации, и AP@50 83,47% и AR@50 87,07% в инстанс-сегментации. Результаты проверки для ширины стручка, длины стручка и толщины стебля показали значения 𝑅2, равные 0,9489, 0,9182 и 0,9209 соответственно. Будущие исследования будут отдавать приоритет разработке легковесных модулей для более быстрого вывода и расширению разнообразных наборов данных для улучшения производительности сети и приложений точного земледелия. Это исследование вносит вклад в область фенотипирования растений и предоставляет ценные идеи для будущего развития технологий точного земледелия.
Ссылки
1. Jiang, G.; Chen, P.; Zhang, J.; Florez-Palacios, L.; Zeng, A.; Wang, X.; Bowen, R.A.; Miller, A.M.; Berry, H. Genetic Analysis of Sugar Composition and Its Relationship with Protein, Oil, and Fiber in Soybean. Crop Sci. 2018, 58, 2413–2421. [Google Scholar] [CrossRef]
2. Florou-Paneri, P.; Christaki, E.; Giannenas, I.; Bonos, E.; Skoufos, I.; Tsinas, A.; Tzora, A.; Peng, J. Alternative Protein Sources to Soybean Meal in Pig Diets. J. Food Agric. Environ. 2014, 12, 655–660. [Google Scholar]
3. Zhang, M.; Liu, S.; Wang, Z.; Yuan, Y.; Zhang, Z.; Liang, Q.; Yang, X.; Duan, Z.; Liu, Y.; Kong, F.; et al. Progress in Soybean Functional Genomics over the Past Decade. Plant Biotechnol. J. 2021, 20, 256–282. [Google Scholar] [CrossRef] [PubMed]
4. Bao, X.; Yao, X. Genetic Improvements in the Root Traits and Fertilizer Tolerance of Soybean Varieties Released during Different Decades. Agronomy 2023, 14, 2. [Google Scholar] [CrossRef]
5. Chang, F.; Lv, W.; Lv, P.; Xiao, Y.; Yan, W.; Chen, S.; Zheng, L.; Xie, P.; Wang, L.; Karikari, B.; et al. Exploring Genetic Architecture for Pod-Related Traits in Soybean Using Image-Based Phenotyping. Mol. Breed. 2021, 41, 28. [Google Scholar] [CrossRef] [PubMed]
6. Parmley, K.A.; Nagasubramanian, K.; Sarkar, S.; Ganapathysubramanian, B.; Singh, A.K. Development of Optimized Phenomic Predictors for Efficient Plant Breeding Decisions Using Phenomic-Assisted Selection in Soybean. Plant Phenomics 2019, 2019, 5809404. [Google Scholar] [CrossRef] [PubMed]
7. Chouhan, S.S.; Singh, U.; Jain, S. Applications of Computer Vision in Plant Pathology: A Survey. Arch. Comput. Methods Eng. 2020, 27, 611–632. [Google Scholar] [CrossRef]
8. Ngugi, L.C.; Abdelwahab, M.; Abo-Zahhad, M. Tomato Leaf Segmentation Algorithms for Mobile Phone Applications Using Deep Learning. Comput. Electron. Agric. 2020, 178, 105788. [Google Scholar] [CrossRef]
9. Miao, C.; Guo, A.; Thompson, A.M.; Yang, J.; Ge, Y.; Schnable, J.C. Automation of Leaf Counting in Maize and Sorghum Using Deep Learning. Plant Phenome J. 2021, 4, e20022. [Google Scholar] [CrossRef]
10. Zhang, Z.; Pope, M.; Shakoor, N.; Pless, R.; Mockler, T.; Stylianou, A. Comparing Deep Learning Approaches for Understanding Genotype × Phenotype Interactions in Biomass Sorghum. Front. Artif. Intell. 2022, 5, 872858. [Google Scholar] [CrossRef] [PubMed]
11. Li, R.; Wang, D.; Zhu, B.; Liu, T.; Sun, C.; Zhang, Z. Estimation of Nitrogen Content in Wheat Using Indices Derived from RGB and Thermal Infrared Imaging. Field Crops Res. 2022, 289, 108735. [Google Scholar] [CrossRef]
12. Wu, T.; Shen, P.; Dai, J.; Ma, Y.; Feng, Y. A Pathway to Assess Genetic Variation of Wheat Germplasm by Multidimensional Traits with Digital Images. Plant Phenomics 2023, 5, 119. [Google Scholar] [CrossRef] [PubMed]
13. Feldmann, M.J.; Tabb, A. Cost-Effective, High-Throughput Phenotyping System for 3D Reconstruction of Fruit Form. bioRxiv 2021. [Google Scholar] [CrossRef]
14. Wu, S.; Wen, W.; Gou, W.; Lu, X.; Zhang, W.; Zheng, C.; Xiang, Z.; Chen, L.; Guo, X. A Miniaturized Phenotyping Platform for Individual Plants Using Multi-View Stereo 3D Reconstruction. Front. Plant Sci. 2022, 13, 897746. [Google Scholar] [CrossRef] [PubMed]
15. Ziamtsov, I.; Navlakha, S. Plant 3D (P3D): A Plant Phenotyping Toolkit for 3D Point Clouds. Bioinformatics 2020, 36, 3949–3950. [Google Scholar] [CrossRef] [PubMed]
16. Qiu, Q.; Sun, N.; Bai, H.; Wang, N.; Fan, Z.; Wang, Y.; Meng, Z.; Li, B.; Cong, Y. Field-Based High-Throughput Phenotyping for Maize Plant Using 3D LiDAR Point Cloud Generated With a “Phenomobile”. Front. Plant Sci. 2019, 10, 554. [Google Scholar] [CrossRef] [PubMed]
17. Rivera, G.; Porras, R.; Florencia, R.; Sanchez-Solis, J.P. LiDAR Applications in Precision Agriculture for Cultivating Crops: A Review of Recent Advances. Comput. Electron. Agric. 2023, 207, 107737. [Google Scholar] [CrossRef]
18. Jin, S.; Sun, X.; Wu, F.; Su, Y.; Li, Y.; Song, S.; Xu, K.; Ma, Q.; Baret, F.; Jiang, D.; et al. Lidar Sheds New Light on Plant Phenomics for Plant Breeding and Management: Recent Advances and Future Prospects. ISPRS-J. Photogramm. Remote Sens. 2021, 171, 202–223. [Google Scholar] [CrossRef]
19. Song, P.; Li, Z.; Yang, M.; Shao, Y.; Pu, Z.; Yang, W.; Zhai, R. Dynamic Detection of Three-Dimensional Crop Phenotypes Based on a Consumer-Grade RGB-D Camera. Front. Plant Sci. 2023, 14, 1097725. [Google Scholar] [CrossRef] [PubMed]
20. Wang, H.; Duan, Y.; Shi, Y.; Kato, Y.; Ninomiya, S.; Guo, W. EasyIDP: A Python Package for Intermediate Data Processing in UAV-Based Plant Phenotyping. Remote Sens. 2021, 13, 2622. [Google Scholar] [CrossRef]
21. Sun, Y.; Zhang, Z.; Sun, K.; Li, S.; Yu, J.; Miao, L.; Zhang, Z.; Li, Y.; Zhao, H.; Hu, Z.; et al. Soybean-MVS: Annotated Three-Dimensional Model Dataset of Whole Growth Period Soybeans for 3D Plant Organ Segmentation. Agriculture 2023, 13, 1321. [Google Scholar] [CrossRef]
22. Luo, L.; Jiang, X.; Yang, Y.; Samy, E.R.A.; Lefsrud, M.; Hoyos-Villegas, V.; Sun, S. Eff-3DPSeg: 3D Organ-Level Plant Shoot Segmentation Using Annotation-Efficient Deep Learning. Plant Phenomics 2023, 5, 80. [Google Scholar] [CrossRef] [PubMed]
23. Lin, Y.; Ruifang, Z.; Pujuan, S.; Pengfei, W. Segmentation of Crop Organs through Region Growing in 3D Space. In Proceedings of the 2016 Fifth International Conference on Agro-Geoinformatics (Agro-Geoinformatics), Tianjin, China, 18–20 July 2016; pp. 1–6. [Google Scholar] [CrossRef]
24. Cuevas-Velasquez, H.; Gallego, A.J.; Fisher, R.B. Segmentation and 3D Reconstruction of Rose Plants from Stereoscopic Images. Comput. Electron. Agric. 2020, 171, 105296. [Google Scholar] [CrossRef]
25. Vo, A.; Truong-Hong, L.; Laefer, D.; Bertolotto, M. Octree-Based Region Growing for Point Cloud Segmentation. ISPRS J. Photogramm. Remote Sens. 2015, 104, 88–100. [Google Scholar] [CrossRef]
26. Han, H.; Han, X.; Gao, T. 3D Mesh Model Segmentation Based on Skeleton Extraction. Imaging Sci. J. 2021, 69, 153–163. [Google Scholar] [CrossRef]
27. Dutagaci, H.; Rasti, P.; Galopin, G.; Rousseau, D. ROSE-X: An Annotated Data Set for Evaluation of 3D Plant Organ Segmentation Methods. Plant Methods 2020, 16, 1–14. [Google Scholar] [CrossRef] [PubMed]
28. Chaudhury, A.; Ward, C.D.W.; Talasaz, A.; Ivanov, A.; Brophy, M.; Grodzinski, B.; Hüner, N.; Patel, R.V.; Barron, J. Machine Vision System for 3D Plant Phenotyping. IEEE/ACM Trans. Comput. Biol. Bioinform. 2017, 16, 2009–2022. [Google Scholar] [CrossRef] [PubMed]
29. Mostafa, S.; Mondal, D.; Panjvani, K.; Kochian, L.; Stavness, I. Explainable Deep Learning in Plant Phenotyping. Front. Artif. Intell. 2023, 6, 1203546. [Google Scholar] [CrossRef]
30. Charles, R.Q.; Su, H.; Kaichun, M.; Guibas, L.J. PointNet: Deep Learning on Point Sets for 3D Classification and Segmentation. In Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Honolulu, HI, USA, 21–26 July 2017; pp. 77–85. [Google Scholar] [CrossRef]
31. Li, Y.; Wen, W.; Miao, T.; Wu, S.; Yu, Z.; Wang, X.; Guo, X.; Zhao, C. Automatic Organ-Level Point Cloud Segmentation of Maize Shoots by Integrating High-Throughput Data Acquisition and Deep Learning. Comput. Electron. Agric. 2022, 193, 106702. [Google Scholar] [CrossRef]
32. Li, D.; Shi, G.; Li, J.; Chen, Y.; Zhang, S.; Xiang, S.; Jin, S. PlantNet: A Dual-Function Point Cloud Segmentation Network for Multiple Plant Species. ISPRS J. Photogramm. Remote Sens. 2022, 184, 243–263. [Google Scholar] [CrossRef]
33. Zhou, Y.; Tuzel, O. VoxelNet: End-to-End Learning for Point Cloud Based 3D Object Detection. In Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition, Salt Lake City, UT, USA, 18–22 June 2018; pp. 4490–4499. [Google Scholar] [CrossRef]
34. Jin, S.; Su, Y.; Gao, S.; Wu, F.; Ma, Q.; Xu, K.; Ma, Q.; Hu, T.; Liu, J.; Pang, S.; et al. Separating the Structural Components of Maize for Field Phenotyping Using Terrestrial LiDAR Data and Deep Convolutional Neural Networks. IEEE Trans. Geosci. Remote Sens. 2020, 58, 2644–2658. [Google Scholar] [CrossRef]
35. Li, D.; Li, J.; Xiang, S.; Pan, A. PSegNet: Simultaneous Semantic and Instance Segmentation for Point Clouds of Plants. Plant Phenomics 2022, 2022. [Google Scholar] [CrossRef] [PubMed]
36. Mukherjee, S.; Lu, D.; Raghavan, B.; Breitkopf, P.; Dutta, S.; Xiao, M.; Zhang, W. Accelerating Large-scale Topology Optimization: State-of-the-Art and Challenges. Arch. Comput. Methods Eng. 2021, 28, 4549–4571. [Google Scholar] [CrossRef]
37. Howe, M.; Repasky, B.; Payne, T. Effective Utilisation of Multiple Open-Source Datasets to Improve Generalisation Performance of Point Cloud Segmentation Models. In Proceedings of the 2022 International Conference on Digital Image Computing: Techniques and Applications (DICTA), Sydney, Australia, 30 November–2 December 2022; pp. 1–7. [Google Scholar] [CrossRef]
38. Schunck, D.; Magistri, F.; Rosu, R.; Cornelissen, A.; Chebrolu, N.; Paulus, S.; Léon, J.; Behnke, S.; Stachniss, C.; Kuhlmann, H.; et al. Pheno4D: A Spatio-Temporal Dataset of Maize and Tomato Plant Point Clouds for Phenotyping and Advanced Plant Analysis. PLoS ONE 2021, 16, e0256340. [Google Scholar] [CrossRef]
39. Boogaard, F.P.; van Henten, E.J.; Kootstra, G. Improved Point-Cloud Segmentation for Plant Phenotyping Through Class-Dependent Sampling of Training Data to Battle Class Imbalance. Front. Plant Sci. 2022, 13, 838190. [Google Scholar] [CrossRef] [PubMed]
40. Zhu, B.; Liu, F.; Che, Y.; Hui, F.; Ma, Y. Three-Dimensional Quantification of Intercropping Crops in Field by Ground and Aerial Photography. In Proceedings of the 2018 6th International Symposium on Plant Growth Modeling, Simulation, Visualization and Applications (PMA), Hefei, China, 4–8 November 2018; pp. 1–5. [Google Scholar] [CrossRef]
41. Chakraborty, S.; Banerjee, A.; Gupta, S.; Christensen, P. Region of Interest Aware Compressive Sensing of THEMIS Images and Its Reconstruction Quality. In Proceedings of the 2018 IEEE Aerospace Conference, Big Sky, MT, USA, 3–10 March 2018; pp. 1–11. [Google Scholar] [CrossRef]
42. López-Torres, C.V.; Salazar-Colores, S.; Kells, K.; Ortega, J.; Arreguín, J.M.R. Improving 3D Reconstruction Accuracy in Wavelet Transform Profilometry by Reducing Shadow Effects. IET Image Process. 2020, 14, 310–317. [Google Scholar] [CrossRef]
43. Liu, Y.; Dai, Q.; Xu, W. A Point-Cloud-Based Multiview Stereo Algorithm for Free-Viewpoint Video. IEEE Trans. Vis. Comput. Graph. 2010, 16, 407–418. [Google Scholar] [CrossRef] [PubMed]
44. Condorelli, F.; Higuchi, R.; Nasu, S.; Rinaudo, F.; Sugawara, H. Improving Performance Of Feature Extraction In Sfm Algorithms For 3d Sparse Point Cloud. ISPRS Int. Arch. Photogramm. Remote Sens. Spat. Inf. Sci. 2019, 42, 101–106. [Google Scholar] [CrossRef]
45. Dascăl, A.; Popa, M. Possibilities of 3D Reconstruction of the Vehicle Collision Scene in the Photogrammetric Environment Agisoft Metashape 1.6.2. J. Physics: Conf. Ser. 2021, 1781, 012053. [Google Scholar] [CrossRef]
46. Kim, J.; Ko, Y.; Seo, J. Construction of Machine-Labeled Data for Improving Named Entity Recognition by Transfer Learning. IEEE Access 2020, 8, 59684–59693. [Google Scholar] [CrossRef]
47. Armeni, I.; Sener, O.; Zamir, A.R.; Jiang, H.; Brilakis, I.; Fischer, M.; Savarese, S. 3D Semantic Parsing of Large-Scale Indoor Spaces. In Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, Las Vegas, NV, USA, 27–30 June 2016; pp. 1534–1543. [Google Scholar]
48. Zhang, H.; Zhu, L.; Cai, X.; Dong, L. Noise Removal Algorithm Based on Point Cloud Classification. In Proceedings of the 2022 International Seminar on Computer Science and Engineering Technology (SCSET), Indianapolis, IN, USA, 8–9 January 2022; pp. 93–96. [Google Scholar] [CrossRef]
49. Wu, Z.; Zeng, Y.; Li, D.; Liu, J.; Feng, L. High-Volume Point Cloud Data Simplification Based on Decomposed Graph Filtering. Autom. Constr. 2021, 129, 103815. [Google Scholar] [CrossRef]
50. Chen, S.; Wang, J.; Pan, W.; Gao, S.; Wang, M.; Lu, X. Towards Uniform Point Distribution in Feature-Preserving Point Cloud Filtering. Comput. Vis. Media 2022, 9, 249–263. [Google Scholar] [CrossRef]
51. Zhou, J.; Fu, X.; Zhou, S.; Zhou, J.; Ye, H.; Nguyen, H. Automated Segmentation of Soybean Plants from 3D Point Cloud Using Machine Learning. Comput. Electron. Agric. 2019, 162, 143–153. [Google Scholar] [CrossRef]
52. Cortés-Ciriano, I.; Bender, A. Improved Chemical Structure-Activity Modeling Through Data Augmentation. J. Chem. Inf. Model. 2015, 55 12, 2682–2692. [Google Scholar] [CrossRef]
53. Wang, M.; Ju, M.; Fan, Y.; Guo, S.; Liao, M.; Yang, H.j.; He, D.; Komura, T. 3D Incomplete Point Cloud Surfaces Reconstruction With Segmentation and Feature-Enhancement. IEEE Access 2019, 7, 15272–15281. [Google Scholar] [CrossRef]
54. Xin, B.; Sun, J.; Bartholomeus, H.; Kootstra, G. 3D Data-Augmentation Methods for Semantic Segmentation of Tomato Plant Parts. Front. Plant Sci. 2023, 14, 1045545. [Google Scholar] [CrossRef] [PubMed]
55. Liu, Z.; Tang, H.; Lin, Y.; Han, S. Point-Voxel CNN for Efficient 3D Deep Learning. arXiv 2019, arXiv:cs/1907.03739. [Google Scholar]
56. Jiang, M.; Wu, Y.; Zhao, T.; Zhao, Z.; Lu, C. PointSIFT: A SIFT-like Network Module for 3D Point Cloud Semantic Segmentation. arXiv 2018, arXiv:cs/1807.00652. [Google Scholar]
57. Magistri, F.; Chebrolu, N.; Stachniss, C. Segmentation-Based 4D Registration of Plants Point Clouds for Phenotyping. In Proceedings of the 2020 IEEE/RSJ International Conference on Intelligent Robots and Systems (IROS), Las Vegas, NV, USA, 25–29 October 2020; pp. 2433–2439. [Google Scholar] [CrossRef]
58. Wahabzada, M.; Paulus, S.; Kersting, K.; Mahlein, A.K. Automated Interpretation of 3D Laserscanned Point Clouds for Plant Organ Segmentation. BMC Bioinform. 2015, 16, 1–11. [Google Scholar] [CrossRef]
59. Jiang, L.; Zhao, H.; Shi, S.; Liu, S.; Fu, C.W.; Jia, J. PointGroup: Dual-Set Point Grouping for 3D Instance Segmentation. In Proceedings of the 2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR), Seattle, WA, USA, 13–19 June 2020; pp. 4866–4875. [Google Scholar] [CrossRef]
60. Saeed, F.; Sun, S.; Rodriguez-Sanchez, J.; Snider, J.; Liu, T.; Li, C. Cotton plant part 3D segmentation and architectural trait extraction using point voxel convolutional neural networks. Plant Methods 2023, 19, 33. [Google Scholar] [CrossRef]
61. Miao, T.; Zhu, C.; Xu, T.; Yang, T.; Li, N.; Zhou, Y.; Deng, H. Automatic Stem-Leaf Segmentation of Maize Shoots Using Three-Dimensional Point Cloud. Comput. Electron. Agric. 2021, 187, 106310. [Google Scholar] [CrossRef]
62. Qi, C.R.; Yi, L.; Su, H.; Guibas, L.J. Pointnet++: Deep hierarchical feature learning on point sets in a metric space. Adv. Neural Inf. Process. Syst. 2017, 30. [Google Scholar]
63. Wang, Y.; Sun, Y.; Liu, Z.; Sarma, S.E.; Bronstein, M.M.; Solomon, J.M. Dynamic Graph CNN for Learning on Point Clouds. ACM Trans. Graph. 2019, 38, 146:1–146:12. [Google Scholar] [CrossRef]
64. Rose, J.; Paulus, S.; Kuhlmann, H. Accuracy Analysis of a Multi-View Stereo Approach for Phenotyping of Tomato Plants at the Organ Level. Sensors 2015, 15, 9651–9665. [Google Scholar] [CrossRef] [PubMed]
65. Yu, S.; Wang, M.; Zhang, C.; Li, J.; Yan, K.; Liang, Z.; Wei, R. A Dynamic Multi-Branch Neural Network Module for 3D Point Cloud Classification and Segmentation Using Structural Re-parametertization. In Proceedings of the 2023 11th International Conference on Agro-Geoinformatics (Agro-Geoinformatics), Wuhan, China, 25–28 July 2023; pp. 1–6. [Google Scholar] [CrossRef]
66. Zhai, R.; Li, X.; Wang, Z.; Guo, S.; Hou, S.; Hou, Y.; Gao, F.; Song, J. Point Cloud Classification Model Based on a Dual-Input Deep Network Framework. IEEE Access 2020, 8, 55991–55999. [Google Scholar] [CrossRef]
Cui D, Liu P, Liu Y, Zhao Z, Feng J. Automated Phenotypic Analysis of Mature Soybean Using Multi-View Stereo 3D Reconstruction and Point Cloud Segmentation. Agriculture. 2025; 15(2):175. https://doi.org/10.3390/agriculture15020175
Перевод статьи «Automated Phenotypic Analysis of Mature Soybean Using Multi-View Stereo 3D Reconstruction and Point Cloud Segmentation» авторов Cui D, Liu P, Liu Y, Zhao Z, Feng J., оригинал доступен по ссылке. Лицензия: CC BY. Изменения: переведено на русский язык
Фото: freepik

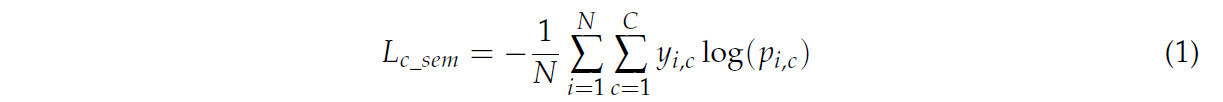
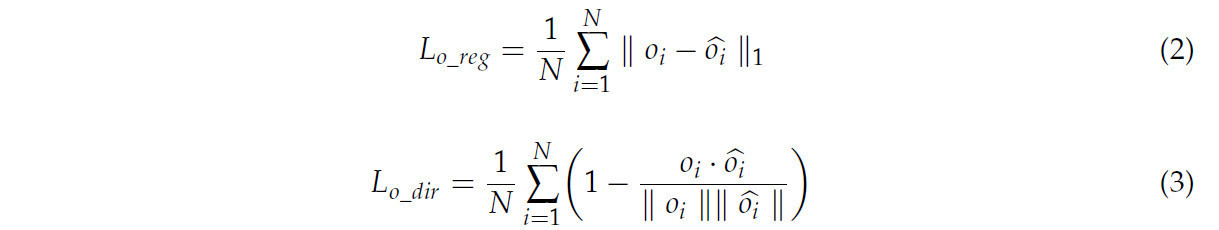
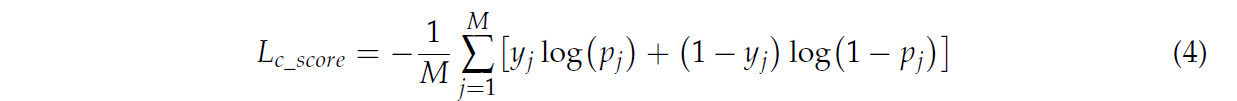
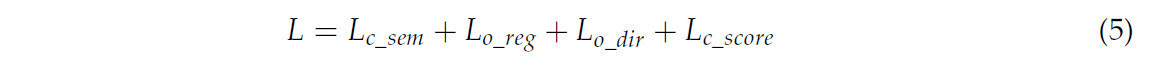

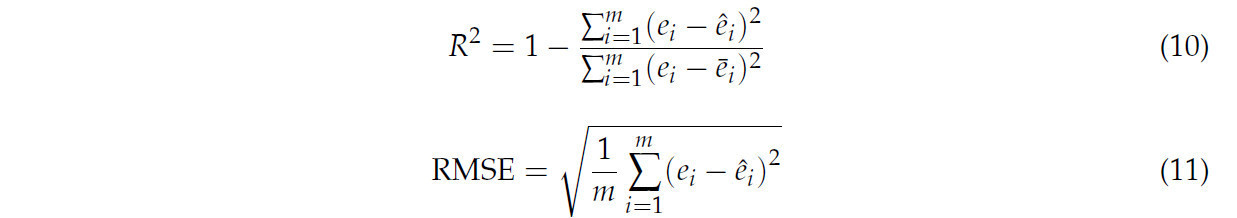
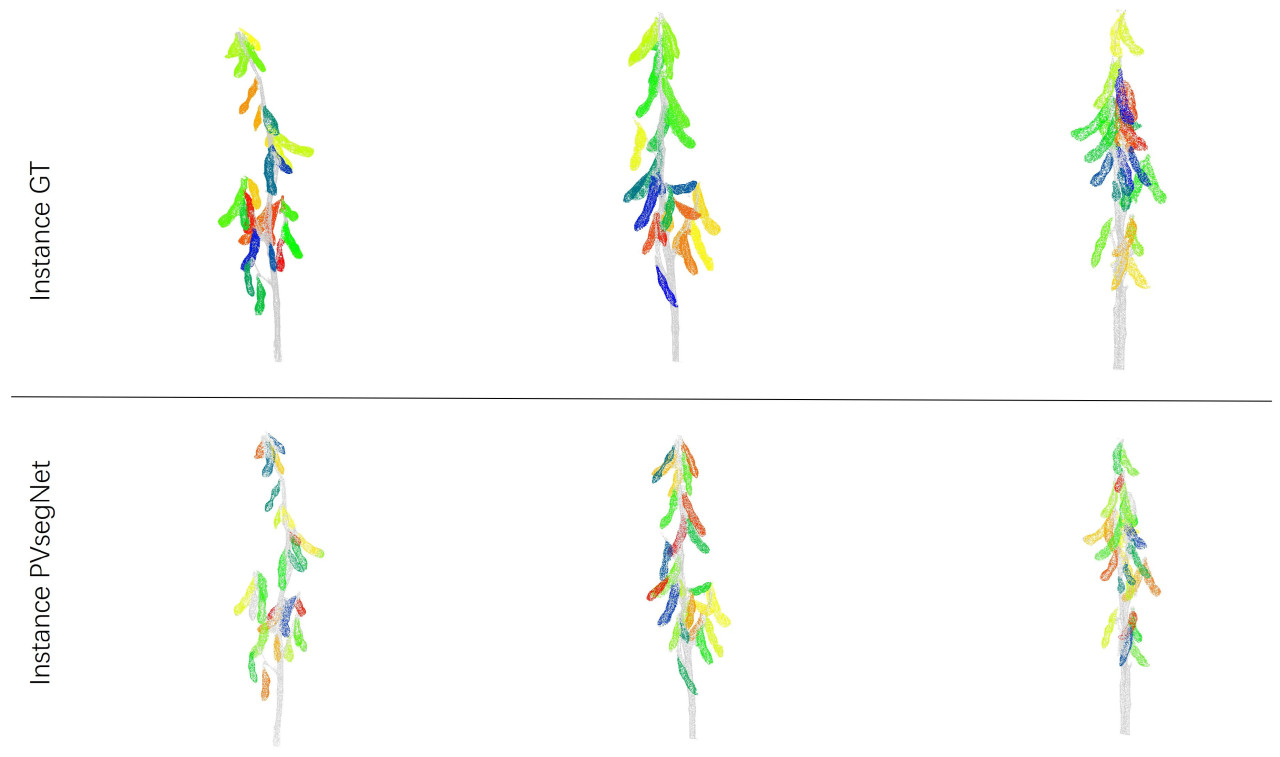































































































Комментарии (0)