Тайная защита огурца: как гены LOX помогают бороться со стрессом
Огурец — культура капризная: жара, холод, мучнистая роса и серая гниль могут уничтожить урожай. Но у растения есть скрытая система защиты. Международная группа исследователей впервые проанализировала пан-геном огурца и нашла 24 гена семейства LOX, которые помогают культуре противостоять стрессам. Один из них, CsLOX22, оказался универсальным «солдатом» — он включается при любых угрозах. Открытие поможет селекционерам создавать сорта, устойчивые к болезням и непогоде без лишней «химии».
Аннотация
Липоксигеназа (LOX) представляет собой диоксигеназу, содержащую негемовое железо, и играет ключевую роль в регуляции роста и развития растений, передачи сигналов, а также в ответах на биотические и абиотические стрессы. В данном исследовании мы идентифицировали 24 гена CsLOX в пан-геноме 12 образцов огурца (Cucumis sativus L.), причем большинство белков CsLOX характеризовались вариациями аминокислотных последовательностей.
Для выяснения их функций мы проанализировали филогенетические связи, структуры генов, консервативные домены, цис-элементы промоторов и коллинеарность 24 генов CsLOX на основе обновленной версии генома 4.0 сорта ‘Chinese Long 9930’. Результаты показали, что CsLOX подразделяются на три подсемейства: 9-LOX, тип I 13-LOX и тип II 13-LOX. Анализ промоторов выявил, что они содержат различные цис-элементы, связанные с ответами на стресс и гормоны. Экспрессия CsLOX демонстрировала тканеспецифичность; каждый ген CsLOX экспрессировался по меньшей мере в одной ткани, а шесть генов CsLOX экспрессировались во всех тканях. Кроме того, в транскриптомных данных ответов огурца на стрессы, вызванные высокой температурой, холодом, мучнистой росой (PM), ложной мучнистой росой (DM) и серой гнилью (GM), дифференциальную экспрессию проявили восемь, четыре, восемь, восемь и четыре гена CsLOX соответственно. Особо выделялся CsLOX22, который реагировал на тепловой, холодовой стрессы, а также на DM и GM. Полученные результаты создают основу для дальнейшего изучения функций генов CsLOX у огурца.
1. Введение
Липоксигеназа (линолеат:кислород оксидоредуктаза, EC:1.13.11.12) — это класс диоксигеназ, содержащих негемовое железо [1]. Она широко распространена у растений и животных, однако каталитический механизм различается у разных видов [2]. У животных липоксигеназа (LOX) катализирует образование нейропротекторов из арахидоновой кислоты (20 углеродов) и эйкозапентаеновой кислоты [3]. В царстве растений фермент LOX катализирует окислительное расщепление полиненасыщенных жирных кислот, включая линолевую, линоленовую и арахидоновую кислоты, что приводит к образованию гидропероксидов [4]. Эти гидропероксиды впоследствии преобразуются в кислородсодержащие производные через специализированные метаболические пути, которые включают растительные гормоны, такие как жасмоновая кислота (JA), эпоксиды [5], а также соединения с антибактериальными и противогрибковыми свойствами, например листовые альдегиды и диеноловые эфиры. Семейство LOX подразделяется на две основные группы: 9-LOX и 13-LOX, в зависимости от положения атома углерода, по которому происходит каталитическое окисление субстрата. Кроме того, 13-LOX дополнительно подразделяется на тип I и тип II [6].
Было показано, что LOX участвуют в биосинтезе летучих органических соединений [7], устойчивости к абиотическим стрессам [8,9,10], созревании плодов [11], биосинтезе JA [12] и защите от биотических воздействий [13,14]. Например, сайленсинг гена TomloxC у томата (Solanum lycopersicum L.) приводит к накоплению C6-короткоцепочечных летучих альдегидов, производных жирных кислот, в плодах и листьях [15]. У персика (Prunus persica L.) ген PpLOX4 тесно связан с синтезом аромата плодов [16]. LOX обычно играет положительную роль в ответах растений на различные стрессовые условия; например, сверхэкспрессия гена OsLOX1 повышает устойчивость риса (Oryza sativa L.) к механическим повреждениям и атакам насекомых [17]. CaLOX1 у перца стручкового (Capsicum annuum L.) играет ключевую роль в адаптации растения к осмотическому стрессу, обезвоживанию и засолению [18]. У дыни (Cucumis melo L.) CmlOX10 улучшает засухоустойчивость растения посредством жасмонат-опосредованного закрытия устьиц [19]. У кукурузы (Zea mays L.) гены ZmLOX10 и ZmLOX8 предоставляют субстраты для путей биосинтеза зеленых листовых летучих соединений (GLV) и JA соответственно, катализируя продукцию летучих веществ, выполняющих защитные функции против насекомых [20]. У хурмы (Diospyros kaki Thunb.) гетерологичная экспрессия DkLOX3 повышала устойчивость трансгенного Arabidopsis к Botrytis cinerea [21]. Кроме того, было показано, что липоксигеназы реагируют на холод [22], жару [23] и заражение Cladosporium fulvum [24].
Семейство LOX широко распространено среди растений. На сегодняшний день в отчетах идентифицировано 6 генов LOX у Arabidopsis (Arabidopsis thaliana) [25], 23 у груши (Pyrus bretschneideri Rehd) [26], 18 у дыни [27], 36 у сои (Glycine max (L.) Merr.) [28], 30 у люцерны серповидной (Medicago truncatula) [29], 10 у нута (Cicer arietinum L.) [30], 18 у винограда (Vitis vinifera L.) [31], 72 у арахиса (Arachis hypogaea L.) [32] и 14 у земляники чилийской (Fragaria chiloensis (L.) Mill.) [33]. В предыдущем исследовании было идентифицировано 23 гена CsLOX в первоначальной версии генома огурца (Cucumis sativus L.) 1.0 (v1.0) сорта 'Chinese Long 9930' ('9930') [34]. Однако геном был обновлен до версии v4.0 [35], и успешно создан пан-геном огурца [36]. Это предоставило хорошие ресурсы для идентификации генов CsLOX и изучения их функций у огурца [37].
Огурец — растение, происходящее с южных склонов Гималаев [38]. Благодаря своим уникальным вкусовым и ароматическим характеристикам он приобрел широкую популярность и стал важной товарной культурой. В процессе выращивания сорта огурца подвержены многочисленным биотическим и абиотическим стрессам, которые могут серьезно влиять на их урожайность [39,40,41]. Таким образом, идентификация генов устойчивости к стрессу, таких как LOX, у огурца имеет большое значение. С созданием пан-генома огурца [36] исследователи получили возможность более глубокого и всестороннего понимания генетического разнообразия, структуры генома и взаимосвязи этих факторов с ключевыми признаками, такими как устойчивость к болезням и стрессоустойчивость, что предоставляет ценную информацию для изучения функций генов и развития молекулярной селекции.
В этом исследовании мы идентифицировали в общей сложности 24 гена CsLOX в пан-геноме огурца и обнаружили, что все гены CsLOX проявляли вариации аминокислот у различных образцов. Мы провели дальнейший анализ локализации на хромосомах, филогении, структуры генов и межвидового синтенического соотношения этих генов CsLOX. Кроме того, на основе транскриптомных данных мы исследовали паттерны дифференциальной экспрессии генов CsLOX в различных тканях и их реакцию на различные стрессы. В заключение, некоторые гены-кандидаты, реагирующие на заражение Botrytis cinerea, были подтверждены с помощью кПЦР. Наше исследование направлено на предоставление всестороннего обзора характеристик семейства LOX огурца.
2. Материалы и методы
2.1. Общегеномная идентификация членов семейства генов LOX у огурца
Данные генома огурца были получены из Cucumber-DB (http://www.cucumberdb.com/, доступ 10 августа 2024 г.) и NCBI (https://www.ncbi.nlm.nih.gov/, доступ 22 марта 2024 г.), профиль HMM домена LOX (PF00305) был получен из базы данных InterPro (https://www.ebi.ac.uk/interpro/, доступ 22 марта 2024 г.). Кандидатные последовательности были отфильтрованы с использованием SMART (http://smart.embl-heidelberg.de/, доступ 26 марта 2024 г.) и NCBI CD-Search (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi, доступ 26 марта 2024 г.), исключая последовательности, не содержащие домен LOX.
2.2. Анализ длины белков и последовательностей
Извлеките последовательности белков CsLOX из различных образцов огурца и измерьте их длину с помощью ExPASy (https://web.expasy.org/compute_pi/, доступ 28 августа 2024 г.). Оцените вариации аминокислот с использованием программы DNAman V7.0.
2.3. Филогенетический анализ
Отберите последовательности белков членов семейства LOX огурца, Arabidopsis, риса и сои. Последовательности белков LOX были получены из Cucumber-DB (http://www.cucumberdb.com/, доступ 10 августа 2024 г.) и с веб-сайта Phytozome 13 (https://phytozome-next.jgi.doe.gov/, доступ 10 августа 2024 г.). Филогенетический анализ проводили с использованием MEGA11, проверку начальной загрузки (bootstrap) выполняли с использованием 1000 повторностей, и сгенерированные филогенетические деревья были графически представлены и оформлены с помощью Evolview (https://evolgenius.info//evolview-v2/#login, доступ 15 августа 2024 г.).
2.4. Анализ состава мотивов, структуры генов и предсказание цис-элементов
Используйте программное обеспечение MEME версии 5.5.5 (https://meme-suite.org/meme/tools/meme, доступ 15 августа 2024 г.) для изучения мотивов, присутствующих в последовательностях белков CsLOX. Максимальное количество мотивов было установлено на 10. Визуализируйте белковые мотивы и структуры генов с помощью TBtools [42]. Извлеките фрагменты промоторной области размером 1,5 т.п.н. с помощью TBtools [42] и проанализируйте цис-действующие элементы в этих фрагментах промоторов с помощью онлайн-инструмента анализа PlantCARE.
2.5. Анализ дупликации генов и синтении
Данные генома огурца, дыни, Arabidopsis, томата, риса и кукурузы были получены из Cucumber-DB (http://www.cucumberdb.com/, доступ 10 августа 2024 г.) и Phytozome 13 (https://phytozome-next.jgi.doe.gov/, доступ 10 августа 2024 г.). Впоследствии синтенические взаимосвязи были идентифицированы с использованием MCScanX [43] с настройками по умолчанию. Полученные результаты затем визуализировали с помощью TBtools [42].
2.6. Транскриптомный анализ генов CsLOX у огурца
Общедоступные транскриптомные данные (SRA071224 и GSE81234) были загружены из NCBI (https://www.ncbi.nlm.nih.gov/, доступ 21 апреля 2024 г.) и Cucumber-DB (http://www.cucumberdb.com/, доступ 21 апреля 2024 г.) для анализа паттернов экспрессии генов LOX в различных тканях [44], при тепловом, холодовом стрессе, стрессе, вызванном мучнистой росой (PM), ложной мучнистой росой (DM) [45] и серой гнилью (GM) огурца. Профили экспрессии затем визуализировали с помощью TBtools [42] для создания тепловых карт.
2.7. Обработка растений и анализ количественной ПЦР в реальном времени (кПЦР-РВ)
Штамм Botrytis cinerea культивировали на среде картофельно-декстрозного агара (PDA) до расширения колоний примерно до 9 см в ширину. Диски диаметром 8 мм извлекали из краев этих колоний с помощью пробоотборника и затем использовали для инокуляции семядолей огурца. Образцы тканей отбирали с интервалами 0, 12, 24, 36 и 48 часов после инокуляции, с тремя повторностями для каждой временной точки. Листья немедленно замораживали в жидком азоте и сохраняли при -80 °C. Тотальную РНК выделяли с использованием реагента TRIzol (Accurate Biotechnology, каталожный номер AG21102, Чанша, Китай), оценивали ее целостность и количество. Комплементарную ДНК (кДНК) синтезировали с помощью набора Evo M-MLV RT Mix Kit (Accurate Biotechnology, каталожный номер AG11728). Количественную ПЦР в реальном времени (кПЦР-РВ) проводили с использованием 2X SYBR Green Pro Taq HS Premix (Accurate Biotechnology, каталожный номер AG11718) на системе CFX Opus 96 (BIORAD, Геркулес, Калифорния, США). Актин огурца (CsaV4_6G003010) служил в качестве эндогенного контрольного гена. Относительные уровни транскриптов генов CsLOX определяли методом 2−∆∆Ct [46]; последовательности праймеров приведены в дополнительной таблице S4.
3. Результаты
3.1. Идентификация и характеристика генов CsLOX у различных образцов огурца
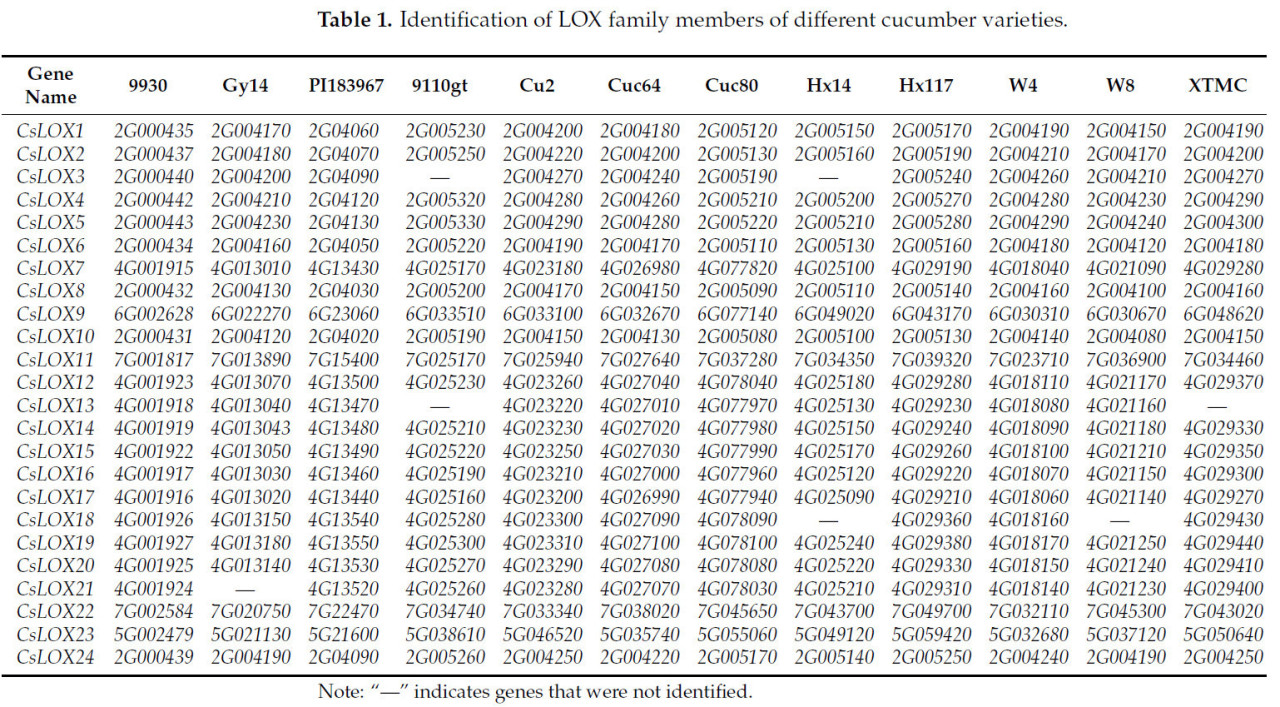
Публикация пан-генома огурца способствует лучшему пониманию генетического разнообразия и адаптивности огурца. Мы загрузили файлы профилей HMM и извлекли белки CsLOX из Cucumber-DB. В общей сложности было идентифицировано 24 гена CsLOX, которые были названы CsLOX1 – CsLOX24 на основе их идентификаторов генов и структур, включая недавно идентифицированный ген CsLOX24 (Таблица 1). Количество генов CsLOX варьировало у различных образцов огурца. В частности, 24 гена были обнаружены у '9930', 'PI183967', 'Cu2', 'Cuc80', 'Hx117' и 'W4'; 23 гена – у 'Gy14', 'W8', 'Cuc64' и 'XTMC'; и 22 гена – у 'Hx14' и '9110gt'. В пан-геноме огурца присутствует сорт 'Cuc37'. Однако из-за проблем с файлами аннотации мы не смогли точно идентифицировать специфичные гены LOX у этого сорта. Поэтому мы исключили этот сорт из данного исследования.
Таблица 1. Идентификация членов семейства LOX различных сортов огурца.
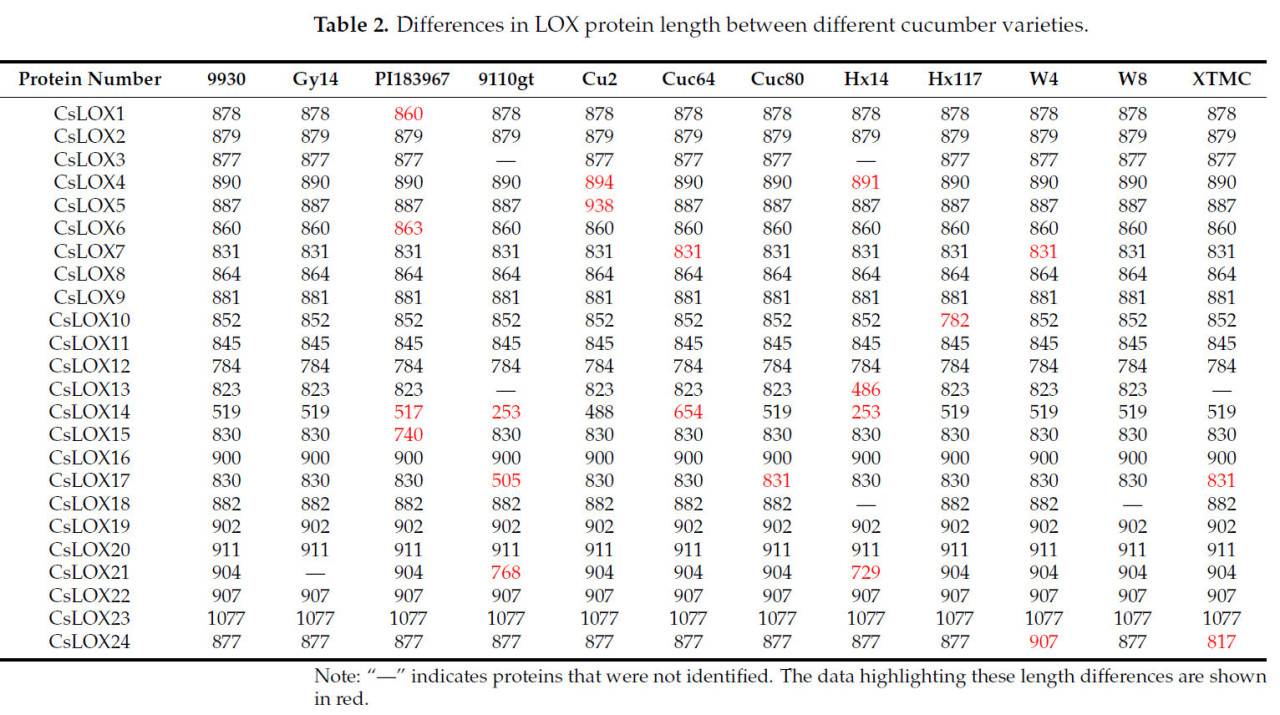
Чтобы дальнейшем понять различия в длине белков CsLOX среди различных образцов огурца, мы провели статистический анализ длин белков CsLOX у идентифицированных образцов (Таблица 2). Мы обнаружили, что 13 белков CsLOX имели одинаковую длину у 12 различных образцов огурца. CsLOX1, CsLOX5, CsLOX6, CsLOX10, CsLOX13 и CsLOX15 имеют различную длину только у одного образца, тогда как CsLOX4, CsLOX21 и CsLOX24 имеют различия в длине белка у двух образцов, а CsLOX17 и CsLOX14 имеют различия в длине белка у трех и четырех образцов соответственно. Изменения аминокислот также могут влиять на функцию белка. Аналогично, мы проанализировали вариации аминокислот CsLOX у различных образцов огурца (Таблица S1). Все белки CsLOX показали вариации аминокислот, и большинство вариаций не влияли на длину белка, за исключением случаев, когда происходили вставки или делеции аминокислот, что приводило к изменениям длины белка. Интересно, что CsLOX10 у 'W8' подвергся делеции шести аминокислот и вставке шести аминокислот (Таблица S1), что не привело к изменению длины белка.
Таблица 2. Различия в длине белка LOX между различными сортами огурца.
Среди 12 изученных образцов только 6 ('9930', 'PI183967', 'Cu2', 'Cuc80', 'Hx117' и 'W4') содержали 24 гена CsLOX (Таблица 1). Следовательно, последующие анализы в основном были сосредоточены на генах, идентифицированных в геноме '9930', так как он содержал все 24 гена CsLOX и был первым секвенированным геномом и первым почти полным геномом огурца.
3.2. Филогенетический анализ членов LOX
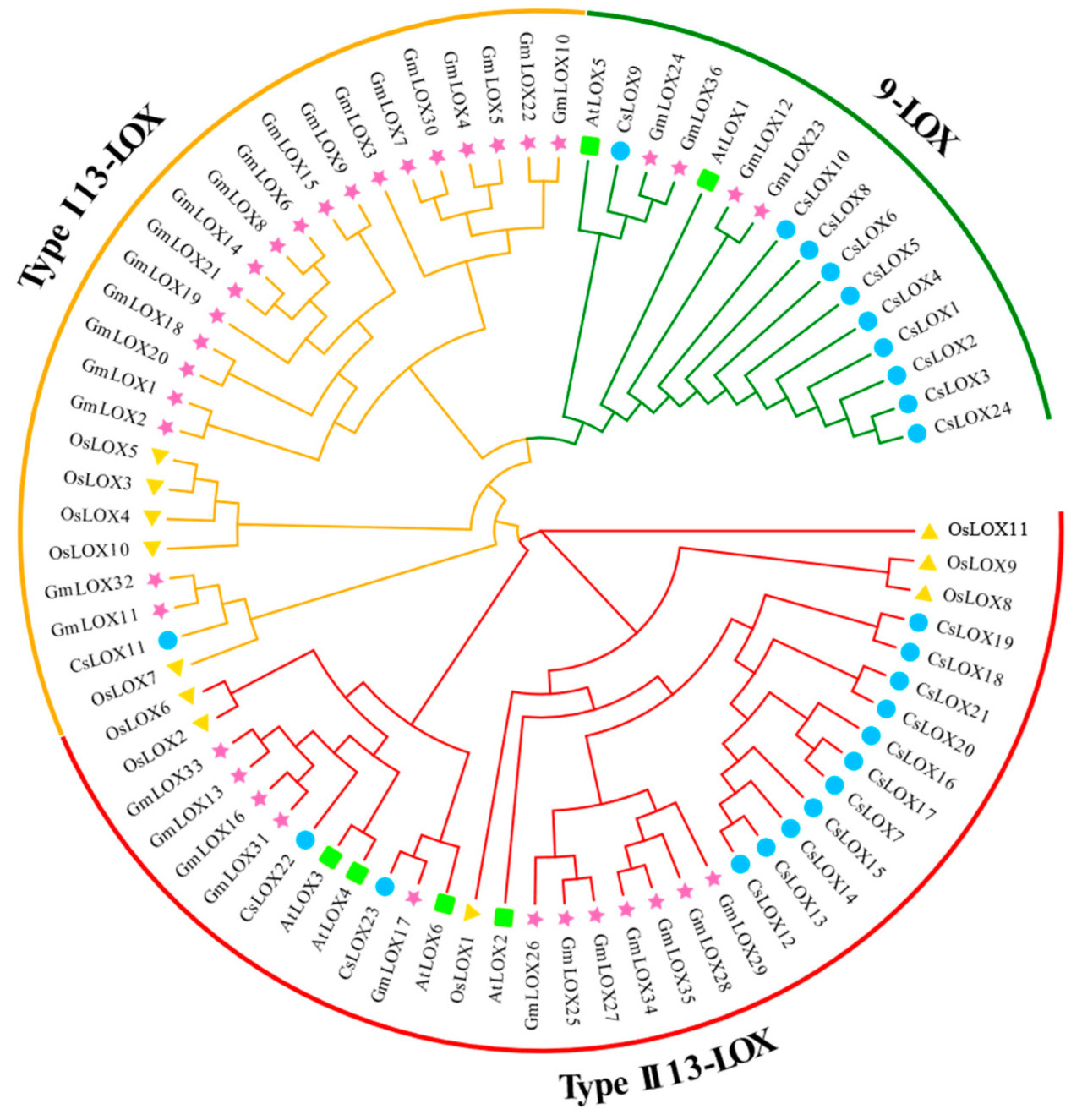
Мы проанализировали филогенетические взаимосвязи между членами LOX у растений с использованием программного обеспечения MEGA (v.11) методом максимального правдоподобия на основе аминокислотных последовательностей генов LOX Arabidopsis, риса, сои и 24 идентифицированных генов CsLOX (Рисунок 1). Белки CsLOX были классифицированы на два подсемейства: 9-LOX и 13-LOX, на основе филогенетического анализа. Кроме того, подсемейство 13-LOX можно разделить на две подгруппы: тип I и тип II. Подсемейство 9-LOX включало 10 белков CsLOX (CsLOX1-6, CsLOX8-10 и CsLOX24), и 13 белков CsLOX (CsLOX7 и CsLOX12-23) принадлежали к подсемейству 13-LOX типа II. Белок CsLOX11 был единственным, который был классифицирован в подсемейство 13-LOX типа I.
Рисунок 1. Филогенетическое дерево семейства генов LOX огурца. Разноцветные символы представляют представителей различных видов: красный — соя, синий — огурец, желтый — рис, зеленый — Arabidopsis. Филогенетический анализ на основе ML был выполнен с использованием MEGA 11, включая 1000 начальных загрузочных (bootstrap) выборок.
3.3. Анализ консервативных мотивов, структуры генов и цис-элементов генов CsLOX
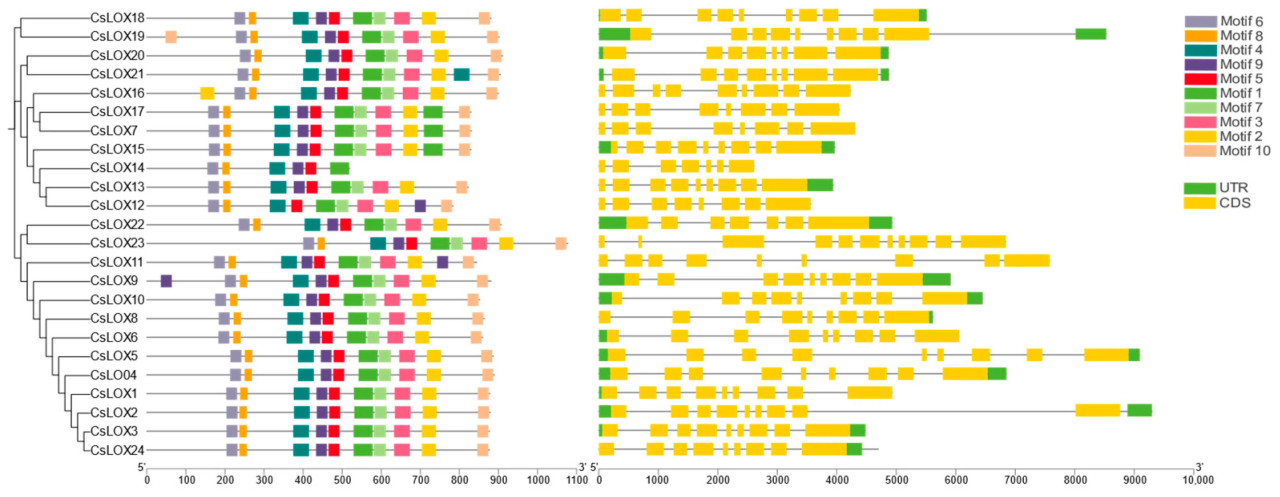
Структура экзонов служит основой для изучения эволюционных взаимосвязей генов CsLOX (Рисунок 2). Количество экзонов в гене LOX огурца варьирует от 7 до 11, что указывает на то, что члены семейства генов CsLOX в процессе эволюции претерпевали как потери, так и приобретения экзонов. Кроме того, количество и размер экзонов у большинства CsLOX довольно схожи. Чтобы улучшить наше понимание консервативности и разнообразия CsLOX, мы идентифицировали 10 консервативных мотивов в белках CsLOX, обозначенных как мотив1 – мотив10. Стоит отметить, что CsLOX14 содержит только шесть консервативных мотивов, что может быть связано с его структурой гена.
Рисунок 2. Эволюция, структура генов и анализ мотивов семейства генов LOX огурца. Филогенетическое дерево максимального правдоподобия с 1000 повторностями было построено с использованием MEGA 11. Десять консервативных мотивов представлены прямоугольниками разных цветов. Желтые прямоугольники представляют экзоны, серые линии — интроны, зеленые прямоугольники — UTR.
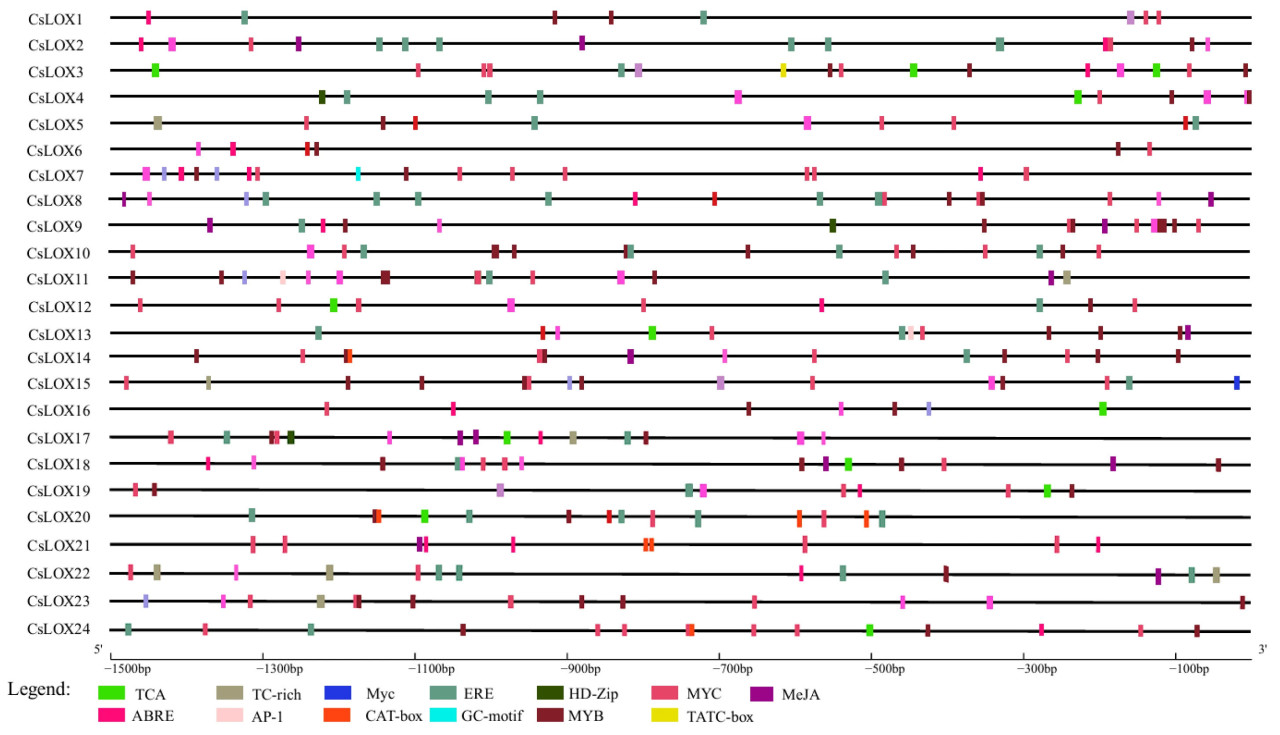
Цис-действующие элементы в промоторах играют ключевую роль в регуляции экспрессии генов. Эти элементы расположены в некодирующих областях ДНК, обычно выше по течению от сайтов начала транскрипции [47]. Поэтому мы выбрали область размером 1,5 т.п.н., фланкирующую 5'-конец выше стартового кодона каждого гена CsLOX. После удаления избыточных результатов в промоторных областях генов CsLOX было обнаружено множество регуляторных элементов, связанных с сигнальными путями растений, отвечающими на гормоны и стресс (Рисунок 3). Элементы, связанные с гормонами, включали TCA (реакция на салициловую кислоту), ERE (элемент реакции на этилен), MeJA (реакция на метилжасмонат), ABRE (элемент реакции на абсцизовую кислоту) и TATC-бокс (реакция на гиббереллин). А элементы, связанные со стрессом, включали TC-rich (реакция на стресс), GC-мотив (индуцибельность в условиях аноксии) и сайт связывания MYB (сайт связывания фактора транскрипции MYB). Некоторые другие цис-действующие элементы также были обнаружены в промоторе, включая HD-Zip (сайт связывания белка), AP-1 (активирующий белок-1), CAT-box (экспрессия в меристеме) и MYC. Все гены CsLOX содержат цис-действующие элементы, связанные с гормонами, а большинство генов CsLOX содержат цис-действующие элементы, связанные со стрессом. Это может показывать потенциальные роли CsLOX в гормональных путях и стрессовых реакциях.
Рисунок 3. Анализ цис-элементов в промоторах генов CsLOX.
3.4. Анализ синтении генов CsLOX
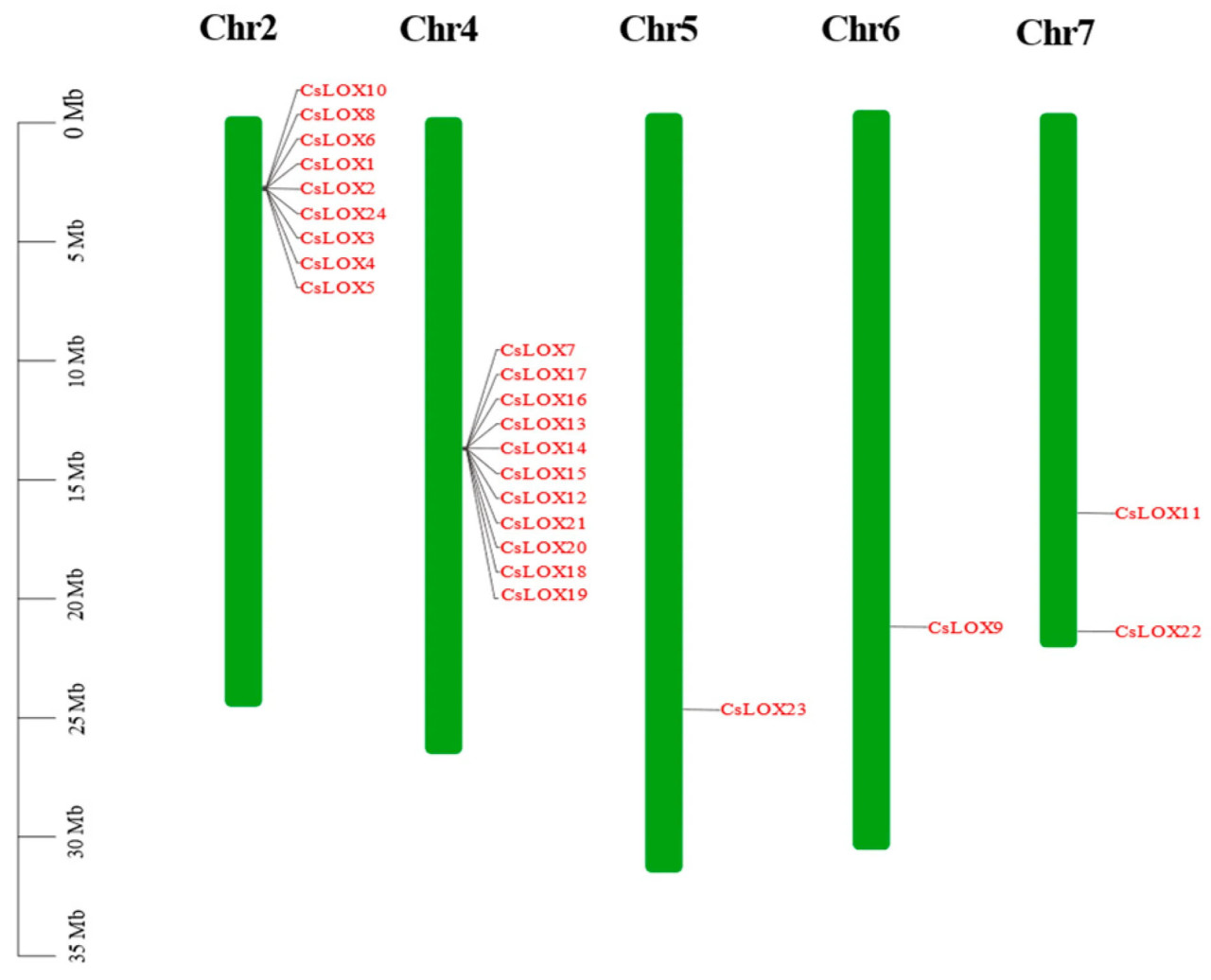
Распределение генов по хромосомам указывает на то, что члены семейства CsLOX неравномерно распределены по пяти из семи хромосом огурца (хромосомы 2, 4, 5, 6 и 7) (Рисунок 4). В частности, хромосома 2 содержит девять генов CsLOX, хромосома 4 содержит одиннадцать генов CsLOX, хромосома 5 содержит один ген CsLOX, хромосома 6 содержит один ген CsLOX, а хромосома 7 содержит два гена CsLOX. Учитывая, что гены на хромосомах часто распределены кластерами, мы предполагаем, что многие гены CsLOX произошли в результате тандемных или сегментных дупликаций.
Рисунок 4. Карта расположения генов CsLOX на хромосомах. Приблизительные положения 24 генов CsLOX указаны справа от диаграмм хромосом.
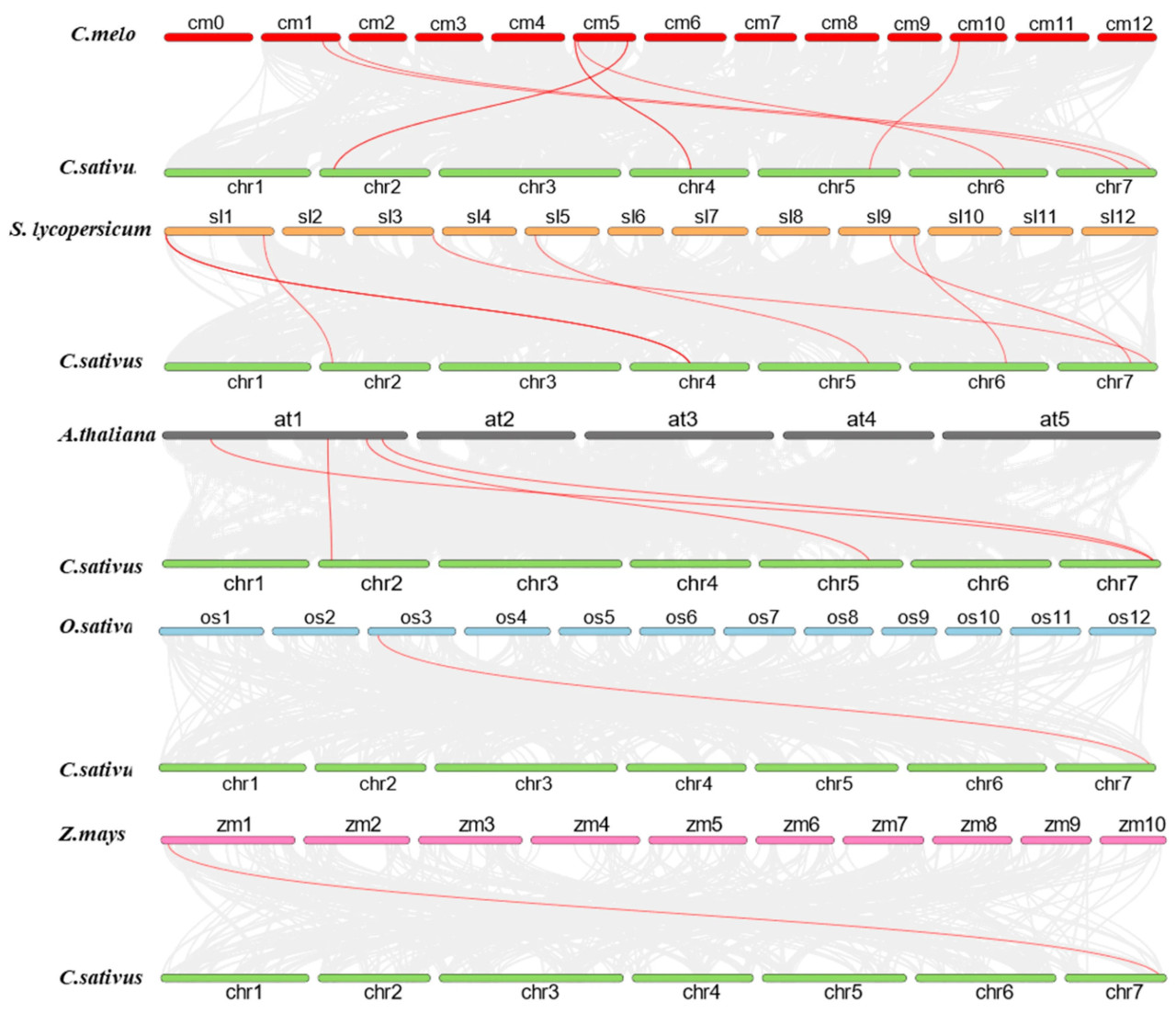
Межвидовой анализ синтении направлен на выявление консервативности и эволюционных взаимосвязей между геномами различных видов. Мы исследовали синтенические взаимосвязи между генами LOX огурца и их гомологичными генами у пяти других видов, включая три двудольных (томат, дыня и Arabidopsis) и два однодольных (рис и кукуруза) (Рисунок 5). Результаты показали, что огурец имел шесть гомологичных пар с дыней, шесть гомологичных пар с томатом, четыре гомологичные пары с Arabidopsis, одну гомологичную пару с рисом и одну гомологичную пару с кукурузой. Интересно, что CsLOX22 имел соответствующие гомологичные гены у пяти других видов, что указывает на высокую консервативность этого гена у различных видов.
Рисунок 5. Анализ коллинеарности генов LOX между C. sativus, C. melo, S. lycopersicum, A. thaliana, O. sativa и Z. mays: Коллинеарные блоки отмечены серыми линиями, а коллинеарные пары генов с генами LOX выделены красными линиями.
3.5. Паттерны экспрессии CsLOX в различных тканях
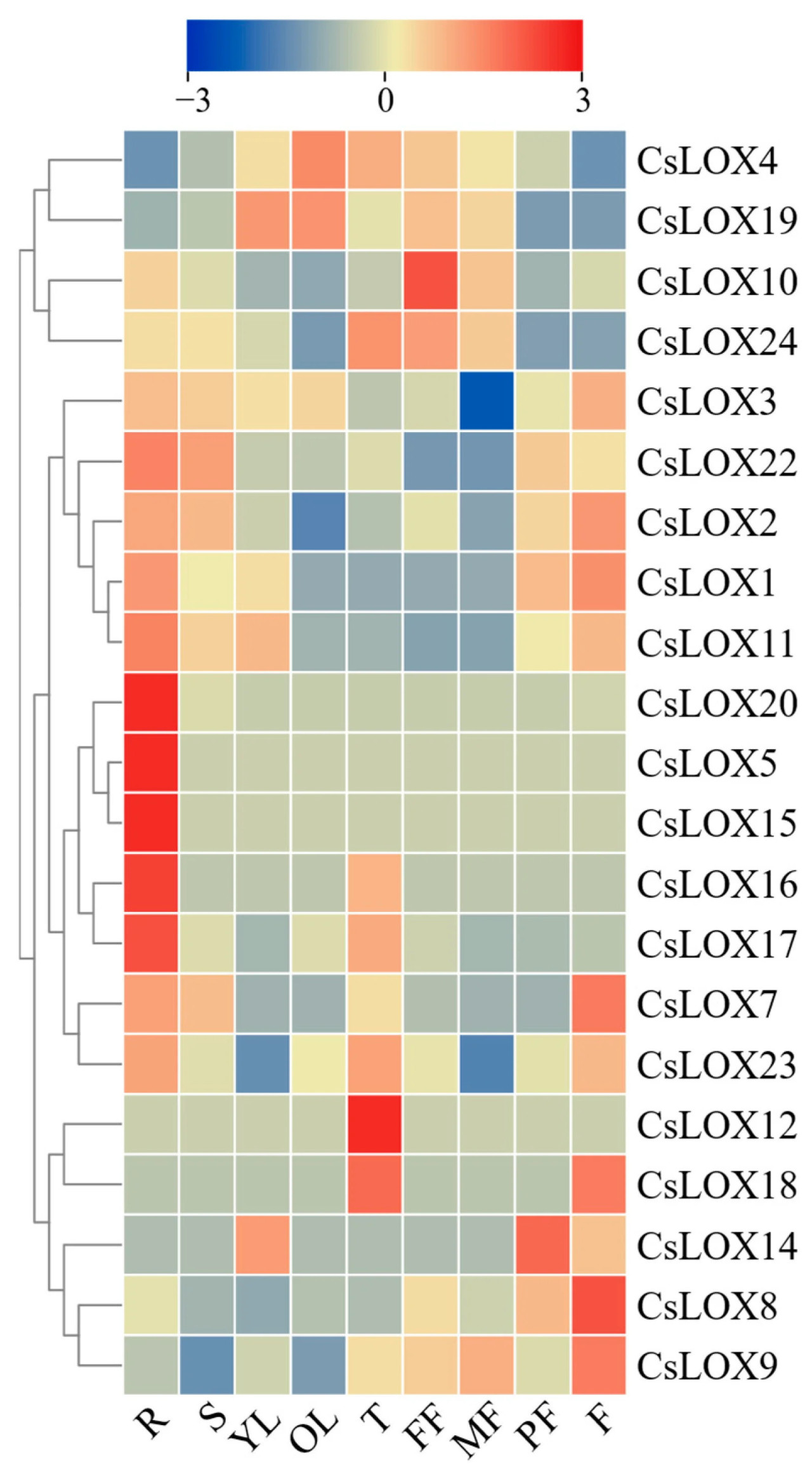
Тканеспецифичная экспрессия может быть связана с функцией гена. Мы загрузили транскриптомные данные из Cucumber-DB для различных тканей огурца, проанализировали уровни экспрессии CsLOX в этих тканях и создали тепловую карту для визуализации (Рисунок 6). Результаты показали, что CsLOX1 и CsLOX2 экспрессировались в корнях, стеблях, листьях и плодах огурца на различных стадиях развития. CsLOX3 экспрессировался во всех тканях, кроме усиков. CsLOX9, CsLOX10, CsLOX19, CsLOX22, CsLOX23 и CsLOX24 были повсеместно экспрессированы во всех тканях. CsLOX12 был специфически экспрессирован в усиках, тогда как CsLOX5, CsLOX15, CsLOX16 и CsLOX20 преимущественно экспрессировались в корнях. Эти результаты свидетельствуют о том, что гены CsLOX могут играть разнообразные роли в росте и развитии огурца.
Рисунок 6. Профили экспрессии генов LOX в различных тканях огурца. Приблизительные положения 24 генов CsLOX отображены справа от хромосом. Цвет на шкале представляет значение Log2(FPKM), синий цвет — низкий уровень, красный — высокий уровень. R: Корень; S: Стебель; YL: Молодой лист; OL: Старый лист; T: Усик; FF: Женский цветок; MF: Мужской цветок; PF: Плод с кожицей; F: Плод. В таблице S2 перечислены значения FPKM CsLOX в различных тканях огурца.
3.6. Паттерны экспрессии генов CsLOX в ответ на абиотические и биотические стрессы
Учитывая потенциальные роли генов LOX и их метаболитов в реагировании как на биотические, так и на абиотические стрессы у различных видов растений, мы использовали общедоступные транскриптомные данные для изучения уровней транскрипции генов CsLOX в условиях теплового, холодового стресса, стресса, вызванного мучнистой росой (PM), ложной мучнистой росой (DM) и серой гнилью (GM). Цель этого исследования — уточнить функцию CsLOX в этих стрессовых условиях, используя общедоступные транскриптомные данные.
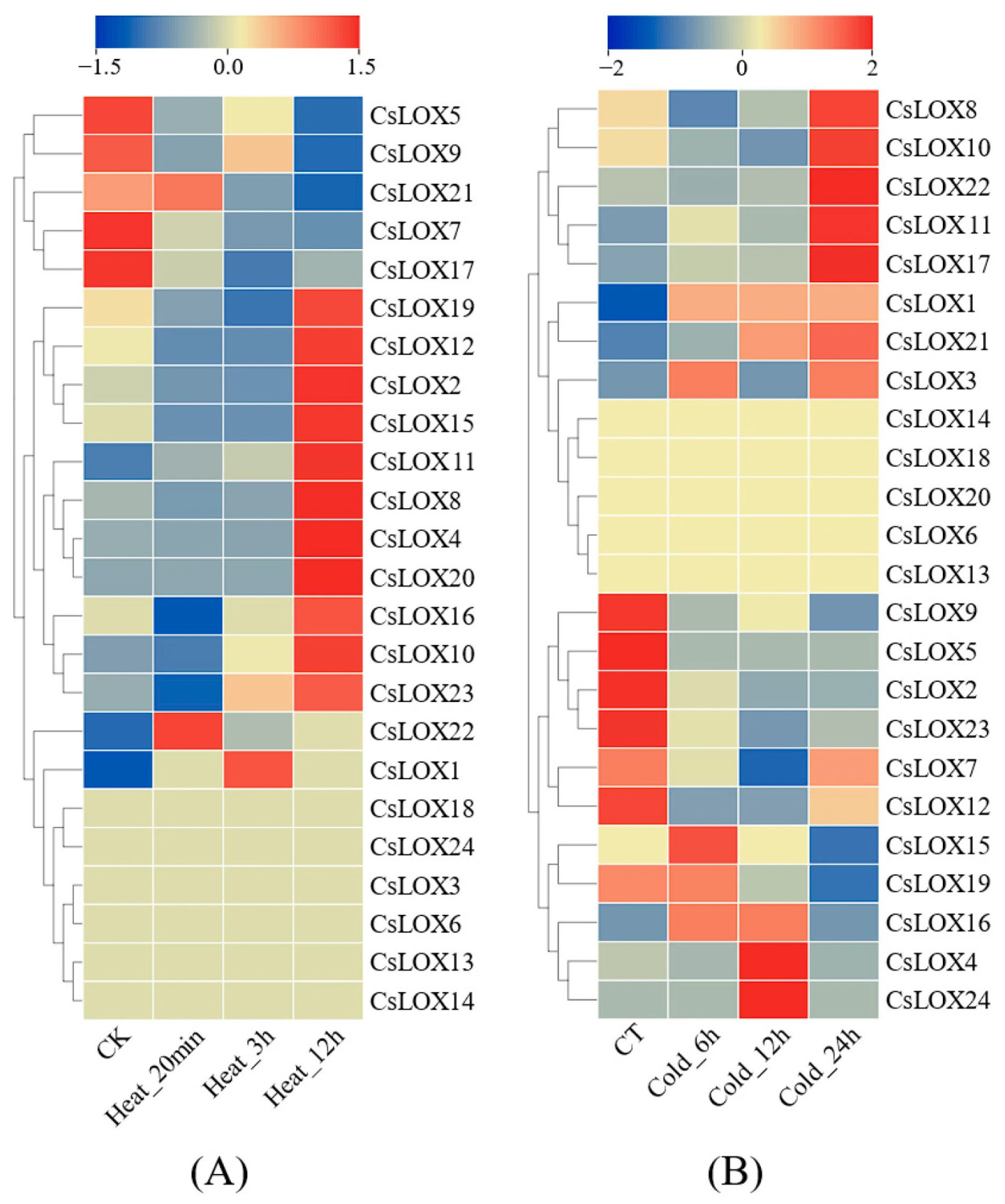
Первоначально мы исследовали транскрипционные профили CsLOX в ответ на тепловой стресс [35] (Рисунок 7). Через 12 часов воздействия высокой температуры уровни экспрессии CsLOX10, CsLOX11 и CsLOX22 у огурца были значительно повышены, тогда как CsLOX9 и CsLOX21 были значительно снижены. Примечательно, что CsLOX2, CsLOX4 и CsLOX19 сначала были понижены, а затем повышены. Мы также исследовали реакцию CsLOX на холодовой стресс [35] (Рисунок 7). После 24 часов холодовой обработки CsLOX4, CsLOX10, CsLOX17 и CsLOX22 были значительно повышены. Эти результаты свидетельствуют о том, что эти гены могут играть важные роли в реакциях на тепловой и холодовой стресс у огурцов.
Рисунок 7. Экспрессия CsLOX при тепловой и холодовой обработке. (А) Тепловая карта показывает паттерн экспрессии CsLOX после 12 ч теплового стресса. (В) Тепловая карта показывает паттерн экспрессии CsLOX после 24 ч холодового стресса. Цвет на шкале представляет значение Log2(FPKM), синий цвет — низкий уровень, красный — высокий уровень. В таблице S3 перечислены значения FPKM CsLOX при тепловом и холодовом стрессе.
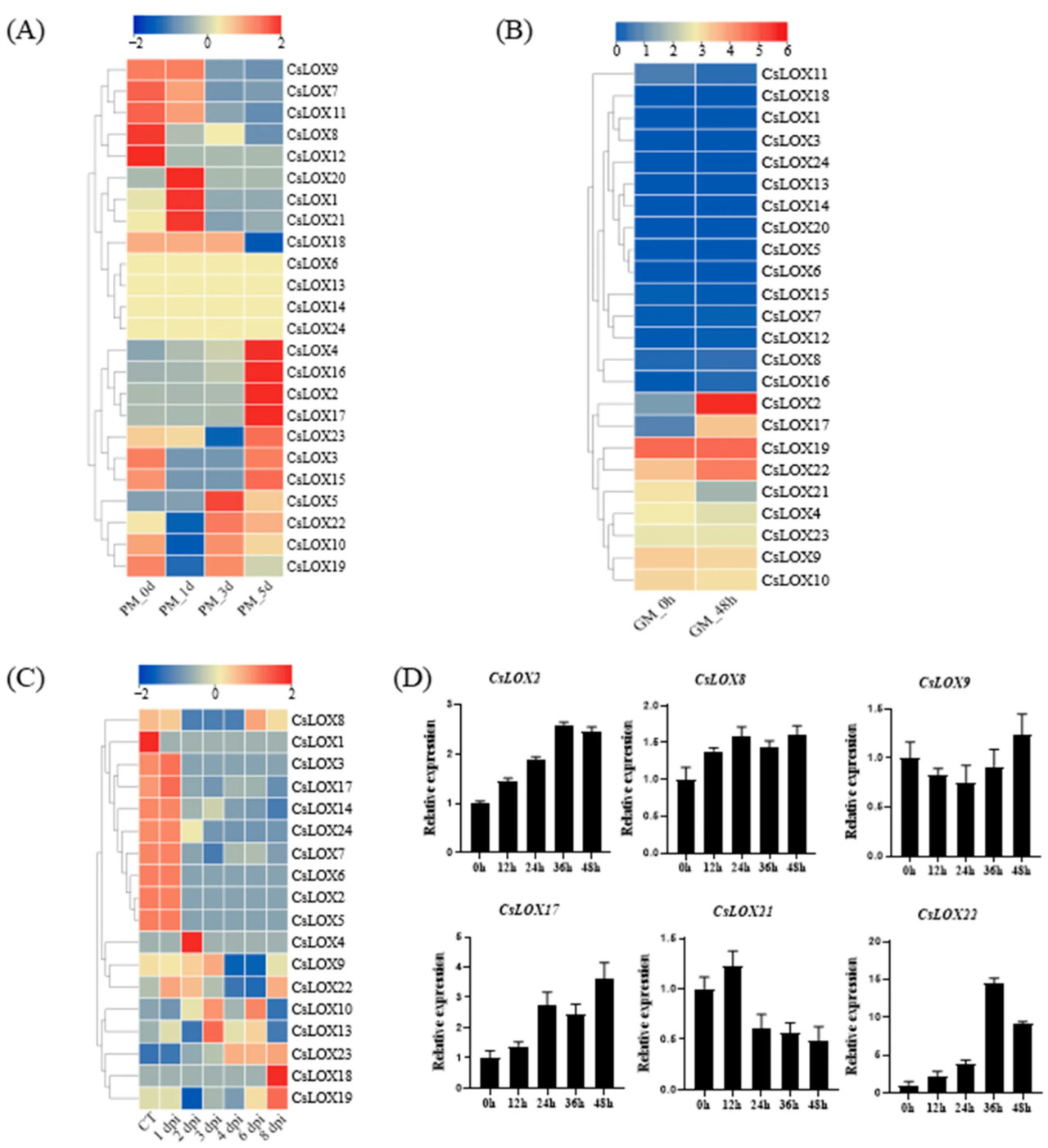
Чтобы раскрыть возможные роли CsLOX в устойчивости к биотическим стрессам, мы оценили уровни экспрессии CsLOX на основе доступных наборов данных RNA-Seq от проростков огурца, зараженных PM в течение 5 дней [35], DM в течение 8 дней [45] и GM в течение 48 часов [35] (Рисунок 8). Через 5 дней после инокуляции PM экспрессия CsLOX2, CsLOX4 и CsLOX17 была повышена, тогда как экспрессия CsLOX7, CsLOX9, CsLOX11, CsLOX19 и CsLOX21 была понижена. В транскриптомном анализе проростков огурца, зараженных DM, восемь CsLOX проявили значительные изменения экспрессии. CsLOX10 и CsLOX23 были повышены, тогда как CsLOX7, CsLOX14, CsLOX17 и CsLOX24 были понижены. Примечательно, что CsLOX19 был повышен на 8-й день, но понижен на 2-й, 3-й и 4-й дни. CsLOX22 был повышен на 1-й, 2-й и 8-й дни, но понижен на 4-й и 6-й дни. В транскриптомных данных огурцов, зараженных GM, только четыре CsLOX проявили значительные изменения экспрессии. В частности, экспрессия CsLOX2, CsLOX17 и CsLOX22 была повышена, тогда как экспрессия CsLOX21 была значительно понижена. Эти результаты свидетельствуют, что CsLOX реагируют на различные биотические стрессы и могут играть как положительную, так и отрицательную роль в устойчивости огурца к инфекциям патогенов. Необходимы дальнейшие исследования для полного понимания специфических функций каждого CsLOX.
Рисунок 8. Экспрессия CsLOX при обработке мучнистой росой (PM), серой гнилью (GM) и ложной мучнистой росой (DM). (А) Тепловая карта показывает паттерн экспрессии CsLOX через пять дней при стрессе PM. (В) Тепловая карта показывает паттерн экспрессии CsLOX через 48 ч при стрессе GM. (С) Тепловая карта показывает паттерн экспрессии CsLOX через восемь дней при стрессе DM. Цвет на шкале представляет значение Log2(FPKM), синий цвет — низкий уровень, красный — высокий уровень. В таблице S3 перечислены значения FPKM CsLOX при стрессе PM, DM и GM. (D) кПЦР-РВ анализ экспрессии CsLOX в проростках огурца, инокулированных серой гнилью (GM) через 0, 12, 24, 36 и 48 ч, с геном актина огурца в качестве референсного гена. Планки погрешностей иллюстрируют стандартную ошибку среднего, n = 3 указывает количество повторностей.
Чтобы объяснить потенциальные функции CsLOX в ответ на Botrytis cinerea, мы выполнили кПЦР-РВ для оценки паттернов экспрессии CsLOX в семядолях проростков огурца с интервалами 0, 12, 24, 36 и 48 часов после инокуляции Botrytis cinerea (Рисунок 8). В целом, данные кПЦР-РВ в значительной степени отражали результаты RNA-seq, тем самым подтверждая точность наборов данных. Среди шести генов-кандидатов уровни экспрессии CsLOX2 увеличились через 12 часов после инокуляции и сохранялись на относительно высоком уровне впоследствии. Уровень экспрессии CsLOX8 немного увеличился после инокуляции, но значительной разницы не было. Уровень экспрессии CsLOX9 остался неизменным после инокуляции. Уровень экспрессии CsLOX17 значительно увеличился через 12 часов после инокуляции. Экспрессия CsLOX21 увеличилась через 12 часов после инокуляции, но быстро снизилась впоследствии. Уровень экспрессии CsLOX22 сначала увеличился, достигнув пика через 36 часов после инокуляции, а затем снизился. Таким образом, данные кПЦР-РВ согласуются с результатами из набора данных RNA-seq.
4. Обсуждение
Огурец — широко возделываемая овощная культура, имеющая значительную экономическую ценность [48]. В процессе выращивания огурец часто страдает от снижения урожайности из-за различных стрессов [49]. Эти стрессы могут серьезно препятствовать росту огурца и могут оказывать значительное влияние на производство. Липоксигеназа (LOXs; линолеат:кислород, EC 1.13.11.12) — это железосодержащий [1], негемовый оксидоредуктазный фермент, который играет важную роль в росте растений, развитии, стрессоустойчивости, созревании и старении. В последние годы он был экстенсивно изучен у многих видов [25,26,27,28,29,30,31,32,33].
Из-за таких факторов, как географическое положение и окружающая среда, существует значительная генетическая изменчивость среди особей внутри одного вида. Референтный геном, полученный от одной особи, неадекватен для представления генетического разнообразия всей популяции вида. Пан-геномика может эффективно решить проблему потери информации и аналитической предвзятости, вызванных зависимостью от одного референтного генома [50,51]. Поэтому мы провели углубленный анализ и идентификацию семейства липоксигеназ (LOX) у 12 различных образцов огурца. Предыдущие исследования идентифицировали 23 члена семейства LOX [34]; однако наше исследование обнаружило нового члена, CsLOX24 (Таблица 1). Кроме того, мы наблюдали различное количество CsLOX у различных образцов огурца (Таблица 2). По сравнению с '9930', другие образцы демонстрировали либо то же количество CsLOX, либо отсутствие одного-двух генов. Длина белков также варьировала у различных образцов; например, белок CsLOX13 у 'Hx14' заметно короткий (Таблица 2). Кроме того, все идентифицированные белки LOX показывают вариации аминокислот, включая вставки, делеции и одиночные аминокислотные замены (Таблица S1). Эти вариации могут быть результатом естественных мутаций или могут происходить из-за погрешностей в предсказании генов и секвенировании. Для установления точной природы этих вариаций необходим анализ специфических генов. Мы также обнаружили, что большинство CsLOX, обладая высокой схожестью, были расположены вместе в двух кластерах на хромосомах 2 и 4 (Рисунок 4); этот феномен существует во всех образцах огурца. Соответственно, это указывает на то, что у огурца произошла дупликация этих двух генов по сравнению с Arabidopsis.
Более ранние исследования показали, что применение летучего фитогормона метилжасмоната (MeJA) вызывало дозозависимую эмиссию метанола и других летучих соединений из пути липоксигеназ [52]. У персиков взаимодействие между связанными с этиленом генами PpERF5 и PpERF7 может повышать экспрессию PpLOX4, тем самым усиливая аромат плодов персика [16]. В нашем исследовании мы идентифицировали множество цис-действующих элементов в промоторах CsLOX (Рисунок 3). Эти связанные с гормонами цис-действующие элементы ассоциированы с такими гормонами, как абсцизовая кислота, этилен, салициловая кислота и JA. Основываясь на предыдущих исследованиях, мы можем предположить, что уровни экспрессии CsLOX могут регулироваться этими гормонами.
У растений липоксигеназа связана с многочисленными процессами развития и функционирует в различных тканях [53,54]. ZmLOX3 у кукурузы регулирует развитие корней и устойчивость к галловым нематодам [55]. Благодаря анализу паттернов экспрессии CsLOX в различных тканях огурца мы обнаружили, что некоторые CsLOX повсеместно экспрессируются во всех тканях огурца, тогда как другие специфически экспрессируются в определенных органах (Рисунок 6). Например, CsLOX12 специфически экспрессируется в усиках, и его промотор содержит цис-регуляторные элементы, связанные с экспрессией в меристеме, что свидетельствует о его возможном участии в развитии усиков. CsLOX16 экспрессируется с высокой специфичностью в корнях, потенциально участвуя в развитии корней или играя роль в устойчивости к галловым нематодам. CsLOX9, CsLOX10, CsLOX19, CsLOX22, CsLOX23 и CsLOX24 повсеместно экспрессируются во всех тканях, что указывает на их роль в росте и развитии огурцов.
Многочисленные исследования установили, что LOX играют ключевую роль в защитных механизмах растений как против биотических, так и абиотических стрессоров. AfLOX4 из Amorpha fruticosa L. [56] и CmLOX10 из дыни [19] могут повышать засухоустойчивость. ZmLOX10 и ZmLOX8 у кукурузы могут продуцировать летучие вещества, выполняющие защитные функции против насекомых [20]. MdLOX3 из яблони положительно регулирует солеустойчивость каллуса яблони и Arabidopsis [8]. Кроме того, CmLOX9 у дыни может противостоять Xanthomonas campestris [57]. В нашем исследовании мы проанализировали паттерны реагирования CsLOX на абиотические стрессы (жара и холод) (Рисунок 7) и биотические стрессы (PM, DM и GM) (Рисунок 8). Многие CsLOX показали значительные изменения экспрессии при различных стрессах. При тепловом и холодовом стрессе CsLOX4, CsLOX10 и CsLOX22 реагировали все (Рисунок 7). Анализ их промоторных последовательностей выявил наличие MYB, ABRE и ERE цис-действующих элементов, что может объяснить, как они реагируют на эти стрессы [58,59,60]. Между тем, CsLOX17 показал значительные изменения экспрессии после инокуляции PM, DM и GM (Рисунок 8). Интересно, что CsLOX22 реагировал на тепловой, холодовой стрессы, а также на DM и GM (Рисунок 7 и Рисунок 8). Предыдущие исследования показали, что жасмоновая кислота играет ключевую роль в старении листьев и устойчивости растений к стрессу. Мы идентифицировали множество цис-действующих элементов MeJA и MYC в промоторах CsLOX17 и CsLOX22, что свидетельствует об их потенциальном участии в пути жасмоновой кислоты в ответ на стресс. Интересно, что CsLOX22 гомологичен CpLOX3a у кабачка (Cucurbita pepo) [61], который, как было показано, участвует в биосинтезе жасмоновой кислоты, указывая на то, что CsLOX22 может иметь сходную функцию. Мы предполагаем, что CsLOX могут играть роль в защите огурца от стрессов и что некоторые CsLOX могут обладать широким спектром стрессоустойчивости. Эти результаты будут способствовать идентификации ценных генов-кандидатов и дальнейшему исследованию стрессоустойчивости огурца. В будущем необходимы исследования для раскрытия молекулярных путей, посредством которых CsLOX способствуют устойчивости к стрессу у растений огурца.
5. Выводы
В этом исследовании мы идентифицировали 24 гена CsLOX в пан-геноме огурца, включая недавно обнаруженный ген CsLOX24. Большинство белков CsLOX проявляли аминокислотные вариации среди 12 образцов. Кроме того, CsLOX показали тканеспецифичную экспрессию, причем все CsLOX экспрессировались по крайней мере в одной ткани. Транскриптомные данные показали, что CsLOX могут реагировать на тепловой, холодовой стрессы, PM, DM и GM, что свидетельствует о потенциальных ролях CsLOX в стрессоустойчивости огурца. Это исследование провело систематический анализ семейства липоксигеназ огурца, что не только предоставляет основу для раскрытия функциональных ролей CsLOX, но также помогает идентифицировать подходящие гены-кандидаты для дальнейшей характеристики развития растений и стрессовых реакций.
Дополнительные материалы
Следующая вспомогательная информация может быть загружена по адресу: https://www.mdpi.com/article/10.3390/agriculture15030245/s1. Таблица S1. Вариация аминокислот у различных образцов огурца. Таблица S2. Данные RNA-seq (значения FPKM) генов CsLOX в различных тканях. Таблица S3. Данные RNA-seq (значения FPKM) CsLOX при абиотических и биотических стрессах. Таблица S4. Последовательности праймеров, использованные для кПЦР-РВ.
Ссылки
1. Brash, A.R. Lipoxygenases: Occurrence, functions, catalysis, and acquisition of substrate. J. Biol. Chem. 1999, 274, 23679–23682. [Google Scholar] [CrossRef] [PubMed]
2. Mack, A.J.; Peterman, T.K.; Siedow, J.N. Lipoxygenase isozymes in higher plants: Biochemical properties and physiological role. Isozymes 1987, 13, 127–154. [Google Scholar] [PubMed]
3. Bezáková, L.; Holková, I.; Balazová, A.; Obložinský, M.; Mikuš, P. Animal lipoxygenases—Structure, positional specificity of lipoxygenase isoenzymes and their importance. Chem. Listy 2017, 111, 567–574. [Google Scholar]
4. Siedow, J.N. Plant Lipoxygenase: Structure and Function. Annu. Rev. Plant Physiol. Plant Mol. Biol. 1991, 42, 145–188. [Google Scholar] [CrossRef]
5. Meng, Y.; Liang, Y.; Liao, B.; He, W.; Liu, Q.; Shen, X.; Xu, J.; Chen, S. Genome-Wide Identification, Characterization and Expression Analysis of Lipoxygenase Gene Family in Artemisia annua L. Plants 2022, 11, 655. [Google Scholar] [CrossRef]
6. Feussner, I.; Wasternack, C. The lipoxygenase pathway. Annu. Rev. Plant Biol. 2002, 53, 275–297. [Google Scholar] [CrossRef]
7. Sun, Y.; Li, X.; Ma, Z.; Chen, S. Quantitative trait locus mapping of fruit aroma compounds in cucumber (Cucumber sativus L.) based on a recombinant inbred line population. Hortic. Res. 2022, 9, uhac151. [Google Scholar] [CrossRef] [PubMed]
8. Chen, X.; Wang, D.; Zhang, C.; Wang, X.; Yang, K.; Wang, Y.; Wang, X.; You, C. The apple lipoxygenase MdLOX3 regulates salt tolerance and ABA sensitivity. Horticulturae 2022, 8, 651. [Google Scholar] [CrossRef]
9. Vellosillo, T. Oxylipins Produced by the 9-Lipoxygenase Pathway in Arabidopsis Regulate Lateral Root Development and Defense Responses through a Specific Signaling Cascade. Plant Cell 2007, 19, 831–846. [Google Scholar] [CrossRef]
10. Wiebke, G. Lipoxygenase6-Dependent Oxylipin Synthesis in Roots Is Required for Abiotic and Biotic Stress Resistance of Arabidopsis. Plant Physiol. 2013, 161, 2159–2170. [Google Scholar]
11. Zhou, S.; Li, D.; Cheng, Y.; Guan, J. Characterization of expression and enzyme activity of lipoxygenases during fruit softening and superficial scald development in ‘Wujiuxiang’ pear. J. Appl. Bot. Food Qual. 2016, 89, 307–314. [Google Scholar]
12. Shi, Y.; Fu, F. Relationship between LOX activity and JA accumulations in cucumber leaves induced by pathogen. Acta Phytophylacica Sin. 2008, 35, 486–490. [Google Scholar]
13. Battilani, P.; Lanubile, A.; Scala, V.; Reverberi, M.; Gregori, R.; Falavigna, C.; Dall’asta, C.; Park, Y.S.; Bennett, J.; Borrego, E.J.; et al. Oxylipins from both pathogen and host antagonize jasmonic acid-mediated defence via the 9-lipoxygenase pathway in Fusarium verticillioides infection of maize. Mol. Plant Pathol. 2018, 19, 2162–2176. [Google Scholar] [CrossRef] [PubMed]
14. Dong, W.; Jiao, B.; Wang, J.; Sun, L.; Li, S.; Wu, Z.; Gao, J.; Zhou, S. Genome-Wide Identification and Expression Analysis of Lipoxygenase Genes in Rose (Rosa chinensis). Genes 2023, 14, 1957. [Google Scholar] [CrossRef] [PubMed]
15. Chen, G.; Hackett, R.; Walker, D.; Taylor, A.; Grierson, D. Identification of a Specific Isoform of Tomato Lipoxygenase (TomloxC) Involved in the Generation of Fatty Acid-Derived Flavor Compounds. Plant Physiol. 2004, 136, 2641–2651. [Google Scholar] [CrossRef] [PubMed]
16. Wang, X.; Zhang, C.; Miao, Y.; Deng, L.; Zhang, B.; Meng, J.; Wang, Y.; Pan, L.; Niu, L.; Liu, H.; et al. Interaction between PpERF5 and PpERF7 enhances peach fruit aroma by upregulating PpLOX4 expression. Plant Physiol. Biochem. 2022, 185, 378–389. [Google Scholar] [CrossRef]
17. Wang, R.; Shen, W.; Liu, L.; Jiang, L.; Liu, Y.; Su, N.; Wan, J. A novel lipoxygenase gene from developing rice seeds confers dual position specificity and responds to wounding and insect attack. Plant Mol. Biol. 2008, 66, 401–414. [Google Scholar] [CrossRef]
18. Lim, C.W.; Han, S.-W.; Hwang, I.S.; Kim, D.S.; Hwang, B.K.; Lee, S.C. The pepper lipoxygenase CaLOX1 plays a role in osmotic, drought, and high salinity. Plant Cell Physiol. 2015, 56, 930–942. [Google Scholar] [CrossRef] [PubMed]
19. Xing, Q.; Liao, J.; Cao, S.; Li, M.; Lv, T.; Qi, H. CmLOX10 positively regulates drought tolerance through jasmonic acid -mediated stomatal closure in oriental melon (Cucumis melo var. makuwa Makino). Sci. Rep. 2020, 10, 17452. [Google Scholar] [CrossRef] [PubMed]
20. Christensen, S.A.; Nemchenko, A.; Borrego, E.; Murray, I.; Sobhy, I.S.; Bosak, L.; DeBlasio, S.; Erb, M.; Robert, C.A.; Vaughn, K.A. The maize lipoxygenase, ZmLOX10, mediates green leaf volatile, jasmonate and herbivore-induced plant volatile production for defense against insect attack. Plant J. 2013, 74, 59–73. [Google Scholar] [CrossRef]
21. Hou, Y.; Ban, Q.; Meng, K.; He, Y.; Han, S.; Jin, M.; Rao, J. Overexpression of persimmon 9-lipoxygenase DkLOX3 confers resistance to Pseudomonas syringae pv. tomato DC3000 and Botrytis cinerea in Arabidopsis. Plant Growth Regul. 2018, 84, 179–189. [Google Scholar] [CrossRef]
22. Zhu, J.; Wang, X.; Guo, L.; Xu, Q.; Zhao, S.; Li, F.; Yan, X.; Liu, S.; Wei, C. Characterization and Alternative Splicing Profiles of the Lipoxygenase Gene Family in Tea Plant (Camellia sinensis). Plant Cell Physiol. 2018, 59, 1765–1781. [Google Scholar] [CrossRef] [PubMed]
23. Rai, A.N.; Mandliya, T.; Kulkarni, P.; Rao, M.; Suprasanna, P. Evolution and Transcriptional Modulation of Lipoxygenase Genes Under Heat, Drought, and Combined Stress in Brassica rapa. Plant Mol. Biol. Rep. 2021, 39, 60–71. [Google Scholar] [CrossRef]
24. Hu, T.; Zeng, H.; Hu, Z.; Qv, X.; Chen, G. Overexpression of the Tomato 13-Lipoxygenase Gene TomloxD Increases Generation of Endogenous Jasmonic Acid and Resistance to Cladosporium fulvum and High Temperature. Plant Mol. Biol. Rep. 2013, 31, 1141–1149. [Google Scholar] [CrossRef]
25. Bannenberg, G.; Martínez, M.; Hamberg, M.; Castresana, C. Diversity of the Enzymatic Activity in the Lipoxygenase Gene Family of Arabidopsis thaliana. Lipids 2009, 44, 85–95. [Google Scholar] [CrossRef]
26. Li, M.; Li, L.; Dunwell, J.M.; Qiao, X.; Liu, X.; Zhang, S. Characterization of the lipoxygenase (LOX) gene family in the Chinese white pear (Pyrusbretschneideri) and comparison with other members of the Rosaceae. BMC Genom. 2014, 15, 444. [Google Scholar] [CrossRef] [PubMed]
27. Zhang, C.; Jin, Y.; Liu, J.; Tang, Y.; Cao, S.; Qi, H. The phylogeny and expression profiles of the lipoxygenase (LOX) family genes in the melon (Cucumis melo L.) genome. Sci. Hortic. 2014, 170, 94–102. [Google Scholar] [CrossRef]
28. Zhang, Q. Genome-Wide Identification and Characterization of Caffeic Acid O-Methyltransferase Gene Family in Soybean. Plants 2021, 10, 2816. [Google Scholar] [CrossRef]
29. Xu, L.; Zhu, X.; Yi, F.; Liu, Y.; Sod, B.; Li, M.; Chen, L.; Kang, J.; Yang, Q.; Long, R. A genome-wide study of the lipoxygenase gene families in Medicago truncatula and Medicago sativa reveals that MtLOX24 participates in the methyl jasmonate response. BMC Genom. 2024, 25, 195. [Google Scholar] [CrossRef] [PubMed]
30. Malviya, R.; Dey, S.; Pandey, A.; Gayen, D. Genome-wide identification and expression pattern analysis of lipoxygenase genes of chickpea (Cicer arietinum L.) in response to accelerated aging. Gene 2023, 874, 147482. [Google Scholar] [CrossRef]
31. Podolyan, A.; White, J.; Jordan, B.; Winefield, C. Identification of the lipoxygenase gene family from Vitis vinifera and biochemical characterisation of two 13-lipoxygenases expressed in grape berries of Sauvignon Blanc. Funct. Plant Biol. 2010, 37, 767–784. [Google Scholar] [CrossRef]
32. Mou, Y.; Sun, Q.; Yuan, C.; Zhao, X.; Wang, J.; Yan, C.; Li, C.; Shan, S. Identification of the LOX gene family in peanut and functional characterization of AhLOX29 in drought tolerance. Front. Plant Sci. 2022, 13, 832785. [Google Scholar] [CrossRef]
33. Li, Z.; Xie, Q.; Yan, J.; Chen, J.; Chen, Q. Genome-Wide Identification and Characterization of the Abiotic-Stress-Responsive GRF Gene Family in Diploid Woodland Strawberry (Fragaria vesca). Plants 2021, 10, 1916. [Google Scholar] [CrossRef] [PubMed]
34. Liu, S.Q.; Liu, X.H.; Jiang, L.W. Genome-wide identification, phylogeny and expression analysis of the lipoxygenase gene family in cucumber. Genet. Mol. Res. 2011, 10, 2613–2636. [Google Scholar] [CrossRef]
35. Guan, J.; Miao, H.; Zhang, Z.; Dong, S.; Zhou, Q.; Liu, X.; Beckles, D.M.; Gu, X.; Huang, S.; Zhang, S. A near-complete cucumber reference genome assembly and Cucumber-DB, a multi-omics database. Mol. Plant 2024, 17, 1178–1182. [Google Scholar] [CrossRef]
36. Li, H.; Wang, S.; Chai, S.; Yang, Z.; Zhang, Q.; Xin, H.; Xu, Y.; Lin, S.; Chen, X.; Yao, Z.; et al. Graph-based pan-genome reveals structural and sequence variations related to agronomic traits and domestication in cucumber. Nat. Commun. 2022, 13, 682. [Google Scholar] [CrossRef]
37. Schreiber, M.; Jayakodi, M.; Stein, N.; Mascher, M. Plant pangenomes for crop improvement; biodiversity and evolution. Nat. Rev. Genet. 2024, 10, 1038. [Google Scholar] [CrossRef] [PubMed]
38. Chen, C.; Chen, X.; Han, J.; Lu, W.; Ren, Z. Genome-wide analysis of the WRKY gene family in the cucumber genome and transcriptome-wide identification of WRKY transcription factors that respond to biotic and abiotic stresses. BMC Plant Biol. 2020, 20, 443. [Google Scholar] [CrossRef] [PubMed]
39. Wang, C.; Han, J.; Wang, T.; Chen, C.; Liu, J.; Xu, Z.; Zhang, Q.; Wang, L.; Ren, Z. Pan-Genome-Wide Identification and Transcriptome-Wide Analysis of DREB Genes That Respond to Biotic and Abiotic Stresses in Cucumber. Agriculture 2022, 12, 1879. [Google Scholar] [CrossRef]
40. Liu, K.; Xu, H.; Gao, X.; Lu, Y.; Wang, L.; Ren, Z.; Chen, C. Pan-Genome Analysis of TIFY Gene Family and Functional Analysis of CsTIFY Genes in Cucumber. Int. J. Mol. Sci. 2024, 25, 185. [Google Scholar] [CrossRef]
41. Qi, C.; Dong, D.; Li, Y.; Wang, X.; Guo, L.; Liu, L.; Dong, X.; Li, X.; Yuan, X.; Ren, S.; et al. Heat shock-induced cold acclimation in cucumber through CsHSFA1d-activated JA biosynthesis and signaling. Plant J. 2022, 111, 85–102. [Google Scholar] [CrossRef] [PubMed]
42. Chen, C.; Chen, H.; Zhang, Y.; Thomas, H.R.; Frank, M.H.; He, Y.; Xia, R. TBtools: An Integrative Toolkit Developed for Interactive Analyses of Big Biological Data. Mol. Plant 2020, 13, 1194–1202. [Google Scholar] [CrossRef] [PubMed]
43. Wang, Y.; Tang, H.; Debarry, J.D.; Tan, X.; Li, J.; Wang, X.; Lee, T.H.; Jin, H.; Marler, B.; Guo, H.; et al. MCScanX: A toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Res. 2012, 40, e49. [Google Scholar] [CrossRef] [PubMed]
44. Wei, G.; Tian, P.; Zhang, F.; Qin, H.; Miao, H.; Chen, Q.; Hu, Z.; Cao, L.; Wang, M.; Gu, X.; et al. Integrative Analyses of Nontargeted Volatile Profiling and Transcriptome Data Provide Molecular Insight into VOC Diversity in Cucumber Plants (Cucumis sativus). Plant Physiol. 2016, 172, 603–618. [Google Scholar] [CrossRef]
45. Adhikari, B.N.; Savory, E.A.; Vaillancourt, B.; Childs, K.L.; Hamilton, J.P.; Day, B.; Buell, C.R. Expression Profiling of Cucumis sativus in Response to Infection by Pseudoperonospora cubensis. PLoS ONE 2012, 7, e34954. [Google Scholar] [CrossRef]
46. Livak, K.J.; Schmittgen, T.D. Analysis of Relative Gene Expression Data Using Real-Time Quantitative PCR and the 2-ΔΔCT Method. Methods 2001, 25, 402–408. [Google Scholar] [CrossRef]
47. Biłas, R.; Szafran, K.; Hnatuszko-Konka, K.; Kononowicz, A.K. Cis-regulatory elements used to control gene expression in plants. Plant Cell Tissue Organ. Cult. 2016, 127, 269–287. [Google Scholar] [CrossRef]
48. Luan, Q.; Chen, C.; Liu, M.; Li, Q.; Wang, L.; Ren, Z. CsWRKY50 mediates defense responses to Pseudoperonospora cubensis infection in Cucumis sativus. Plant Sci. 2019, 279, 59–69. [Google Scholar] [CrossRef]
49. Gao, X.B.; Guo, P.; Wang, Z.Y.; Chen, C.H.; Ren, Z.H. Transcriptome profiling reveals response genes for downy mildew resistance in cucumber. Planta 2021, 253, 112. [Google Scholar] [CrossRef]
50. Zhao, Q.; Feng, Q.; Lu, H.; Li, Y.; Wang, A.; Tian, Q.; Zhan, Q.; Lu, Y.; Zhang, L.; Huang, T.; et al. Pan-genome analysis highlights the extent of genomic variation in cultivated and wild rice. Nat. Genet. 2018, 50, 278–284. [Google Scholar] [CrossRef] [PubMed]
51. Liu, Y.; Du, H.; Li, P.; Shen, Y.; Peng, H.; Liu, S.; Zhou, G.; Zhang, H.; Liu, Z.; Shi, M.; et al. Pan-Genome of Wild and Cultivated Soybeans. Cell 2020, 182, 162–176. [Google Scholar] [CrossRef] [PubMed]
52. Jiang, Y.; Ye, J.; Rasulov, B.; Niinemets, Ü. Role of Stomatal Conductance in Modifying the Dose Response of Stress-Volatile Emissions in Methyl Jasmonate Treated Leaves of Cucumber (Cucumis sativa). Int. J. Mol. Sci. 2020, 21, 1018. [Google Scholar] [CrossRef]
53. Porta, H.; Rocha-Sosa, M. Plant lipoxygenases. Physiological and molecular features. Plant Physiol. 2002, 130, 15–21. [Google Scholar] [CrossRef] [PubMed]
54. Shaban, M.; Ahmed, M.M.; Sun, H.; Ullah, A.; Zhu, L. Genome-wide identification of lipoxygenase gene family in cotton and functional characterization in response to abiotic stresses. BMC Genom. 2018, 19, 599. [Google Scholar] [CrossRef]
55. Zhang, Y.; Wang, K.; Wang, Z.; Li, X.; Li, M.; Zhu, F.; Majeed, Z.; Lan, X.; Guan, Q. The lipoxygenase gene AfLOX4 of Amorpha fruticosa L. is a potential regulator of drought stress tolerance pathways under saline and alkaline conditions. Acta Physiol. Plant. 2023, 45, 72. [Google Scholar] [CrossRef]
56. Gao, X.Q.; Starr, J.; Göbel, C.; Engelberth, J.; Feussner, I.; Tumlinson, J.; Kolomiets, M. Maize 9-lipoxygenase ZmLOX3 controls development, root-specific expression of defense genes, and resistance to root-knot nematodes. Mol. Plant-Microbe Interact. 2008, 21, 98–109. [Google Scholar] [CrossRef] [PubMed]
57. Ju, L.J.; Zhang, C.; Liao, J.J.; Li, Y.P.; Qi, H.Y. An oriental melon 9-lipoxygenase gene CmLOX09 response to stresses, hormones, and signal substances. J. Zhejiang Univ. Sci. B (Biomed. Biotechnol.) 2018, 19, 596–609. [Google Scholar] [CrossRef]
58. Baron, K.N.; Schroeder, D.F.; Stasolla, C. Transcriptional response of abscisic acid (ABA) metabolism and transport to cold and heat stress applied at the reproductive stage of development in Arabidopsis thaliana. Plant Sci. 2012, 188–189, 48–59. [Google Scholar] [CrossRef]
59. Wang, X.; Niu, Y.; Zheng, Y. Multiple Functions of MYB Transcription Factors in Abiotic Stress Responses. Int. J. Mol. Sci. 2021, 22, 6125. [Google Scholar] [CrossRef]
60. Yao, M.; Zhou, X.; Ji, Y.; Luo, M.; Sun, Y.; Zhou, Q.; Ji, S. Potential of Ethylene in Alleviating Cold-Induced Volatile Esters Loss of ‘Nanguo’ Pears by Regulating the Lipoxygenase Pathway. Environ. Exp. Bot. 2022, 194, 104723. [Google Scholar] [CrossRef]
61. Wan, S.; Xin, X.F. Regulation and Integration of Plant Jasmonate Signaling: A Comparative View of Monocot and Dicot. J. Genet. Genom. 2022, 49, 704–714. [Google Scholar] [CrossRef] [PubMed]
Xu H, Liu K, Zhao L, Chen C, Wang L, Ren Z. Pan-Genome Identification and Expression Analysis of Lipoxygenase Genes in Cucumber. Agriculture. 2025; 15(3):245. https://doi.org/10.3390/agriculture15030245
Перевод статьи «Pan-Genome Identification and Expression Analysis of Lipoxygenase Genes in Cucumber» авторов Xu H, Liu K, Zhao L, Chen C, Wang L, Ren Z., оригинал доступен по ссылке. Лицензия: CC BY. Изменения: переведено на русский язык
Фото: freepik





























































































Комментарии (0)